用popart构建常染色体单倍型网络(Autosomal haplotypes network construction with popart)
1)将vcf转化为plink格式,假定输入的vcf文件名为:17893893-17898893.vcf,也可以参考链接:将vcf文件转化为plink格式并且保持phasing状态
/vcftools --vcf 17893893-17898893.vcf --plink-tped --out 17893893-17898893
/plink --tfile 17893893-17898893 --recode --out 17893893-17898893
2) 用PLINK确定要研究的位点是否处于连锁的状态;生成blocks和blocks.det两种后缀格式文件;
/plink --file 17893893-17898893 --blocks no-pheno-req --out 17893893-17898893

以上结果说明rs62033246|rs7189274|rs7187782|rs62033247|rs7194607|rs7200159|rs199711091|rs2361776|rs8054992|rs1845376|rs35902958|rs9928657|rs9928743|rs56078865|rs62033249|rs62033250|rs9936044|rs7184460|rs4781927|rs7202082|rs4780669|rs7202439|rs7195939|rs57418698|rs34615631|rs533867711|rs555603620|rs567435894|rs7499772|rs62033251|rs56248612|rs7202488|rs61671028|rs62033253|rs9928974这35个位点是连锁的
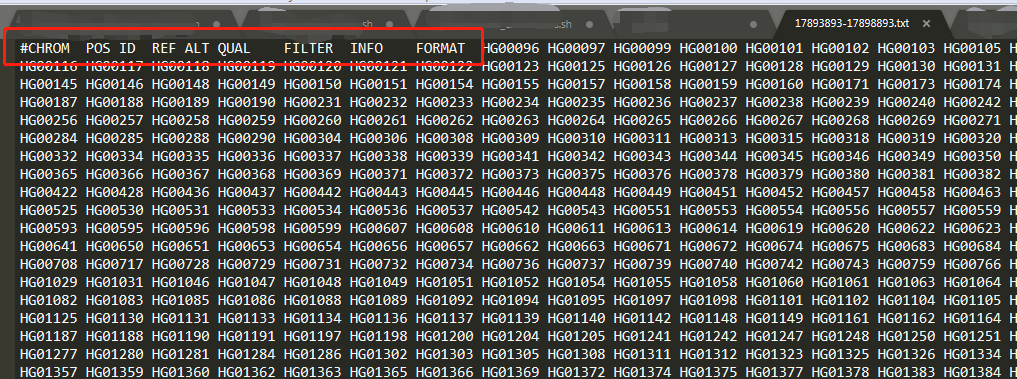
3) 提取这35个位点作为接下来的单倍型网络构建;去掉vcf的头文件,保存为txt格式,见如下图,17893893-17898893.txt:
4)准备17893893-17898893_singstring.txt文件,该文件其实就是去掉 0|0,0|1,1|0,1|1的“|”。

5)接下来,生成两条单链,用R输入如下命令:
install.packages("xlsx")
library(xlsx)
kg_ame<-read.table("E:/17893893-17898893.txt")
kg_ame<-data.frame(kg_ame);
library("stringr");
newdata=c()
for (i in 0:34){
i=i+1
kk=kg_ame[i,6:2509];
kk[which(kk=="0|0")]=paste(kg_ame[i,4],kg_ame[i,4]);
kk[which(kk=="0|1")]=paste(kg_ame[i,4],kg_ame[i,5]);
kk[which(kk=="1|0")]=paste(kg_ame[i,5],kg_ame[i,4]);
kk[which(kk=="1|1")]=paste(kg_ame[i,5],kg_ame[i,5]);
newdata=rbind(newdata,kk)
}
##6:2509指的是2000多个样本对应的碱基;
##0:34指的是35个碱基;这一步是将0|0,0|1,1|0,1|1转化为碱基形式,类似vcf转化为Ped的步骤;
openxlsx::write.xlsx(newdata,file ="E:/17893893-17898893_change.xlsx")
kk=kg_ame[1,7:15];kk
kk[which(kk=="0|0")]=paste(kg_ame[1,4],kg_ame[1,4]);kk
#####分为两条链,转换为ped格式;
library(xlsx)
kg_ame<-read.table("E:/17893893-17898893_singstring.txt")
kg_ame<-data.frame(kg_ame);
library("stringr");
newdata=c()
for (i in 0:34){
i=i+1
kk=kg_ame[i,6:5013];
kk[which(kk=="0")]=kg_ame[i,4];
kk[which(kk=="1")]=kg_ame[i,5];
newdata=rbind(newdata,kk)
}
openxlsx::write.xlsx(newdata,file ="E:/17893893-17898893_changesingstring.xlsx")
se1<-seq(from=1,to=5008,by=2);
se2<-seq(from=2,to=5008,by=2);
stringone<-newdata[,se1];
stringtwo<-newdata[,se2];
openxlsx::write.xlsx(stringone,file ="E:/17893893-17898893_changesingstringone.xlsx")
openxlsx::write.xlsx(stringtwo,file ="E:/17893893-17898893_changesingstringtwo.xlsx")
####以上步骤为转为两条单链
6) 将上述的两条单链生成fas格式;
paste -d "\n" 17893893-17898893_changesingstringone_firstdot.txt 17893893-17898893_changesingstringone.txt > 17893893-17898893_changesingstringone_dot.fas
paste -d "\n" 17893893-17898893_changesingstringtwo_firstdot.txt 17893893-17898893_changesingstringtwo.txt > 17893893-17898893_changesingstringtwo_dot.fas
cat 17893893-17898893_changesingstringone_dot.fas 17893893-17898893_changesingstringtwo_dot.fas >> 17893893-17898893_changesingstring_onetwo_dot.fas #这两条链合并在一起
17893893-17898893_changesingstringone_firstdot.txt 文件格式如下:
17893893-17898893_changesingstringone.txt的文件格式如下:这个文件就是5)生成出来的其中一条单链。
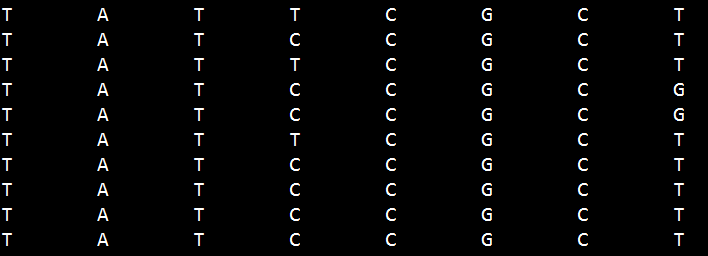
生成的fas文件如下:
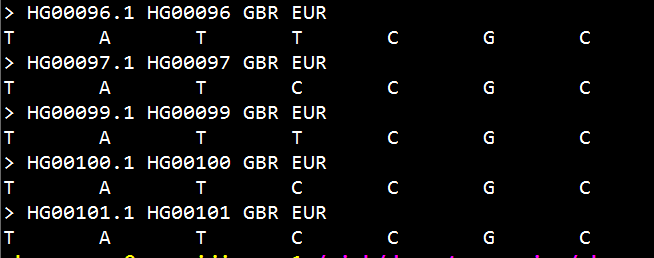
第二条链的处理同上。最后一步,就是把这两条链合并在一起
7)用DnaSP6将fas文件生成nex文件

8)统计nex文件中不同群体的haplotype的数量
准备17893893-17898893_hap.xlsx文件

准备allsample.txt文件
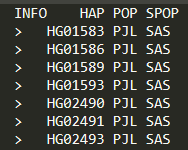
输入如下命令:
#####统计nex生成的所有haplotype所在的样本和群体,这里假定生成了100个haplotype
library(dplyr)
library(xlsx)
model_57hanchip<-read.xlsx("E:/17893893-17898893_hap.xlsx",2)
model_57hanchip<-data.frame(model_57hanchip)
kg_ame<-read.table("E:/allsample.txt")
kg_ame<-data.frame(kg_ame);
newdata=c()
for (i in 0:99){
i=i+1
HAP1<-kg_ame[,2];
df1 <- data.frame(id = HAP1, kg_ame[,3],kg_ame[,4])
HAP2<-model_57hanchip[,i]
df2 <- data.frame(id = HAP2)
k=merge(df1, df2, by = "id",all = FALSE)
j=table(k[,3])
newdata=rbind(newdata,j)
print(i)
}
openxlsx::write.xlsx(newdata,file ="E:/17893893-17898893_hapcount.xlsx")
9)修改生成的nex文件
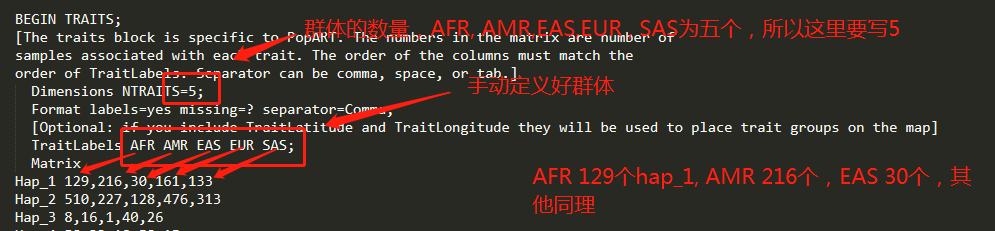
DnaSP6生成的nex文件并没有BEGIN TRAITS的头文件,因此这一步是需要手动修改的。
这一步,除了圈出来的数字和AFR AMR EAS EUR SAS是需要修改,其他照抄。
10)用popart构建单体型网络
导入文件后,选择适合的算法,即可生成。
用popart构建常染色体单倍型网络(Autosomal haplotypes network construction with popart)的更多相关文章
- 构建 CDN 分发网络架构简析
构建 CDN 分发网络架构 CDN的基本目的:1.通过本地缓存实现网站的访问速度的提升 CDN的关键点:CNAME在域名解析:split智能分发,引流到最近缓存节点
- 基因调控网络 (Gene Regulatory Network) 01
本文为入门级的基因调控网络文章,主要介绍一些基本概念及常见的GRN模型. 概念:基因调控网络 (Gene Regulatory Network, GRN),简称调控网络,指细胞内或一个基因组内基因和基 ...
- VMware虚拟机上网络连接(network type)的三种模式--bridged、host-only、NAT
VMware虚拟机上网络连接(network type)的三种模式--bridged.host-only.NAT VMWare提供了三种工作模式,它们是bridged(桥接模式).NAT(网络地址转换 ...
- [USACO08JAN]手机网络Cell Phone Network
[USACO08JAN]手机网络Cell Phone Network 题目描述 Farmer John has decided to give each of his cows a cell phon ...
- linux 网络虚拟化: network namespace 简介
linux 网络虚拟化: network namespace 简介 network namespace 是实现网络虚拟化的重要功能,它能创建多个隔离的网络空间,它们有独自的网络栈信息.不管是虚拟机还是 ...
- 洛谷 P2812 校园网络【[USACO]Network of Schools加强版】 解题报告
P2812 校园网络[[USACO]Network of Schools加强版] 题目背景 浙江省的几所OI强校的神犇发明了一种人工智能,可以AC任何题目,所以他们决定建立一个网络来共享这个软件.但是 ...
- Solaris11.1网络配置(Fixed Network)
Solaris11的网络配置与Solaris10有很大不同,Solaris11通过network configuration profiles(NCP)来管理网络配置. Solaris11网络配置分为 ...
- 洛谷P2899 [USACO08JAN]手机网络Cell Phone Network
P2899 [USACO08JAN]手机网络Cell Phone Network 题目描述 Farmer John has decided to give each of his cows a cel ...
- (58)zabbix网络拓扑图配置network map
zabbix网络地图介绍 “zabbix network map”可以简单的理解为动态网络拓扑图,可以针对业务来配置zabbix map, 通过map可以了解应用的整体状况:服务器是否异常.网络是否有 ...
随机推荐
- linux 挂载windows下目录,其它linux机器nfs的目录,自己dd的文件
如有转载,不胜荣幸.http://www.cnblogs.com/aaron-agu/ 挂载window下共享的目录 //192.168.0.11/share /mnt 挂载其它linux机器下目录 ...
- Git拉取项目时报错“remote: HTTP Basic: Access denied”解决方法
问题: Git拉取项目时报错“remote: HTTP Basic: Access denied”,此问题多为本地密码与远端密码不符导致. 解决方法: 在下载地址中加上用户名和密码即可,如下: htt ...
- SQL Server 2008 开启远程连接
除了 IP1.IP2 外,也要把 IPALL 的端口也设置为 1433 参考:SQL Server开启1433端口,彻底解决方案
- SharePoint Server 2016 - Configure Office Online Server
Step 1: Create the binding between SharePoint 2016 and Office Web Apps Server To get started, open ...
- python基础数据类型—int、bool、字符串的常用方法
1.int int为整型数据,主要用于计算和类型转化(将字符串转为数字) 常用方法 #bit_length()当用二进制表示数字时所用最少位数,如下十进制数12用二进制表示是1100(bin),所以# ...
- FFmpeg 将YUV数据转RGB
只要开始初始化一次,结束后释放就好,中间可以循环转码 AVFrame *m_pFrameRGB,*m_pFrameYUV; uint8_t *m_rgbBuffer,*m_yuvBuffer; str ...
- IntelliJ IDEA 取消【import .;】星号导包
Setting -- Editor -- Code Style -- Java -- Imports 在 在 具体数值自行填写,够大即可,截图如下:
- 第四十一天 socker server和 event
今日内容 1.基于TCP的socketserver 2.基于UDP的socketserver 3.event 一.TCP的socketserver #服务器 import socketserver f ...
- 【XSY2787】Mythological VII 贪心
题目描述 有两个指针\(l,r\),初始时\(l=r=k\) 给你\(a_1,\ldots,a_n\),你要移动\(l,r\),\(l\)只能每次向左移一个数,\(r\)只能向右移一个数,要求任意时刻 ...
- python构建bp神经网络_鸢尾花分类(一个隐藏层)__2.代码实现
IDE:jupyter 数据集请查看:鸢尾花数据集 测试效果预览 成功率96.7% 代码已上传到码云
