python版的MCScan绘图
最近发现了python版的MCScan,是个大宝藏。由于走了不少弯路,终于画出美图,赶紧记录下来。
github地址 https://github.com/tanghaibao/jcvi/wiki/MCscan-(Python-version)
软件安装
1 ## 安装lastal
2 网址:http://last.cbrc.jp
3 unzip last-1060.zip
4 cd last-1060
5 make
6
7 # 把scripts, src 添加到环境变量
8
9 ## jcvi
10 pip install jcvi
11
12 ## 若出现 from rillib.parse import urlparse 缺少parse模块,则装parse模块,然后将urllib.parse 改为urlparse; 因为urlparse模块在Python 3中重命名为urllib.parse,所以模块在Python 2.7下应该使用urlparse。输入数据
gff文件转bed格式
1 ## 以spinach,和sugar为例子
2 python -m jcvi.formats.gff bed --type=mRNA --key=ID spinach_gene_v1.gff3 -o spinach.bed
3 python -m jcvi.formats.gff bed --type=mRNA --key=ID BeetSet-2.unfiltered_genes.1408.gff3.txt -o sugar.bed
4
5 ##参数
6 --type:gff文件中第三列
7 --key:type对应的第九列信息前缀
8
9 我们分析只需要用到每个基因最长的转录本就行,在sugar中是以多个转录本进行存储,因为需要获取最长转录本
10
11 ## 将sugar中bed进行去重复
12 python -m jcvi.formats.bed uniq sugar.bed获取cds/pep序列
1 ## cds和pep序列均可以进行共线性分析
2 ## 根据上述得到的.bed文件调取对应cds和蛋白序列
3 # spinach
4 seqkit grep -f <(cut -f4 spianch.bed) spinach.cds.fa | seqkit seq -I >spinach.cds
5 seqkit grep -f <(cut -f4 spianch.bed) spinach.pep.fa | seqkit seq -I >spinach.pep
6
7 # sugar
8 seqkit grep -f <(cut -f4 sugar.uniq.bed) BeetSet-2.genes.1408.cds.fa | seqkit seq -i >sugar.cds
9 seqkit grep -f <(cut -f4 sugar.uniq.bed) BeetSet-2.genes.1408.pep.fa | seqkit seq -i >sugar.pep小知识:也可以根据gff文件,基因组ref.fa文件中直接调取cds,和pep序列
1 ## 需要安装cufflinks
2
3 ## 提取cds
4 gffread in.gff3 -g ref.fa -x cds.fa
5
6 ## 提取pep
7 gffread in.gff3 -g ref.fa -y pep.fa
共线性分析
1 ## 新建一个文件夹,方便在报错的时候,把全部都给删了
2 mkdir cds && cd cds
3 ln -s ../sugar.cd ./
4 ln -s ../sugar.uniq.bed ./sugar.bed
5 ln -s ../spinach.cds ./
6 ln -s ../spinch.bed ./
7
8 ## 运行代码
9 python -m jcvi.compara.catalog ortholog (--dbtype prot[蛋白分析]) --no_strip_names spinach sugar
10
11 结果:
12 spinach.sugar.anchors:共线性block区块(高质量)
13 spinach.sugar.last:原始的last输出
14 spinach.sugar.last.filtered:过滤后的last输出
15 spinach.sugar.pdf:点阵图
16
17 ## 如果遇到报错,多半是要安装python包,更新Latex可视化
共线性图
1 ## 首先生成.sinple文件
2 python -m jcvi.compara.synteny screen --minspan=30 --simple spinach.sugar.anchors spinach.sugar.anchors.new
3
4 ## 编辑配置文件seqids 和layout
5
6 #设置需要展示染色体号
7 vi seqids
8 chr1,chr2,chr3,chr4,chr5,chr6 #spinach
9 Bvchr1.sca001,Bvchr2.sca001,Bvchr3.sca001 #sugar
10
11 # 设置颜色,长宽等
12 vi layout
13 # y, xstart, xend, rotation, color, label, va, bed
14 .6, .1, .8, 0, red, spinach, top, spinach.bed
15 .4, .1, ,8, 0, blue, sugar, top, sugar.bed
16 # edges
17 e, 0, 1, spinach.sugar.anchors.simple
18
19 注意, #edges下的每一行开头都不能有空格
20
21 ## 运行代码
22 python -m jcvi.graphics.karyotype seqids layout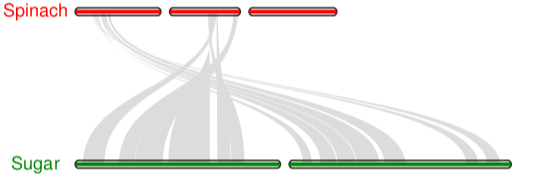
若要突出显示某一共线性则可以在对应的位置添加g*
1 vi spinach.sugar.anchors.simple
2 g*Spo03717 Spo03716 Bv3_048620_odzi.t1 Bv3_049090_cxmm.t1 46 +
3 Spo17356 Spo17350 Bv1_001140_tedw.t1 Bv1_001540_xzdn.t1 41 -
4 Spo13685 Spo13730 Bv5_123480_yfcy.t1 Bv5_123900_rucq.t1 46 -
5 Spo26250 Spo26280 Bv5_117270_qhwj.t1 Bv5_117680_iykf.t1 36 +
6 Spo19005 Spo06982 Bv7_173830_frmo.t1 Bv7_174150_pckw.t1 37 +
7 Spo19374 Spo20559 Bv4_081440_riqq.t1 Bv4_081750_yeuy.t1 32 -
8
9 #运行
10 python -m jcvi.graphics.karyotype seqids layout
若要显示3个物种的共线性,则应两两比对,得到两个*.simple文件,并对其进行配置(来自https://www.jianshu.com/p/39448b970287)
1 $ vi layout
2 # y, xstart, xend, rotation, color, label, va, bed
3 .7, .1, .8, 15, , Grape, top, grape.bed
4 .5, .1, .8, 0, , Peach, top, peach.bed
5 .3, .1, .8, -15, , Cacao, bottom, cacao.bed
6 # edges
7 e, 0, 1, grape.peach.anchors.simple
8 e, 1, 2, peach.cacao.anchors.simple
9
10 $ vi seqids
11 chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19
12 scaffold_1,scaffold_2,scaffold_3,scaffold_4,scaffold_5,scaffold_6,scaffold_7,scaffold_8
13 scaffold_1,scaffold_2,scaffold_3,scaffold_4,scaffold_5,scaffold_6,scaffold_7,scaffold_8,scaffold_9,scaffold_10r
14
15 $ python -m jcvi.graphics.karyotype seqids layout

3个物种三角形排序配置文件(来自https://www.jianshu.com/p/f7971dbf5f85)
1 # y, xstart, xend, rotation, color, label, va, bed
2 .5, 0.025, 0.625, 60, , Grape, top, grape.bed
3 .2, 0.2, .8, 0, , Peach, top, peach.bed
4 .5, 0.375, 0.975, -60, , Cacao, top, cacao.bed
5 # edges
6 e, 0, 1, grape.peach.anchors.simple
7 e, 1, 2, peach.cacao.anchors.simple
8
9 #运行
10 python -m jcvi.graphics.karyotype seqids layout

除此之外,Tbtools也可以完成类似图片,对于新手来说更加容易上手,具体可查看以下内容
参考
1、其实MCScan画图也可以很好看
2、「JCVI教程」如何绘制CNS级别的共线性图(上)
3、「JCVI教程」如何绘制CNS级别的共线性图(中)
关注下方公众号可获得更多精彩

python版的MCScan绘图的更多相关文章
- 【原】Learning Spark (Python版) 学习笔记(三)----工作原理、调优与Spark SQL
周末的任务是更新Learning Spark系列第三篇,以为自己写不完了,但为了改正拖延症,还是得完成给自己定的任务啊 = =.这三章主要讲Spark的运行过程(本地+集群),性能调优以及Spark ...
- 数据结构:顺序表(python版)
顺序表python版的实现(部分功能未实现) #!/usr/bin/env python # -*- coding:utf-8 -*- class SeqList(object): def __ini ...
- python版恶俗古风自动生成器.py
python版恶俗古风自动生成器.py """ python版恶俗古风自动生成器.py 模仿自: http://www.jianshu.com/p/f893291674c ...
- LAMP一键安装包(Python版)
去年有出一个python整的LAMP自动安装,不过比较傻,直接调用的yum 去安装了XXX...不过这次一样有用shell..我也想如何不调用shell 来弄一个LAMP自动安装部署啥啥的..不过尼玛 ...
- 编码的秘密(python版)
编码(python版) 最近在学习python的过程中,被不同的编码搞得有点晕,于是看了前人的留下的文档,加上自己的理解,准备写下来,分享给正在为编码苦苦了挣扎的你. 编码的概念 编码就是将信息从一种 ...
- Zabbix 微信报警Python版(带监控项波动图片)
#!/usr/bin/python # -*- coding: UTF- -*- #Function: 微信报警python版(带波动图) #Environment: python import ur ...
- 豆瓣top250(go版以及python版)
最近学习go,就找了一个例子练习[go语言爬虫]go语言爬取豆瓣电影top250,思路大概就是获取网页,然后根据页面元素,用正则表达式匹配电影名称.评分.评论人数.原文有个地方需要修改下patte ...
- python版接口自动化测试框架源码完整版(requests + unittest)
python版接口自动化测试框架:https://gitee.com/UncleYong/my_rf [框架目录结构介绍] bin: 可执行文件,程序入口 conf: 配置文件 core: 核心文件 ...
- python中利用matplotlib绘图可视化知识归纳
python中利用matplotlib绘图可视化知识归纳: (1)matplotlib图标正常显示中文 import matplotlib.pyplot as plt plt.rcParams['fo ...
随机推荐
- 天脉2(ACoreOS653)操作系统学习02
天脉2(ACoreOS653)操作系统学习02 一.分区内通信方法 分区内通信指同一分区内进程之间的通信.ARINC 653定义的分区内进程通信机制,包括:缓存队列(Buffers-Queue).黑板 ...
- 实现服务器和客户端数据交互,Java Socket有妙招
摘要:在Java SDK中,对于Socket原生提供了支持,它分为ServerSocket和Socket. 本文分享自华为云社区<Java Socket 如何实现服务器和客户端数据交互>, ...
- 【二食堂】Beta - 测试报告
Beta - 测试报告 测试过程中发现的bug Beta阶段的新bug 我们在Beta阶段的开发过程中就进行了测试,发现了许多bug.这其中后端的bug比较多,在这里我列举一些比较重要的功能性bug. ...
- 了解 js 堆内存 、栈内存 。
js中的堆内存与栈内存 在js引擎中对变量的存储主要有两种位置,堆内存和栈内存. 和java中对内存的处理类似,栈内存主要用于存储各种基本类型的变量,包括Boolean.Number.String.U ...
- mongodb的简单查询
此篇文章简单的记录一下mongodb 的简单查询操作. 一.数据准备: db.persons.insertMany([ {'userId':1,name:'张三','age':20,'scores': ...
- DDD领域驱动设计-设计规范-Ⅵ
不以规矩,不能成方圆. -战国·邹·孟轲<孟子·离娄章句上 ...
- Python3 TypeError: initial_value must be str or None, not bytes
response.read() returns an instance of bytes while StringIO is an in-memory stream for text only. Us ...
- poj 3020 Antenna Placement(二分图最大匹配)
题意: N行M列的矩阵,每个格子里不是 * 就是 O . * :是一个利益点. O:是一个空白点. 每次可以用一个圈覆盖相邻的两个*.(左右相邻或上下相邻). 问最少需要多少个圈可以覆盖所有的*. 思 ...
- Spring事务的介绍,以及基于注解@Transactional的声明式事务
前言 事务是一个非常重要的知识点,前面的文章已经有介绍了关于SpringAOP代理的实现过程:事务管理也是AOP的一个重要的功能. 事务的基本介绍 数据库事务特性: 原子性 一致性 隔离性 持久性 事 ...
- 修改记事本PE结构弹计算器Shellcode
目录 修改记事本PE结构弹计算器Shellcode 0x00 前言 0x01 添加新节 修改节数量 节表位置 添加新节表信息 0x02 添加弹计算器Shellcode 修改代码 0x03 修改入口点 ...
