全基因组关联分析(Genome-Wide Association Study,GWAS)流程
全基因组关联分析流程:
一、准备plink文件
1、准备PED文件
PED文件至少有六列,内容如下:
Family ID
Individual ID
Paternal ID
Maternal ID
Sex (1=male; 2=female; other=unknown)
Phenotype(-9 missing 0 missing 1 unaffected 2 affected)
genotype( 1,2,3,4 or A,C,G,T missing 0)
PED文件是空格(空格或制表符)分隔的文件。
PED文件长这个样:

2、准备MAP文件
MAP文件有四列,四列内容如下:
chromosome (1-22, X, Y or 0 if unplaced)
rs# or snp identifier
Genetic distance (morgans)
Base-pair position (bp units)
MAP文件长这个样:
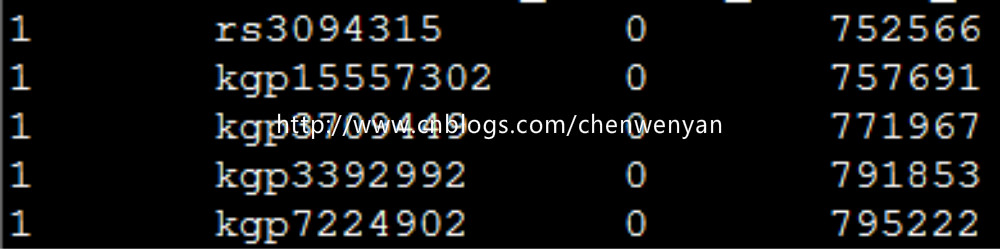
3、生成bed、fam、bim、文件
输入命令
plink --file mydata --out mydata --make-bed
注:plink指的是plink软件,如果软件安装在某个指定的路径的话,前面还要加上路径,比如安装在路径为/your/pathway/的文件夹下,则命令应该为“/your/pathway/plink --file mydata --out mydata --make-bed”
mydata指的是1和2生成的PED和MAP文件名,不需要写.ped和.map后缀
二、准备表型文件(Alternate phenotype files)
一般表型文件为txt格式,表型文件有三列,分别为:
Family ID
Individual ID
Phenotype
假如有多种表型,第一列和第二列还是Family ID、Individual ID,第三列及以后的每列都是表型,例如以下:
Family ID
Individual ID
Phenotype A
Phenotype B
Phenotype C
Phenotype D
Phenotype E
……
表型文件长这样:
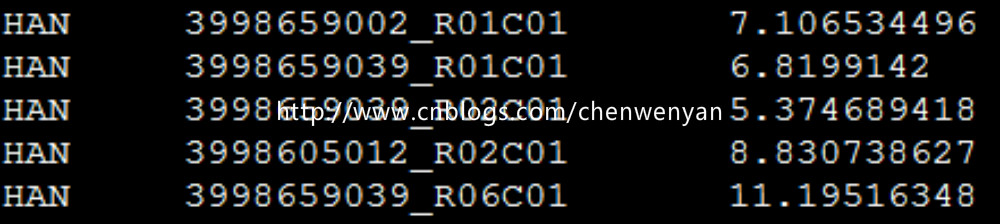
缺失值的处理:缺失值的表型用-9表示;
case和control的处理:通常情况下,1表示control,2表示case,0表示缺失,但如果你加上--1的参数,则0表示control,1表示case。
三、准备协变量文件(Covariate files)
协变量文件同表型文件类似,第一列和第二列是Family ID、Individual ID,第三列及以后的每列都是协变量
Family ID
Individual ID
Covariate A
Covariate B
Covariate C
Covariate D
Covariate E
……
协变量文件长这个样(这里有三个协变量,分别为Sex,Age,temperature):
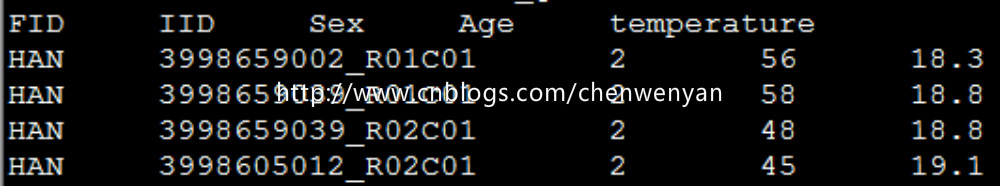
四、plink进行表型和基因型以及协变量的关联分析
命令如下:
plink --bfile mydata --linear --pheno pheno.txt --mpheno 1 --covar covar.txt --covar-number 1,2,3 --out mydata –noweb
生成的文件为mydata.assoc.linear
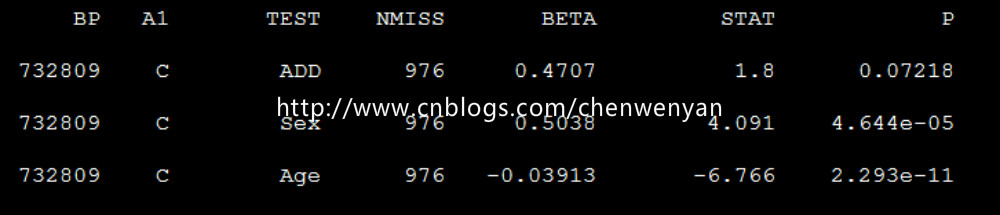
注:“mydata”mydata文件不需要后缀,“--mpheno 1”指的是表型文件的第三列(即第一个表型)
“--covar-number 1,2,3”指的是协变量文件的第三列、第四列、第五列(即第一个、第二个、第三个协变量)
“--linear”指的是用的连续型线性回归,如果表型为二项式(即0、1)类型,则用“--logistic”
五、画曼哈顿图
安装R语言的CpGassoc包,其中的manhattan(),即可画曼哈顿图,或者参照本文R语言画全基因组关联分析中的曼哈顿图(manhattan plot)
全基因组关联分析(Genome-Wide Association Study,GWAS)流程的更多相关文章
- GWAS 全基因组关联分析 | summary statistic 概括统计 | meta-analysis 综合分析
有很多概念需要明确区分: 人有23对染色体,其中22对常染色体autosome,另外一对为性染色体sex chromosome,XX为女,XY为男. 染色体区带命名:在标示一特定的带时需要包括4项:① ...
- 【GWAS文献解读】疟原虫青蒿素抗药性的全基因组关联分析
英文名:Genetic architecture of artemisinin-resistant Plasmodium falciparum 中文名:疟原虫青蒿素抗药性的全基因组关联分析 期刊:Na ...
- 全基因组关联分析(GWAS)的计算原理
前言 关于全基因组关联分析(GWAS)原理的资料,网上有很多. 这也是我写了这么多GWAS的软件教程,却从来没有写过GWAS计算原理的原因. 恰巧之前微博上某位小可爱提问能否写一下GWAS的计算原理. ...
- GWAS | 全基因组关联分析 | Linkage disequilibrium (LD)连锁不平衡 | 曼哈顿图 Manhattan_plot | QQ_plot | haplotype phasing
现在GWAS已经属于比较古老的技术了,主要是碰到严重的瓶颈了,单纯的snp与表现的关联已经不够,需要具体的生物学解释,这些snp是如何具体导致疾病的发生的. 而且,大多数病找到的都不是个别显著的snp ...
- 全基因组关联分析(GWAS):为何我的QQ图那么飘
前段时间有位小可爱问我,为什么她的QQ图特别飘,如果你不理解怎样算飘,请看下图: 理想的QQ图应该是这样的: 我当时的第一反应是:1)群体分层造成的:2)表型分布有问题.因此让她检查一下数据的群体分层 ...
- 一行命令学会全基因组关联分析(GWAS)的meta分析
为什么需要做meta分析 群体分层是GWAS研究中一个比较常见的假阳性来源. 也就是说,如果数据存在群体分层,却不加以控制,那么很容易得到一堆假阳性位点. 当群体出现分层时,常规手段就是将分层的群体独 ...
- 全基因组关联分析(GWAS)扫不出信号怎么办(文献解读)
假如你的GWAS结果出现如下图的时候,怎么办呢?GWAS没有如预期般的扫出完美的显著信号,也就没法继续发挥后续研究的套路了. 最近,nature发表了一篇文献“Common genetic varia ...
- R语言画全基因组关联分析中的曼哈顿图(manhattan plot)
1.在linux中安装好R 2.准备好画曼哈顿图的R脚本即manhattan.r,manhattan.r内容如下: #!/usr/bin/Rscript #example : Rscript plot ...
- 全基因组关联分析学习资料(GWAS tutorial)
前言 很多人问我有没有关于全基因组关联分析(GWAS)原理的书籍或者文章推荐. 其实我个人觉得,做这个分析,先从跑流程开始,再去看原理. 为什么这么说呢,因为对于初学者来说,跑流程就像一个大黑洞,学习 ...
随机推荐
- Boot 44b0x by uboot
1. Creat a branch from tag v2013.10-rc4 2. Build it: make B2 Install NFS service for Ubuntu 12.04 1. ...
- sqlce中不支持sp_rename修改表名
The sp_rename procedure is not avialable in SQL CE! In Sql Server 2005 Management Studio you have to ...
- SVN show log failed
Q: SVN 不能显示日志 能正常update, commit,但是show log的时候报错 A:可能原因是服务器权限配置问题 修改配置文件svnserve.conf 和 authz, 修改前请先备 ...
- 7、IMS - DNS & ENUM
1.相关基础SBC:http://blog.sina.com.cn/s/blog_7a6f76080100vp9r.html 2.ENUM/DNS查询过程:http://blog.sina.com.c ...
- Linux学习四:UDP编程(上)
关于UDP和TCP对比优缺,这里就不说了. 使用UDP代码所掉用的函数和用于TCP的函数非常类似,这主要因为套接口库在底层的TCP和UDP的函数上加了一层抽象,通过这层抽象使得编程更容易,但失去了一些 ...
- 关于jq+devexpress基础知识总结(随便的基础)
//获取某行某列的值 onSelectionChanged: function (selectedItems) { ]; if (data != null) postionno = data.POST ...
- MVC之路由规则 (自定义,约束,debug)
自定义路由规则的要求,小范围写在前,大范围写在后.路由规则可以注册多条,路由规则的名称不能重复路由规则有顺序,并且按照顺序进行匹配,建议小范围写在前,大范围写在后.路由规则可以设置约束 即正则表达式路 ...
- Django快速学习搭建blog项目
新手学习Django,本文学习的文档是<Django Web开发指南>.好了我也是新手,没什么好说了,go!- 首先先确定环境,我是在linux(Ubuntu14.04 gnome)下. ...
- jQuery EasyUI CheckBoxTree的级联选中
:子结点选中,父节点随之选中,父节点取消,子节点随之取消 代码: <!DOCTYPE html> <html xmlns="http://www.w3.org/1999/x ...
- bashrc
# ~/.bashrc: executed by bash(1) for non-login shells.# see /usr/share/doc/bash/examples/startup-fil ...
