VCF和GVCF格式说明
注意:本文的内容主要来自于GATK官网的讲解,所以vcf也是GATK产生的,用其他caller,比如varscan2产生的vcf文件的内容注释可能不一致。
参考:https://gatkforums.broadinstitute.org/gatk/discussion/1268/what-is-a-vcf-and-how-should-i-interpret-it
VCF:由HEADER和RECORDS组成。
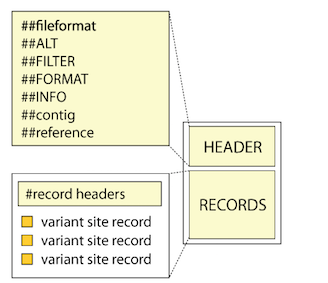
RECORDS的FORMAT内容详解:
QUAL:指的是caller正确的识别该变异位点的可能性,属于phred-scale quality score的一个应用。
GT,GQ,PL三者的关系:
GT是指该位点最有可能的基因型。
GQ是该位点第二有可能的基因型的PL值。
PL是不同基因型对应的标准化的可能性。
对于二倍体生物来说,PL有三个值,分别对应0/0,0/1,1/1。最有可能的基因型的PL值为0,第二小的是第二个可能。GQ反映的是第二个小的基因型的PL值,如果该值超过99,则定位99,因为超过了99,其几乎不能威胁第一个可能的地位。
计算方法:
PL(0/1) = 0 (the normalized value that corresponds to a likelihood of 1.0) as is always the case for the assigned allele,
but the next PL is PL(1/1) = 26 (which corresponds to 10^(-2.6), or 0.0025).
QUAL和GQ的区别:

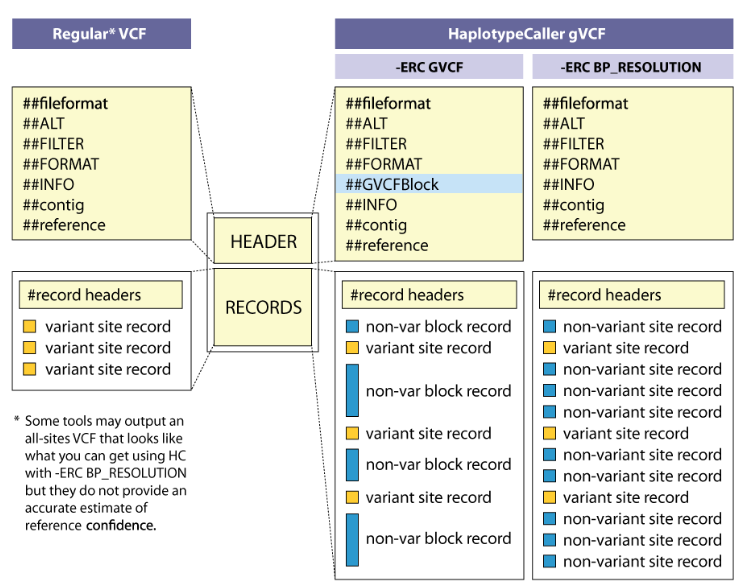
GVCF和VCF的最大区别是在于GVCF文件会记录所有的点,包括哪些没有突变的点。
在GVCF模式下,那些没有变异的点会形成一个未变异块,non-var block record。
GVCF的好处:能更方便把一群样本的GVCF联合起来,以便进行下一步分析,提高分析效率。而且GVCF的records还会提供一个
The records in a gVCF include an accurate estimation of how confident we are in the determination that the sites are homozygous-reference or not. This estimation is generated by the HaplotypeCaller's built-in reference model
VCF和GVCF格式说明的更多相关文章
- bcftools或vcftools提取指定区段的vcf文件(extract specified position )
下载安装bcftools 见如下命令: bcftools filter 1000Genomes.vcf.gz --regions 9:4700000-4800000 > 4700000-4800 ...
- GWAS | 全基因组关联分析 | Linkage disequilibrium (LD)连锁不平衡 | 曼哈顿图 Manhattan_plot | QQ_plot | haplotype phasing
现在GWAS已经属于比较古老的技术了,主要是碰到严重的瓶颈了,单纯的snp与表现的关联已经不够,需要具体的生物学解释,这些snp是如何具体导致疾病的发生的. 而且,大多数病找到的都不是个别显著的snp ...
- 收集vcftools所有用法
VCFtools用来处理VCF文档. 筛选特定突变 比较文件 总结突变 转化文件格式 验证并合并文件 取突变交集和差集 Get basic file statistics input可以为VCF或BC ...
- the pipeline of call SNP
######################################## ############### Mapping ################ ################## ...
- 【转】GATK使用方法详解(包含bwa使用)
一.使用GATK前须知事项: (1)对GATK的测试主要使用的是人类全基因组和外显子组的测序数据,而且全部是基于illumina数据格式,目前还没有提供其他格式文件(如Ion Torrent)或者实验 ...
- GWAS Catalog数据库简介
GWAS Catalog The NHGRI-EBI Catalog of published genome-wide association studies EBI负责维护的一个收集已发表的GWAS ...
- admixture 群体结构分析
tructure是与PCA.进化树相似的方法,就是利用分子标记的基因型信息对一组样本进行分类,分子标记可以是SNP.indel.SSR.相比于PCA,进化树,群体结构分析可明确各个群之间是否存在交流及 ...
- plink 进行PCA分析
当我们进行群体遗传分析时,得到vcf后,可利用plink进行主成分(PCA)分析: 一.软件安装 1 conda install plink 二.使用流程 第一步:将vcf转换为plink格式 1 p ...
- pysam - 多种格式基因组数据(sam/bam/vcf/bcf/cram/…)读写与处理模块(python)
在开发基因组相关流程或工具时,经常需要读取.处理和创建bam.vcf.bcf文件.目前已经有一些主流的处理此类格式文件的工具,如samtools.picard.vcftools.bcftools,但此 ...
随机推荐
- 【BZOJ1441】Min 拓展裴蜀定理
[BZOJ1441]Min Description 给出n个数(A1...An)现求一组整数序列(X1...Xn)使得S=A1*X1+...An*Xn>0,且S的值最小 Input 第一行给出数 ...
- R中基本命名(未完)
ls() #查看 rm(list=ls()) #清除内存变量 library() #载入库包 help() #查看帮助文档 head(iris) #查看数据集 class(iris) #查看数据集的类 ...
- Android中可自由移动悬浮窗口的实现
http://www.xsmile.net/?p=538 调用WindowManager,并设置WindowManager.LayoutParams的相关属性,通过WindowManager的ad ...
- linux 命令行 执行 php
w为监控响应功能做准备. ubuntu@VM-52-248-ubuntu:~$ php -f /var/www/html/wlinux.phpwwubuntu@VM-52-248-ubuntu:~$ ...
- 网络编程 - socket通信/粘包/文件传输/udp - 总结
socket通信 1.简单的套接字通信 import socket phone = socket.socket(socket.AF_INET,socket.SOCK_STREAM) phone.bin ...
- Fibonacci----poj3070(矩阵快速幂, 模板)
题目链接:http://poj.org/problem?id=3070 . 就是斐波那契的另一种表示方法是矩阵的幂: 所以是矩阵快速幂:矩阵快速幂学习 #include <cstdio> ...
- linux下的KSM内存共享机制分析
2017-04-26 KSM是内核中的一种内存共享机制,在2.6.36版本的内核中开始引入,简单来说就是其会 合并某些相同的页面以减少页面冗余.在内核中有一个KSM守护进程 ksmd,它定期扫描用户向 ...
- ssm框架集成Quartz定时器
第一步:添加依赖 <dependency> <groupId>org.quartz-scheduler</groupId> <artifactId>qu ...
- Commons Email使用
Apache Commons Email Apache的一个开源项目,是基于另一个开源项目Java Mail上进行封装的,使用起来更加简单方便: http://commons.apache.org/p ...
- mysql 约束条件 auto_increment 自动增长 创建表时设置自增字段
auto_increment mysql) )auto_increment; Query OK, rows affected (0.01 sec) mysql> show create tabl ...
