Journal of Proteome Research | Utilization of the Proteome Data Deposited in SRMAtlas for Validating the Existence of the Human Missing Proteins in GPM (解读人:梁嘉琪)
文献名:Utilization of the Proteome Data Deposited in SRMAtlas for Validating the Existence of the Human Missing Proteins in GPM(利用SRMAtlas中的已有的蛋白质组数据验证GPM中人类缺失蛋白的存在)
期刊名:journal of proteome
发表时间:2019.10.24
IF:3.780
单位:
1、日本Niigata大学
2、开罗Al-Azhar大学
物种:人类
技术:生物信息学;LC-MS/MS
一、 概述
2012年以来,人类蛋白质组计划(HPP)致力于研究人类所有蛋白质。然而,根据neXtProt(2019−1)的最新发布,大约10%的人类基因仍然没有充分或没有实验证据证明它们在蛋白质水平上的翻译。它们被归类为缺失蛋白(PE2 - PE4)。为了进一步实现HPP的目标,开发了两步生物信息学策略,解决了如何利用与缺失蛋白相对应的SRMAtlas肽作为唯一参考,以探索它们在GPM中的天然对应物。第一步,我们在GPM中搜索与缺失蛋白相对应的SRMAtlas肽,在35个不同的蛋白质组学研究中,新发现了51个缺失蛋白。第二步,我们根据合成肽和天然肽在SRMAtlas和GPM中的谱图进行比对,验证这些新发现的缺失蛋白。结果表明,经谱图匹配验证23个缺失蛋白中含有≥2个肽段。
二、 研究背景
人类蛋白质组计划是一项旨在对人类基因组中编码的所有人类蛋白质进行编目的世界性项目,通过对每个已知的人类基因序列识别至少一种蛋白质,验证人类蛋白的存在。根据neXtProt数据库最新版本,17694个蛋白质(89.26%)在蛋白质水平上有足够的实验证据(PE1),而2129个蛋白质(10.74%)被认为是缺失的(PE2-PE4),没有实验证据。
缺乏实验证据的原因有:1、没有表达(沉默基因);2、在某些组织中没有表达;3、表达水平极低;4、具有极端的物化性质,影响蛋白的溶解和提取;5、存在未知修饰
目前对缺失蛋白的发现速度很慢。GPM(Global Proteome Machine)可重新分析与Peptide Atlas不同的LC-MS/MS数据,综合利用它们的蛋白质组数据有可能加速缺失蛋白的发现。
SRMAtlas是迄今为止人类蛋白质组中最全面的实验资源,拥有166174个肽的实验数据,几乎涵盖所有缺失蛋白,本研究的主要目的是利用与缺失蛋白相对应的SRMAtlas肽的实验数据作为参考,以探索它们在GPM中的天然对应物。
三、 实验设计
分为两步策略:1、在GPM中寻找与缺失蛋白对应的SRMAtlas肽段(基于肽段一级氨基酸序列);2、基于SRMAtlas中的合成肽段和GPM中的天然肽段的谱图匹配验证新检测到的蛋白质肽段
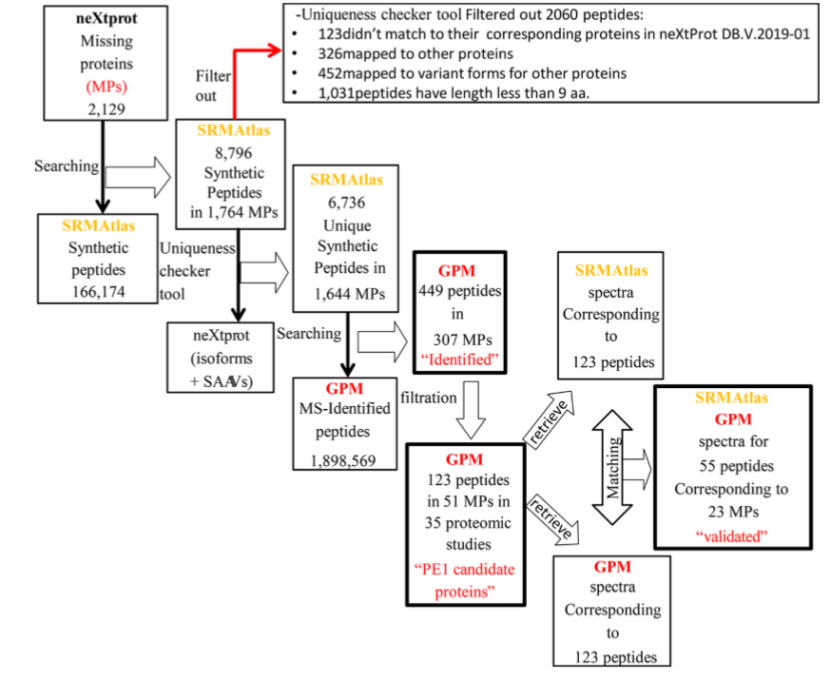
四、 研究成果
1、 在SRMAtlas数据库中寻找与缺失蛋白相对应的肽段,利用最新的neXtProt缺失蛋白数据库,在SRMAtlas中检索到与1764个缺失蛋白相对应的8796个肽段,。
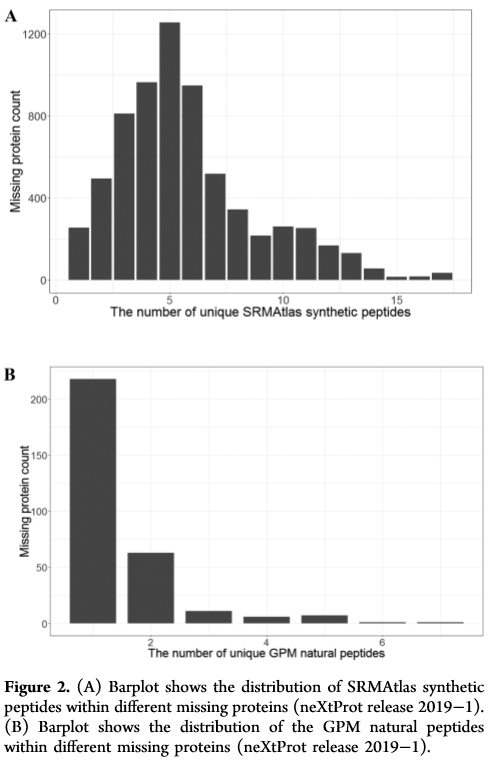
2、 利用neXtProt中的肽段唯一性检测工具检测与缺失蛋白相对应的肽段的唯一性,结果表明与1644个缺失蛋白相对应的6736个肽是长度≥9aa的唯一性肽段(table s1),2060个肽段被过滤掉。对6736个肽段的分布研究表明超过96%的是至少含有2个肽段(fig2A),这表明SRMAtlas数据库中不同蛋白质组学的研究对验证缺失蛋白的存在是至关重要的。
3、 使用SRMAtlas数据检测和验证GPM中的人类缺失蛋白,基于氨基酸序列,编写了一个perl脚本在GPM中的1898569个肽中检索缺失蛋白对应的SRMAtlas中的肽。脚本返回匹配到的肽及其在GPM和SRMAtlas中的注释。结果是在GPM中检测到与307个缺失蛋白对应的449个肽段。89个缺失蛋白拥有至少2个天然肽段(fig2B),对89个缺失蛋白的的谱数中位数为每肽23个谱,一个肽段的最小谱数为5个谱。(fig3A)
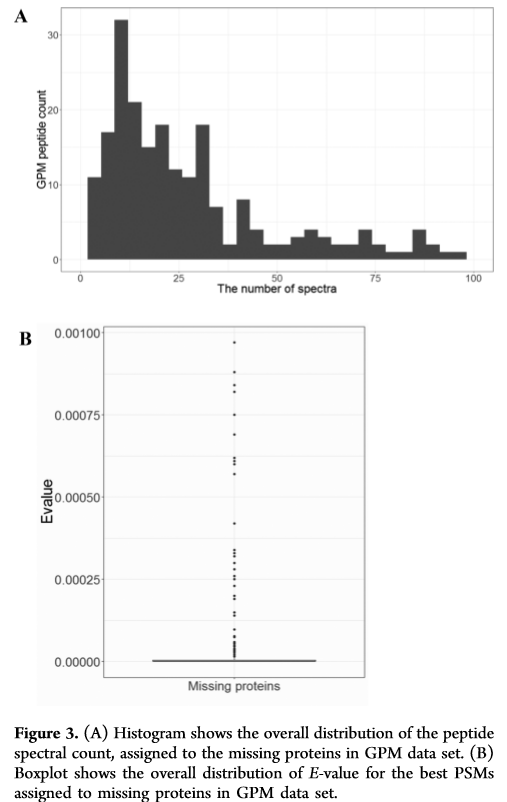
另外,与GPM数据库中发现的缺失蛋白匹配的最佳PSMs的中位E-value是2400x10-8,最大E-value是9700x10-4(fig3B)。
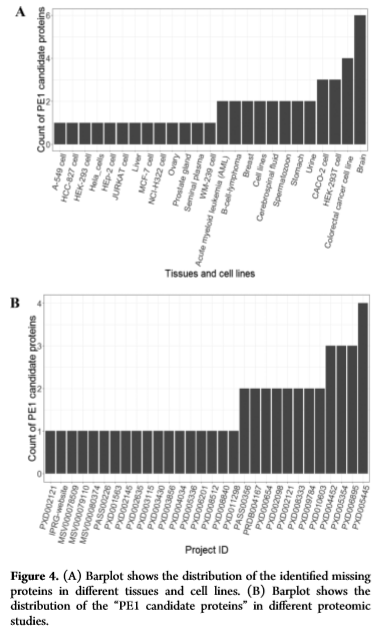
4、 一些候选的PE1蛋白仅在某些蛋白质组学实验中被检出,有趣的是,观察这些蛋白在不同组织和细胞系中的分布发现(fig4A),大脑是缺失蛋白最易被检出的区域之一,在一项关于人脑的研究中有6个缺失蛋白被明确。另外4个缺失蛋白在HEK293和HEK293 T细胞中被发现,表明这些细胞系可能为缺失蛋白的表达提供了舒适的环境(fig4B)。
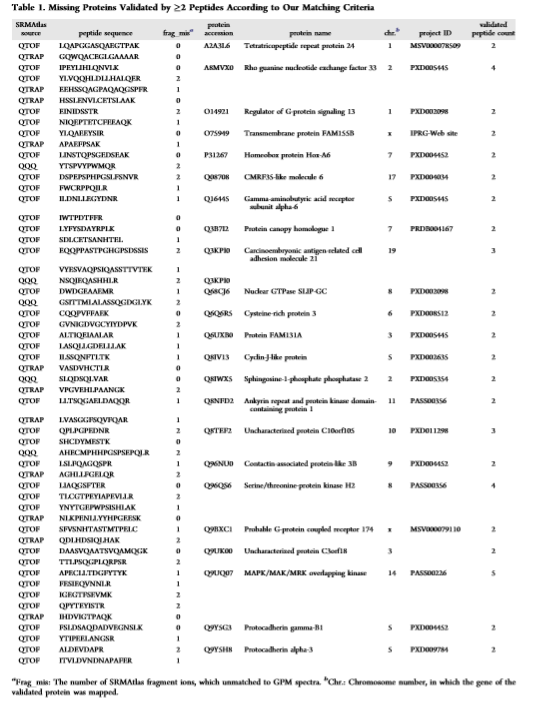
5、检索SRMAtlas中“PE1候选蛋白”的光谱。在SRMAtlas中,通过SRMAtlas查询工具搜索“PE1候选蛋白”对应的肽段,从不同的质谱源(QQQ、QTOF和QTRAP)检索这些肽段的片段离子。获得了123个与缺失蛋白对应的光谱。在母离子水平。我们检查了GPM和SRMAtlas的一对光谱是否与具有相同母离子电荷和相同m/ z的相同肽序列在0.01 Da差异内匹配(fig5A)。结果表明,从QTOF、QQQ和QTRAP中分别得到的65、54和73个肽段光谱与GPM中的天然对应物光谱匹配良好。在碎片离子水平,与23个缺失蛋白对应的55个多肽有≥2个多肽 (表1)。值得注意的是,所有QQQ、QTRAP和QTOF光谱中,23个缺失蛋白所对应的碎片离子中,有74%存在m/z的0.02 Da的差异。
五、 结论讨论
SRMAtlas是唯一一个含有实验数据的数据库资源,这些实验数据都是针对那些与缺失蛋白质相关的肽段。利用这些数据对缺失蛋白的发现有重要作用。在GPM中搜索SRMAtlas中与缺失蛋白对应的肽,发现了123个天然肽段,它们与51个缺失蛋白匹配。此外,通过匹配GPM和SRMAtlas之间新发现的缺失蛋白的MS/MS谱,这51个缺失蛋白中的23拥有≥2个肽段且≥9aa。最后,我们建议,挖掘更多的蛋白质组学研究以外的其他分析PeptideAtlas将帮助我们发现更多缺失蛋白
Journal of Proteome Research | Utilization of the Proteome Data Deposited in SRMAtlas for Validating the Existence of the Human Missing Proteins in GPM (解读人:梁嘉琪)的更多相关文章
- Journal of Proteome Research | 人类牙槽骨蛋白的蛋白质组学和n端分析:改进的蛋白质提取方法和LysargiNase消化策略增加了蛋白质组的覆盖率和缺失蛋白的识别 | (解读人:卜繁宇)
文献名:Proteomic and N-Terminomic TAILS Analyses of Human Alveolar Bone Proteins: Improved Protein Extr ...
- Journal of Proteome Research | SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants for proteogenomic interpretation | SAAV的识别、功能注释和检索 | (解读人:徐洪凯)
文献名:SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants fo ...
- Journal of Proteome Research | iHPDM: In Silico Human Proteome Digestion Map with Proteolytic Peptide Analysis and Graphical Visualizations(iHPDM: 人类蛋白质组理论酶解图谱的水解肽段分析和可视化展示)| (解读人:邓亚美)
文献名:iHPDM: In Silico Human Proteome Digestion Map with Proteolytic Peptide Analysis and Graphical Vi ...
- Journal of Proteome Research | Down-Regulation of a Male-Specific H3K4 Demethylase, KDM5D, Impairs Cardiomyocyte Differentiation (男性特有的H3K4脱甲基酶基因(KDM5D)下调会损伤心肌细胞分化) | (解读人:徐宁)
文献名:Down-Regulation of a Male-Specific H3K4 Demethylase, KDM5D, Impairs Cardiomyocyte Differentiatio ...
- Journal of Proteome Research | Quantitative Subcellular Proteomics of the Orbitofrontal Cortex of Schizophrenia Patients (精神分裂症病人眶额叶皮层亚细胞结构的定量蛋白质组学研究)(解读人:王聚)
期刊名:Journal of Proteome Research 发表时间:(2019年10月) IF:3.78 单位: 里约热内卢联邦大学 坎皮纳斯州立大学 坎皮纳斯州立大学神经生物学中心 卡拉博大 ...
- Journal of Proteome Research | Proteomic Profiling of Rhabdomyosarcoma-Derived Exosomes Yield Insights into Their Functional Role in Paracrine Signaling (解读人:孙国莹)
文献名:Proteomic Profiling of Rhabdomyosarcoma-Derived Exosomes Yield Insights into Their Functional Ro ...
- Journal of Proteome Research | Global Proteomic Analysis of Lysine Succinylation in Zebrafish (Danio rerio) (解读人:关姣)
文献名:Global Proteomic Analysis of Lysine Succinylation in Zebrafish (Danio rerio)(斑马鱼赖氨酸琥珀酰化的全球蛋白质组学分 ...
- Journal of Proteome Research | An automated ‘cells-to-peptides’ sample preparation workflow for high-throughput, quantitative proteomic assays of microbes (解读人:陈浩)
文献名:An automated ‘cells-to-peptides’ sample preparation workflow for high-throughput, quantitative p ...
- Journal of Proteome Research | Proteomic analysis of Rhizobium favelukesii LPU83 in response to acid stress.(酸胁迫下根瘤菌LPU83(Rhizobium favelukesii)的蛋白质组学分析)(解读人:丑天胜)
文献名:Proteomic analysis of Rhizobium favelukesii LPU83 in response to acid stress.(酸胁迫下根瘤菌LPU83(Rhizo ...
随机推荐
- jdk源码理解-String类
String类的理解 简记录一下对于jdk的学习,做一下记录,会持续补充,不断学习,加油 1.String的hash值的计算方法. hash值的计算方法多种多样,jdk中String的计算方法如下,比 ...
- hql错误:No data type for node: org.hibernate.hql.ast.tree.IdentNode
今天写了一个查询,用的是hql,数据库是mysql.多表联查,结果报错了报: \-[IDENT] IdentNode: 'routerNumber' {originalText=routerNumbe ...
- 吴裕雄--天生自然 R语言开发学习:回归(续三)
#------------------------------------------------------------# # R in Action (2nd ed): Chapter 8 # # ...
- python字符串复制的几种方法
>>> list1 = [1,2] >>> id(list1) 50081032 >>> list2 = list1.copy() >> ...
- 使用Xshell进行vi编辑时,按下end、home和Delete不能使用,解决解决办法
使用Xshell连接到Linux进行vi编辑时,进入编辑模式,按下end键,光标无法移到行位,home也不能到行首,其它的Delete键也是不能使用,如何解决? Xshell选项设置如下: 文件→属性 ...
- 用Python搭建简单的HTTP服务 · Zhangxu's Blog
分享一个快速用Python搭建简单的HTTP服务的方法. 平时我们可能有需要,传输某个文件到手机,或者工作中某台服务器的电脑. 假如这个手机是个测试手机/服务器,并没有微信QQ之类的软件,而且你也不想 ...
- 【转载】(String)、toString、String.valueOf的区别
用于个人参考,查看请前往原地址http://blog.csdn.net/springk/article/details/6414017 问题讨论http://bbs.csdn.net/topics/2 ...
- 【自己的下载平台】搭建aria2网站
前言 本文章将带你搭建一个自己的服务器下载平台:aria2,它的用途是什么? 下载用途 百度网盘 普通文件 迅雷种子 等等 准备工具 服务器连接软件xshell或者putty 一台服务器 安装宝塔面板 ...
- PyQt5之俄罗斯方块
上个礼拜有个需求,对csv里的数据按条件进行拆分计算.一想到要做计算,少不了pandas.还有个要求最好是生成命令行工具或者带有界面. 于是尝试下,使用PyQt5做了个简单的UI界面给程序包个壳子,然 ...
- CMSampleBufferRef解析
CMTime:64位的value,32位的scale, media的时间格式 CMVideoFormatDesc:video的格式,包括宽高.颜色空间.编码格式.SPS.PPS CVPixelBuff ...
