基于ggplot2的解剖图和组织模块可视化
摘要
将数据显示到解剖结构上,是一种可以快速观察组织相关信息的便捷技术。然而,绘制组织是一项复杂的任务(a complex task),需要解剖学和艺术方面的专业知识。虽然已经存在可用于在解剖图上显示基因表达的基于网络的应用技术,但是其他非遗传学科(non-genetic disciplines)缺乏类似的工具。此外,基于网络的工具通常缺乏与编程语言(如 R)中的包相关联的模块性( lack the modularity associated with packages in programming languages)。这里我介绍 gganatogram,一个根据 ggplot2 的图形语法和来自 Expression Atlas 公众可用解剖图的组合,来绘制模块化物种解剖图的 R 包。这种组合允许快速,简单,模块化和可重复地生成解剖图。仅使用一个命令以及一个具有组织名称,分组,颜色和值的数据框,该工具就可以使用户能够以特定的颜色可视化特定的人体和小鼠组织,或者根据变量进行分组,或显示特定的值,例如基因-表达(gene-expression),药代动力学(pharmacokinetics)或所选组织之间的细菌负荷(bacterial load)。我希望这个工具对更广泛的生物科学界有用。也欢迎社区成员提交额外的解剖图,并将其纳入该软件包中。
介绍
在多细胞生物中有效地显示组织信息可能是一个费力且耗时的过程(a laborious and time consuming process)。 研究人员通常希望展示值之间的差异,例如一个生物体组织之间,或不同组中具有相似组织之间的基因表达值或药代动力学值。
虽然条形图和热图提供了组间差异的信息视图,但很难立即观察到生物学意义(图 1a-b)。 与解剖图相比,后者更易于快速发现组织或组之间的差异,并可立即为这些观察提供生物学背景(biological context)(图 1c)。 这还有一个额外的好处,即无论是阅读论文还是参加讲座,观众都必须花费更少的时间和精力来掌握结果。
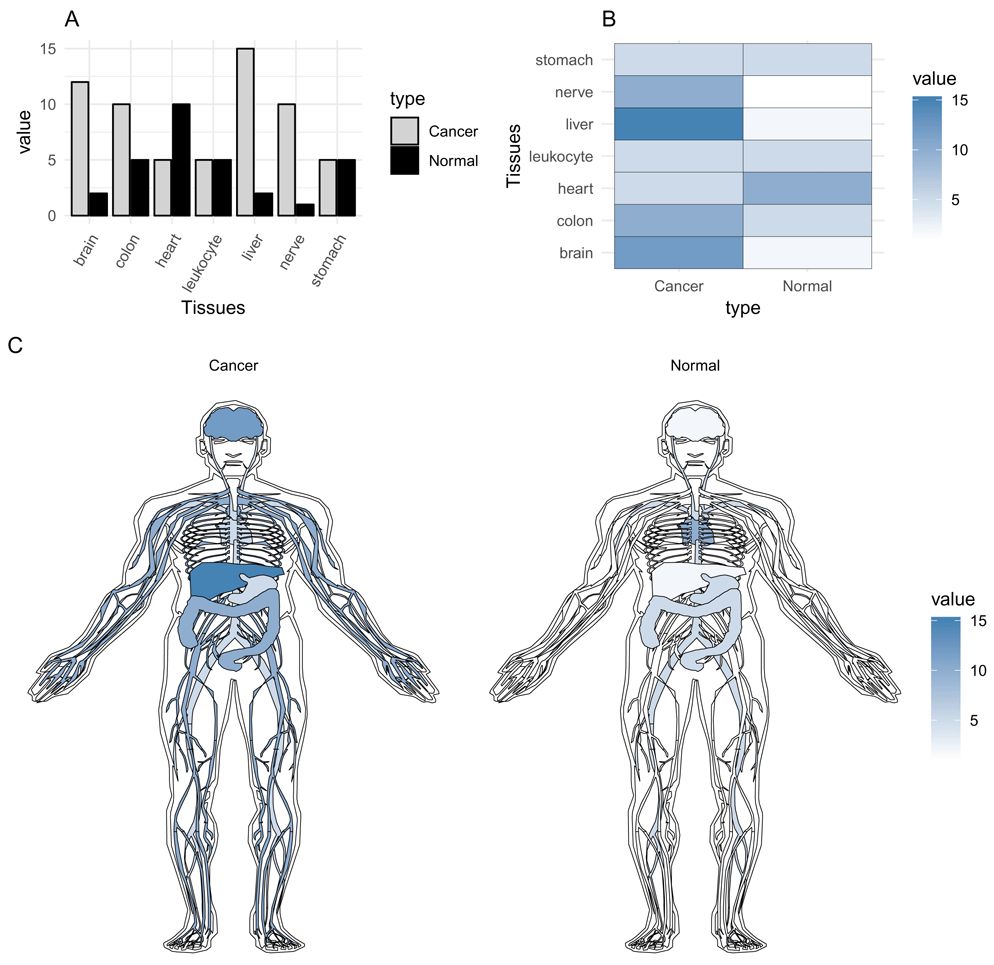
Figure 1. Comparison between barplot (top left), heatmap (top right), and anatogram (bottom) to display tissue values between groups.
已经存在几种在不同组织中显示基因表达的在线工具(如 Expression Atlas、Semantic Body Browser、TISSUES 2.0)。 虽然这些工具提供了关于各种组织和生物体中基因表达的重要信息,但除遗传学之外的其他学科由于(缺少)对基因的关注而无法利用这些应用(other disciplines besides genetics are unable to utilise these applications due to the focus on genes)。 此外,这些工具通常只包含一组可预测的预定义实验,导致难以呈现我们自己的数据。 还有其他值得注意是,使用这些工具,重建绘图或从结果中自动创建绘图可能很费力。
这里介绍的 gganatogram,是一个利用 Expression Atlas 中公开提供的鼠和人体解剖图,基于 ggplot2 的开源 R 软件包。 使用此软件包,任何 R 用户都可以轻松快速地查看具有指定颜色,组和值的解剖图。 使用 ggplot2 中熟悉的语法,该程序可用于模块化解剖图的生成。
方法
实现(Implementation)
gganatogram 存储在 neuroconductor,一个用于快速测试(rapid testing)和可重现计算成像软件(reproducible computational imaging software)传播的开源平台上, 其开发版本可以在 https://github.com/jespermaag/gganatogram 找到,它允许社区通过创建坐标文件来发布包的 issues,提交请求或添加解剖图。
source("https://neuroconductor.org/neurocLite.R")
neuro_install("gganatogram", release = "stable",
release_repo = latest_neuroc_release(release = "stable"))
开发版本可以从 github 安装:
devtools::install_github("jespermaag/gganatogram")
简而言之,为了生成包含所有组织坐标的主列表对象,作者从 Expression Atlas 下载了SVG文件(可从 gganatogram GitHub 中获取)并使用自定义的 python 脚本(可从 GitHub 中获得)处理它们。 该脚本通过 SVG 文件来提取名称,坐标和 SVG 转换信息。 然后在 R 中对它们进行后处理以创建构成组织坐标的 rda 文件。
操作(Operation)
gganatogram 需要满足 R≥3.0.0,ggplot2 v.3.0.0 和 ggpolypath v.0.1.0 。 该程序应该能够在任何具有 R 系统要求的计算机上运行。可以使用包含器官名称,颜色,类型或值的基本 data.frame 生成图形。 根据 data.frame 的顺序,一次一个地绘制器官。 每个连续行的组织将在前一个上面分层。 gganatogram 包提供了四个这样的 data.frame,其中包含可用于绘图的所有组织,每个人和小鼠一个,并按性别划分。
hgMale_key, hgFemale_key, mmMale_key, mmFemale_key
这些数据框已经指定了颜色,类型和指定的随机数,以便于开始绘图。
head(hgFemale_key)
organ colour type value
1 pancreas orange digestion 10.373146
2 liver orange digestion 19.723172
3 colon orange digestion 14.853335
4 bone_marrow #41ab5d other 19.681587
5 urinary_bladder orange digestion 14.914273
6 stomach orange digestion 2.667599
主要功能是 gganatogram()。 默认情况下,没有任何参数,它会绘制具有标准 ggplot2 参数的男性人的轮廓。 通过添加几个选项,可以快速更改为女性,通过选定的颜色填充指定的器官,或者根据值填充器官(图2)。
library(gganatogram)
library(gridExtra)
organPlot <- data.frame(organ = c("heart", "leukocyte", "nerve", "brain", "liver", "stomach", "colon"),
type = c("circulation", "circulation", "nervous␣system", "nervous␣system", "digestion", "digestion", "digestion"),
colour = c("red", "red", "purple", "purple", "orange", "orange", "orange"),
value = c(10, 5, 1, 8, 10, 5, 10),
stringsAsFactors=F)
A <- gganatogram()
B <- gganatogram(fillOutline="#a6bddb", sex="female") + theme_void()
C <- gganatogram(data=organPlot, fillOutline="#a6bddb", organism="human",
sex="female", fill="colour")+ theme_void()
D <- gganatogram(data=organPlot, fillOutline="#a6bddb", organism="human",
sex="female", fill="value")+ theme_void()
grid.arrange(A, B, C, D, ncol=4)
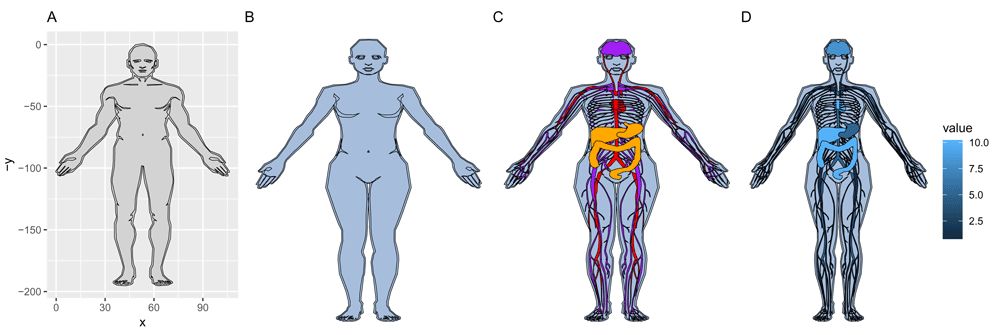
Figure 2. (A) Default plot generated by calling gganatogram(), (B) adding female, plotting specified organs by (C) colour, (D) value.
示例
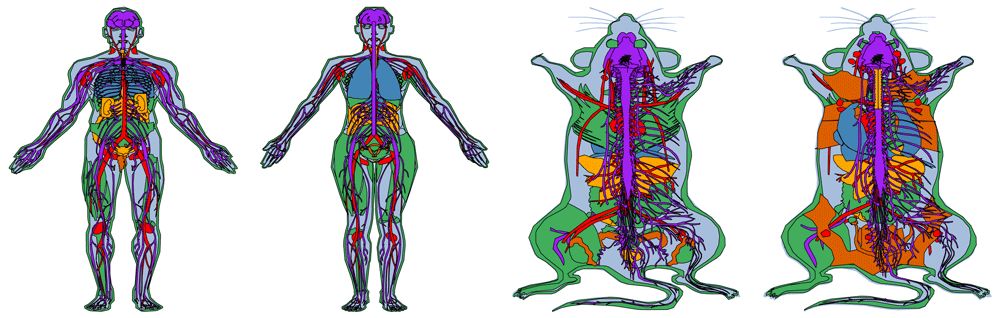
要绘制每个生物体的所有组织,请使用每个生物体和性别所提供的密钥文件。 这将按照每个 data frame 的顺序显示所有组织。 要更改器官彼此叠加的顺序,请重新排序数据框以使这些组织位于底部。
library(gganatogram)
library(gridExtra)
hgMale <- gganatogram(data=hgMale_key, fillOutline="#a6bddb", organism="human",
sex="male", fill="colour") + theme_void()
hgFemale <- gganatogram(data=hgFemale_key, fillOutline="#a6bddb",
organism="human", sex="female", fill="colour") + theme_void()
mmMale <- gganatogram(data=mmMale_key, fillOutline="#a6bddb", organism="mouse",
sex="male", fill="colour") + theme_void()
mmFemale <- gganatogram(data=mmFemale_key, outline = T, fillOutline="#a6bddb",
organism="mouse", sex="female", fill="colour") + theme_void()
grid.arrange(hgMale, hgFemale, mmMale, mmFemale, ncol=4)
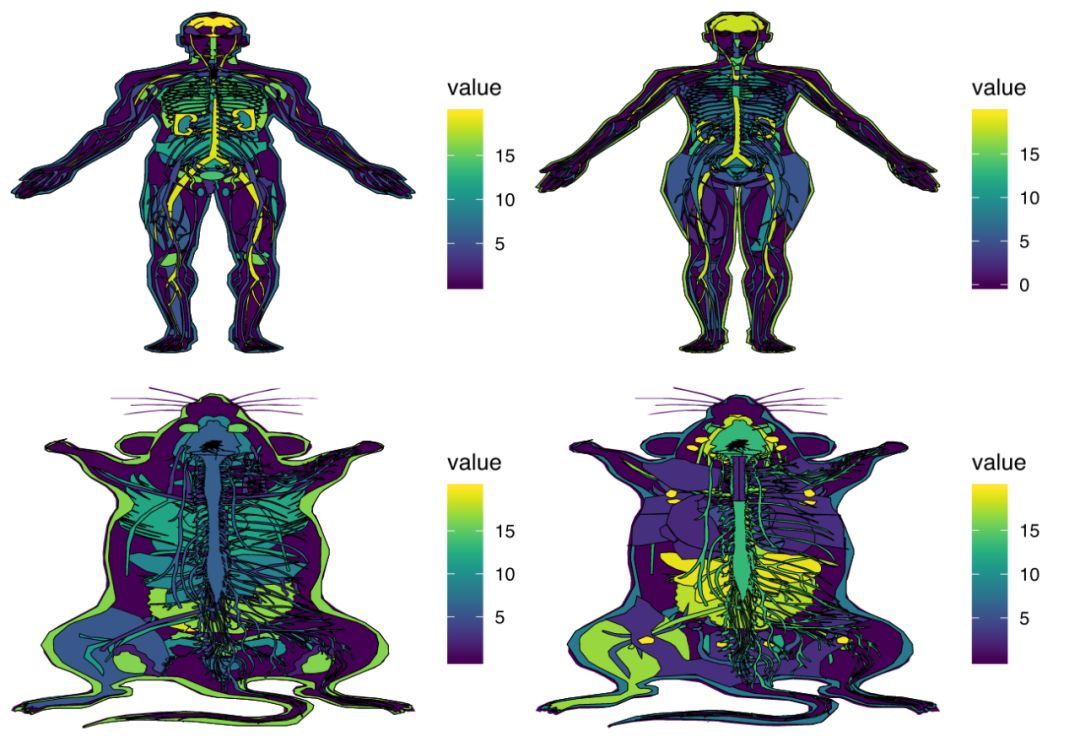
hgMale <- gganatogram(data=hgMale_key, fillOutline='#440154FF', organism='human', sex='male', fill="value") + theme_void() + scale_fill_viridis()
hgFemale <- gganatogram(data=hgFemale_key, fillOutline='#440154FF', organism='human', sex='female', fill="value") + theme_void() + scale_fill_viridis()
mmMale <- gganatogram(data=mmMale_key, fillOutline='#440154FF', organism='mouse', sex='male', fill="value") + theme_void() + scale_fill_viridis()
mmFemale <- gganatogram(data=mmFemale_key, outline = T, fillOutline='#440154FF', organism='mouse', sex='female', fill="value") +theme_void() + scale_fill_viridis()
grid.arrange(hgMale, hgFemale, mmMale, mmFemale, ncol=2)
要使用
gganatogram 函数,我们需要具有器官(organ)、颜色(colour)和相关值(value)的数据框(data frame)。
organPlot <- data.frame(organ = c("heart", "leukocyte", "nerve", "brain", "liver", "stomach", "colon"),
type = c("circulation", "circulation", "nervous system", "nervous system", "digestion", "digestion", "digestion"),
colour = c("red", "red", "purple", "purple", "orange", "orange", "orange"),
value = c(10, 5, 1, 8, 2, 5, 5),
stringsAsFactors=F)
head(organPlot)
organ type colour value
1 heart circulation red 10
2 leukocyte circulation red 5
3 nerve nervous system purple 1
4 brain nervous system purple 8
5 liver digestion orange 2
6 stomach digestion orange 5
使用 gganatogram 函数,根据颜色填充器官。
gganatogram(data=organPlot, fillOutline='#a6bddb', organism='human', sex='male', fill="colour")
当然,我们可以使用
ggplot 主题和函数来调整绘图。
gganatogram(data=organPlot, fillOutline='#a6bddb', organism='human', sex='male', fill="colour") +
theme_void()
我们也可以根据给予每个器官的值来填充各个组织的不同颜色;甚至,gganatogram 可以绘制细胞亚结构的相关组织图;或者是其他物种对应的解剖表达图谱。
更多
gganatogram 解剖图谱绘制示例可以参考其在 Github 的介绍,
https://github.com/jespermaag/gganatogram, 感兴趣的童鞋可以去尝试研究一下。
总的来说,
gganatogram 是一款强大的 R 包,它可以根据
ggplot2 和
Expression Atlas 的信息轻松地显示解剖图,这些工具组合成了一个更加强大的工具来绘制和显示组织信息。
参考资料:
Maag JLV. gganatogram: An R package for modular visualisation of anatograms and tissues based on ggplot2 [version 1; referees: 1 approved]. F1000Research 2018, 7:1576 (doi: 10.12688/f1000research.16409.1)
Jesper Maag,Create anatograms using ggplot2,Github
——The End——

本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
基于ggplot2的解剖图和组织模块可视化的更多相关文章
- 基于Asterisk的VoIP开发指南——Asterisk 模块编写指南(1)
原文:基于Asterisk的VoIP开发指南--Asterisk 模块编写指南(1) 1 开源项目概述 Asterisk是一个开源的软件包,通常运行在Linux操作系统平台上.Asterisk可以用三 ...
- 基于MarkDown和Github图床以及SourceTree的一站式文章编辑和发布
标题: 基于MarkDown和Github图床以及SourceTree的一站式文章编辑和发布 作者: 梦幻之心星 sky-seeker@qq.com 标签: [MarkDown,Github,图床,S ...
- 论文解读丨基于局部特征保留的图卷积神经网络架构(LPD-GCN)
摘要:本文提出一种基于局部特征保留的图卷积网络架构,与最新的对比算法相比,该方法在多个数据集上的图分类性能得到大幅度提升,泛化性能也得到了改善. 本文分享自华为云社区<论文解读:基于局部特征保留 ...
- matlab中,在灰度解剖图上叠加阈值图,by by DR. Rajeev Raizada
1.参考 reference 1. tutorial主页:http://www.bcs.rochester.edu/people/raizada/fmri-matlab.htm. 2.speech_b ...
- 一款基于jQuery饼状图比例分布数据报表
今天给大家带来一款基于jQuery饼状图比例分布数据报表.这款报表插件适用浏览器:IE8.360.FireFox.Chrome.Safari.Opera.傲游.搜狗.世界之窗.效果图如下: 在线预览 ...
- 第一篇:R语言数据可视化概述(基于ggplot2)
前言 ggplot2是R语言最为强大的作图软件包,强于其自成一派的数据可视化理念.当熟悉了ggplot2的基本套路后,数据可视化工作将变得非常轻松而有条理. 本文主要对ggplot2的可视化理念及开发 ...
- [python] 基于matplotlib实现圆环图的绘制
圆环图本质上是一个中间切出一块区域的饼状图.可以使用python和matplotlib库来实现.本文主要介绍基于matplotlib实现圆环图.本文所有代码见:Python-Study-Notes # ...
- 一个画组织解剖图R包
地址: https://github.com/jespermaag/gganatogram
- C# Winform开发框架模块图(平台核心模块+示例模块)
企业版V4.0 - 模块图 企业版V4.0 - 项目解决方案 Client/Server构架,有两个解决方案: 客户端解决方案说明: 服务端解决方案说明: C/S系统开发框架- ...
- 基于Neptune开发板的键盘蓝牙模块DIY指南
目录: 1.下载开发板程序2.安装USB串口(CH340)驱动3.安装烧写工具4.烧写开发板程序 本期我们带来基于润和Neptune开发板(以下简称Neptune开发板)的键盘蓝牙模块DIY指南,利用 ...
随机推荐
- C#笔记之泛型
泛型是C#中应用极为广泛的一种语法,本篇文章将详细介绍泛型的定义.使用.性能等. 一.什么是泛型 首先需要记住的是,泛型是.NET 2.0推出的语法,这样的话,泛型基本可以用于所有程序的开发,而不需要 ...
- Django笔记九之model查询filter、exclude、annotate、order_by
在接下来四五篇笔记中,将介绍 model 查询方法的各个细节,为我们的查询操作提供各种便利. 本篇笔记将介绍惰性查找.filter.exclude.annotate等方法,目录如下: 惰性查找 fil ...
- jquery中判断复选框有没有被选上
页面部分: <input type="checkbox" id="cbx" /><label for="cbx">点 ...
- QML 界面切换的几种方法(带示例代码)
QML 界面切换的几种方法(带示例代码)
- JavaScript中计时器requestAnimationFrame、setTimeout、setInterval、setImmediate的使用和区别
在JavaScript中,我们经常使用requestAnimationFrame.setTimeout.setInterval和setImmediate来控制代码的执行时机.它们各有特点和适用场景: ...
- TypeScript 学习笔记 — 自定义类型:部分属性可选,反选 key,求对象交差并补集等(十三)
目录 将部分属性变为可选属性 根据值的类型 反选 key 写法一:基础原理写法,使用不同的内置类型,Pick 和 Omit 写法二:基础原理写法,使用 Pick 内置类型 + 传参的方式 写法三:使用 ...
- kubernetes(k8s) 中安装kuboard面板
kubernetes(k8s) 中安装kuboard面板 01 - 背景及安装 Kuboard 是一款专为 Kubernetes 设计的免费管理界面,兼容 Kubernetes 版本 1.13 及以上 ...
- [Shell] Windows上支持Linux Shell的工具/方法
0 概述 1 方式一 : Windows Terminal 局限性: 不支持 xargs等命令 支持sed,find等命令 安装方式 安装Windows Terminal的最简单方法是通过Micros ...
- [Java]【异常处理】在项目开发中的通用规范
这一问题,已苦恼多日,上述答案系近整整2日广泛阅读书籍.博客.网络问答后,归纳并苦思而成. 本博文仅罗列总的观点,各观点的具体解释,可私信Word笔记. 如需转载or复制时,请注明出处. 本博文同步发 ...
- 【Vue项目】尚品汇(三)Home模块+Floor模块+Swiper轮播图
写在前面 今天是7.23,这一篇内容主要完成了Home模块和部分Search模块的开发,主要是使用了swiper轮播图插件获取vuex仓库数据展示组件以及其他信息. 1 Search模块 1.1 Se ...
