DNA比对算法:BWT
DNA比对算法:BWT
BWT算法,实质上是前缀树的一种实现。那么什么是前缀树呢?
一、前缀树
对于问题p in S?如果S=rpq,那么p为S前缀rp的一个后缀。
于是,为了判断p in S 是否成立,我们找到S的所有前缀,然后逐一判断p是不是它们的后缀。为了加快效率,我们将所有的前缀建成一颗树,这棵树便是前缀树。下面,我们举例说明前缀树的建立过程和如何使用前缀树进行模式匹配。
前缀树的建立
假设S='acaacg',p='aac',那么我们首先找到S的所有前缀,如下
- a
- ac
- aca
- acaa
- acaac
- acaacg
于是,我们将这些前缀翻转过来,然后建立为一颗字典树,如下图
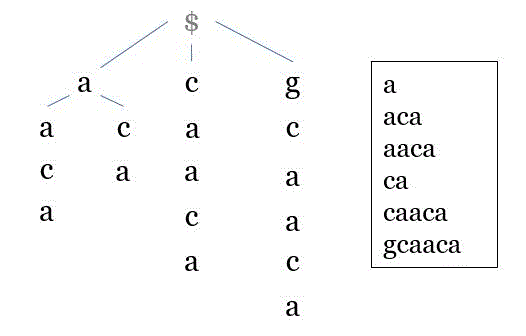
模式匹配
\(p='aac'\),令\(p'=caa\)(即p的翻转)。显然,现在只需进行一次树的搜索,即可完成匹配。
如果在判断p in S 的同时,还需要得到p 在S 中的位置,那么只需在建树的时候,将每个字符的索引加上,例如
当然,也可以不保存索引,每次模式匹配结束时,沿着当前节点走下去,一直到为S[0]。
在节点中添加数字,有其他用处,详见我的另一篇博文广义后缀树的简介。
评价
我们可以看到,相对于常规的匹配算法,前缀树时间复杂度比较小,但占用空间较大。下面要说的BWT算法,就是解决这个问题的。
二、构建BWT(S)
仍然,以S='acaacg'为例。
- 令S1=S+'\$'='acaacg\$';
- 循环左移S1 6次,得到S2,S3,S4,S5,S6,S7;
- 'acaacg\$'
- 'caacg\$a'
- 'aacg\$ac'
- 'acg\$aca'
- 'cg\$acaa'
- 'g\$acaac'
- '\$acaacg'
- 对S1到S7按字典序排序(\$字符的字典序最小),取每个串的最后一个字符,连成一个序列'gc\$aaac'。于是为BWT(S)='gc\$aaac'。
- '\$acaacg'
- 'aacg\$ac'
- 'acaacg\$'
- 'acg\$aca'
- 'caacg\$a'
- 'cg\$acaa'
- 'g\$acaac'
也许,到这里,你还不清楚BWT变换和前缀树,有什么关系。那就接着往下看吧。
三、使用BWT,进行模式匹配
我们已经知道BWT(S)='gc\$aaac',对BWT(S)中的字符进行排序得到S'='\$aaaccg',得到下图形式的矩阵。
这个矩阵看起来,有些规律,但是又很奇怪。下面通过复原S的过程,我们来理解以下这个矩阵。
复原S
这个过程用语言描述比较麻烦,直接看图
按照图中(1)到(7)步,我们即可得到'$gcaaca',于是S='acaacg'。
其中,斜线表示是,我们找到最后一列的某个符号,然后跳至这个符号在第一列的位置。比如,在第(2)步中,最后一列为第2个c,我们跳到第一行中第2个c的位置。
模式匹配
p='aac',令\(p'='caa'\),选取c作为起点,由于S中有两个c,因此有两种可能 的匹配。
- 从第一个c出发
- 从第二个c出发
因此,在方案2得到p',因此p in S是正确的。
几个问题
- 问题一:如何得到某个符号,在本列中是第几个?
显然,我们可以使用一个数组来保存。例如,对于'$gcaaca',数组a=[1,1,1,1,2,2,3]。
$ g c a a c a
[1,1,1,1,2,2,3]
但是,还有一种省空间的办法。我们只保存串'$gcaaca'中某些字符的位置,这些字符我们称为checkpoint。
- 问题二:如何得到模式p在S中的位置?
匹配模式串之后,继续运行,直至\$,但是这样比较耗时。
另一种办法,在BWT串中记录相应的偏移。这种办法空间开销比较大,也可以采取类似于checkpoint的方法,记录部分的偏移。
四、待研究的问题
- 如何快速得到一个串的BWT编码?
- 如何允许部分匹配?
题外话
DNA比对还有一类快速的办法——使用哈希。
DNA比对算法:BWT的更多相关文章
- LeetCode-Repeated DNA Sequences (位图算法减少内存)
Repeated DNA Sequences All DNA is composed of a series of nucleotides abbreviated as A, C, G, and T, ...
- 51nod 1445 变色DNA ( Bellman-Ford算法求单源最短路径)
1445 变色DNA 基准时间限制:1 秒 空间限制:131072 KB 分值: 40 难度:4级算法题 有一只特别的狼,它在每个夜晚会进行变色,研究发现它可以变成N种颜色之一,将这些颜色标号为0,1 ...
- HDU1560 DNA sequence —— IDA*算法
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=1560 DNA sequence Time Limit: 15000/5000 MS (Java/Oth ...
- 算法 - DNA搜索 - Ako Corasick
场景:从很长的字符串(输入字符串.DNA)中搜索大量固定字符串(字典.基因) 题目:Determining DNA Health | HackerRank 算法:Aho–Corasick algori ...
- 字符串与模式匹配算法(六):Needleman–Wunsch算法
一.Needleman-Wunsch 算法 尼德曼-翁施算法(英语:Needleman-Wunsch Algorithm)是基于生物信息学的知识来匹配蛋白序列或者DNA序列的算法.这是将动态算法应用于 ...
- 一个简单算法题引发的思考<DNA sorting>(about cin/template/new etc)
首先是昨天在北京大学oj网上看到一个简单的算法题目,虽然简单,但是如何完成一段高效.简洁.让人容易看懂的代码对于我这个基础不好,刚刚进入计算机行业的小白来说还是有意义的.而且在写代码的过程中,会发现自 ...
- 利用Needleman–Wunsch算法进行DNA序列全局比对
生物信息学原理作业第二弹:利用Needleman–Wunsch算法进行DNA序列全局比对. 具体原理:https://en.wikipedia.org/wiki/Needleman%E2%80%93W ...
- DNA binding motif比对算法
DNA binding motif比对算法 2012-08-31 ~ ADMIN 之前介绍了序列比对的一些算法.本节主要讲述motif(有人翻译成结构模式,但本文一律使用基模)的比对算法. 那么什么是 ...
- 算法:POJ1007 DNA sorting
这题比较简单,重点应该在如何减少循环次数. package practice; import java.io.BufferedInputStream; import java.util.Map; im ...
随机推荐
- 《连载 | 物联网框架ServerSuperIO教程》- 17.支持实时数据库,高并发保存测点数据。附:3.4 发布与版本更新说明。
1.C#跨平台物联网通讯框架ServerSuperIO(SSIO)介绍 <连载 | 物联网框架ServerSuperIO教程>1.4种通讯模式机制. <连载 | 物联网框架Serve ...
- 回答: 2017-03-19的关于css+div布局的疑问 2017-04-05
原问题为红色,回答为黑色 第一次布局一个静态网页,发现许多细节都需要自己探索,出现诸如以下问题: 1.布局问题:经常出现一个div被挤出来?做到一半少一个div布局? 布局之前,要点打好框架,明确每个 ...
- Xamarin+Prism开发之.net standard化
前面[Visual Studio 2017创建.net standard类库编译出错原因]已经解决.net standard类库的编译问题,现在可以正式进入主题了.作为.net 跨平台开发者也得跟上时 ...
- python_Tornado_web_框架_分页
如何实现web_框架_分页? -- 思考什么是xss跨站攻击? --别人把js代码提交到后台,然后从后台返回数据的时候,别人上传的js变成html中的代码, 就会插入别人的代码,带来极大的信息泄露的风 ...
- mac的终端为什么会显示git:(master),如何取消掉?
今天在终端误操作,在主目录下执行git init命令,结果杯具了, 总是出现这个提示. 各种搜索解决方案,终于退出了. 方法如下: 删掉.git目录: rm -rf ~/.git
- javaScript 基本类型之间转换
在Java中,基本类型之间的强制转换也不是这样的,比如,整数要转换成字符串,必须使用Integer.toString()静态方法或者String.valueOf()静态方法,把字符串转换为整数,必须使 ...
- jQuery animate()动画效果
1.jQuery动画效果 jQuery animate()方法用于创建自定义动画 $(selector).animate({params},speed,callback); //必需的 params ...
- PHP7中我们应该学习会用的新特性
PHP7于2015年11月正式发布,本次更新可谓是PHP的重要里程碑,它将带来显著的性能改进和新特性,并对之前版本的一些特性进行改进.本文小编将和大家一起来了解探讨PHP7中的新特性. 1. 标量类型 ...
- Caffe学习系列(四)之--训练自己的模型
前言: 本文章记录了我将自己的数据集处理并训练的流程,帮助一些刚入门的学习者,也记录自己的成长,万事起于忽微,量变引起质变. 正文: 一.流程 1)准备数据集 2)数据转换为lmdb格式 3)计算 ...
- 第六章 Hibernate关联映射
第六章 hibernate关联映射一.本章知识点分为2部分:1.关联关系:单向多对一关联关系,双向一对多关联关系(含一对多关联关系),多对多关联关系2.延迟加载:类级别加载策略,一对多加载策略,多对一 ...
