R EnhancedVolcano 绘制火山图
火山图是用于差异表达分析结果可视化的一种有效方法。今天,我们来介绍一个用于增强火山图绘制的强大 R 包:EnhancedVolcano ,该包拥有强大的绘图功能,用户可以简单的通过设置颜色、形状、大小和阴影等参数定义不同的绘图属性,此外通过可以通过添加连线的方式有效避免数据点之间的重叠现象。使用 EnhancedVocalno 包绘制的火山图基本可以直接用于文献发表,可以说非常简单又实用的一款神器了。
1. 下载与安装
R 版本:3.6.1。从 Bioconductor 中下载包:
if (!requireNamespace('BiocManager', quietly = TRUE))
install.packages('BiocManager')
BiocManager::install('EnhancedVolcano')
2. 简单使用
2.1 输入数据格式
首先,我们先来介绍一下 EnhancedVolcan o 输入数据格式。EnhancedVolcano 包可以使用多种差异算法(例如 DESeq2 等)的结果作为输入,数据中需包含 log2FC 、Pvalue 或(和) qvalue 结果,示例数据如下:
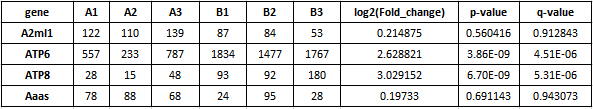
2.2 基础绘图
library(EnhancedVolcano)
res <- read.table(diffexpress,
sep="\t",
head=T,
row.names=1,
check.names=F,
quote="")
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4))
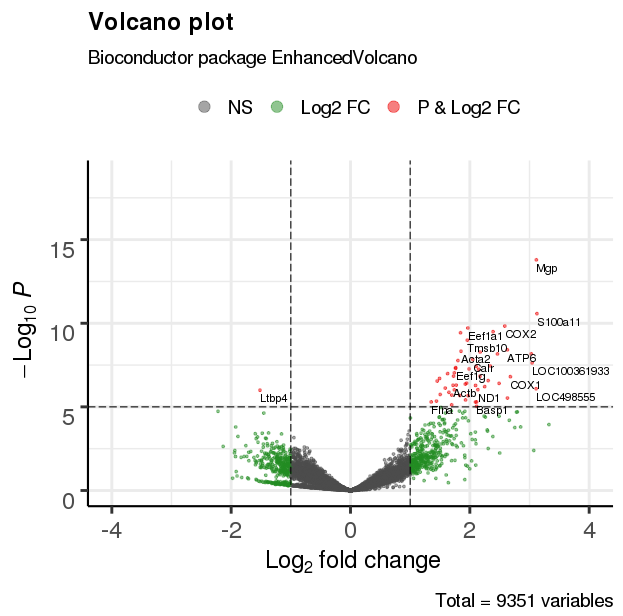
3、进阶功能
3.1 调整阈值,设置点及标签大小
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
transcriptPointSize = 3.0,
transcriptLabSize = 3.0)
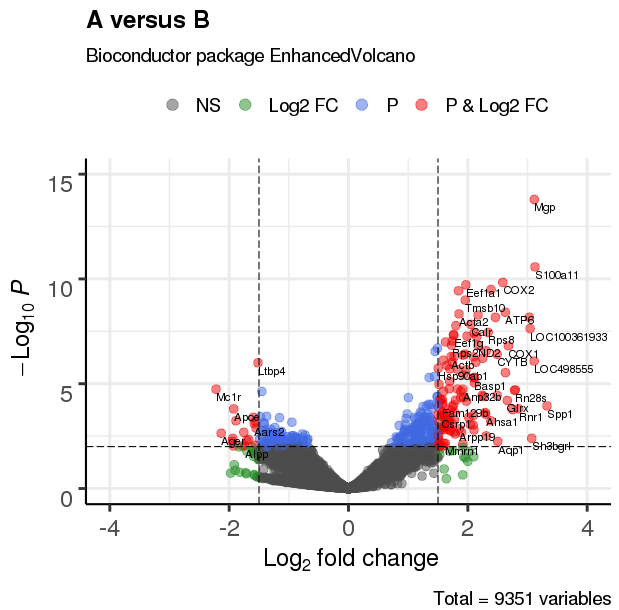
3.2 调整颜色及点透明度
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
col = c('black', 'black', 'black', 'red3'),
colAlpha = 1)
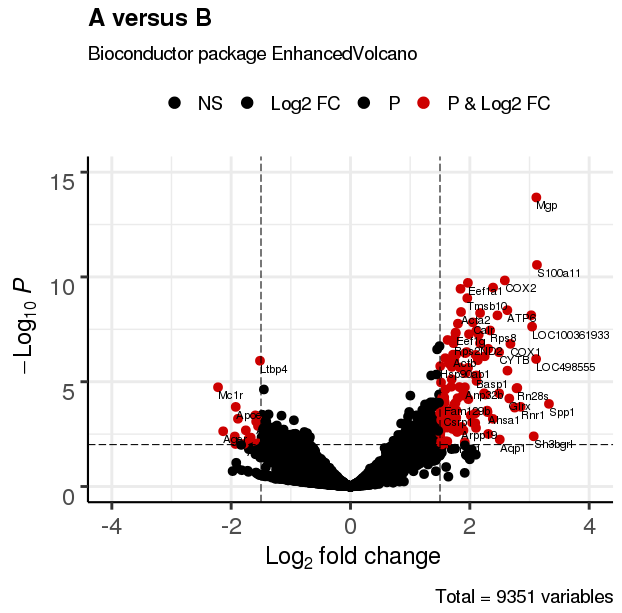
3.3 调整绘图点形状
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
#shape = 8, #点形状
shape = c(1, 4, 23, 25), #形状列表
colAlpha = 1)
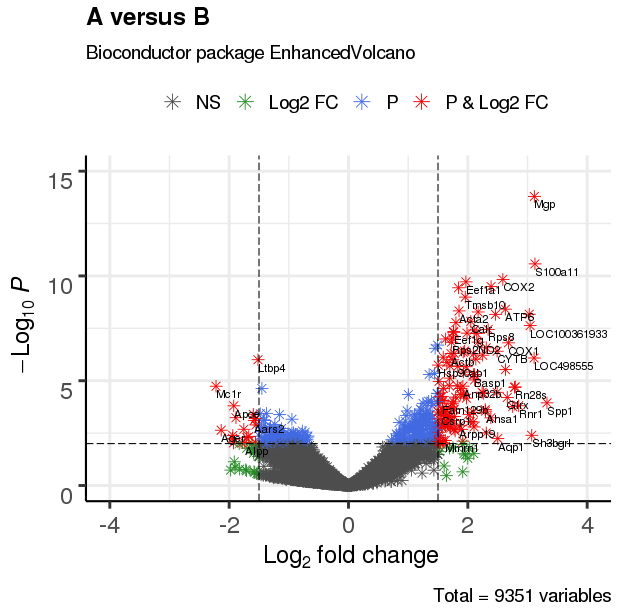
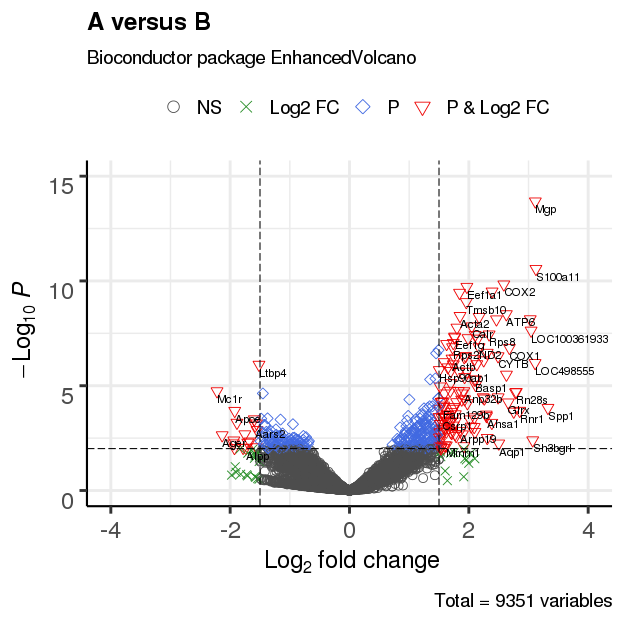
3.4 改变截止线及添加阈值线
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'blank',
cutoffLineCol = 'black',
cutoffLineWidth = 0.8,
hline = c(10e-4, 10e-8, 10e-12, 10e-15),
hlineCol = c('grey0', 'grey25','grey50','grey75'),
hlineType = 'longdash',
hlineWidth = 0.8,
gridlines.major = FALSE,
gridlines.minor = FALSE)
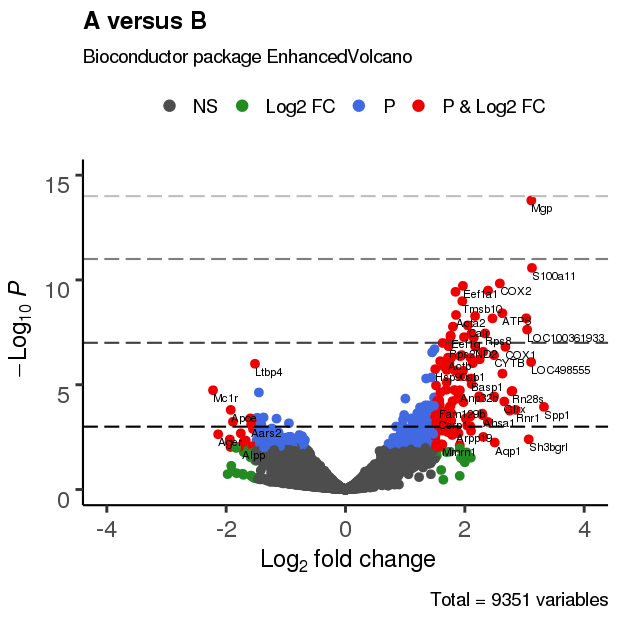
3.5 调整标注位置、大小及文字
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
legendLabSize = 16,
legendIconSize = 5.0)
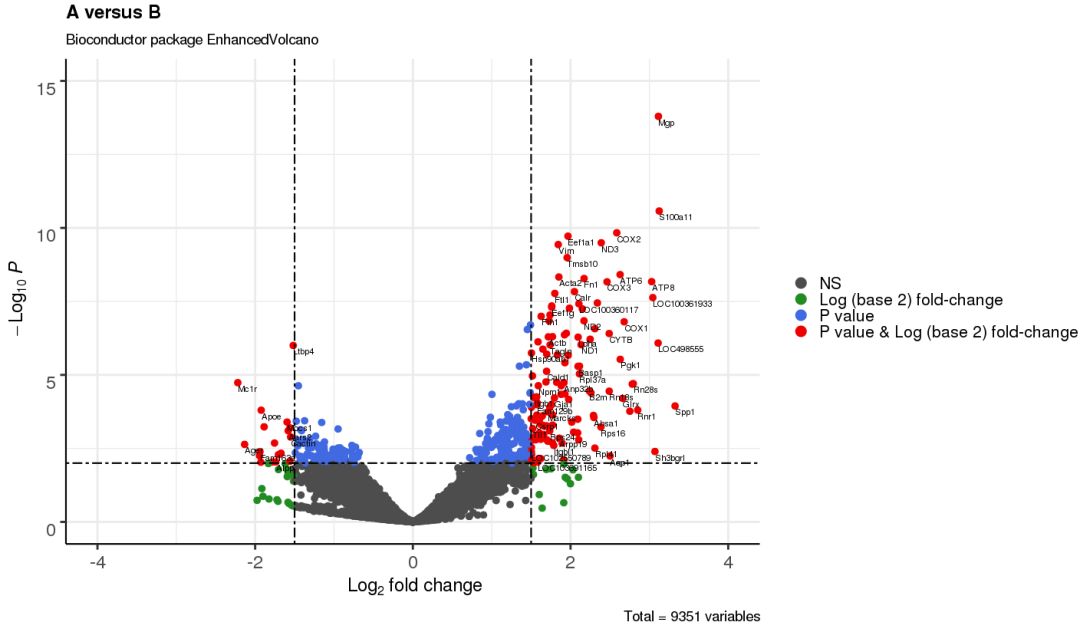
可以不展示图注
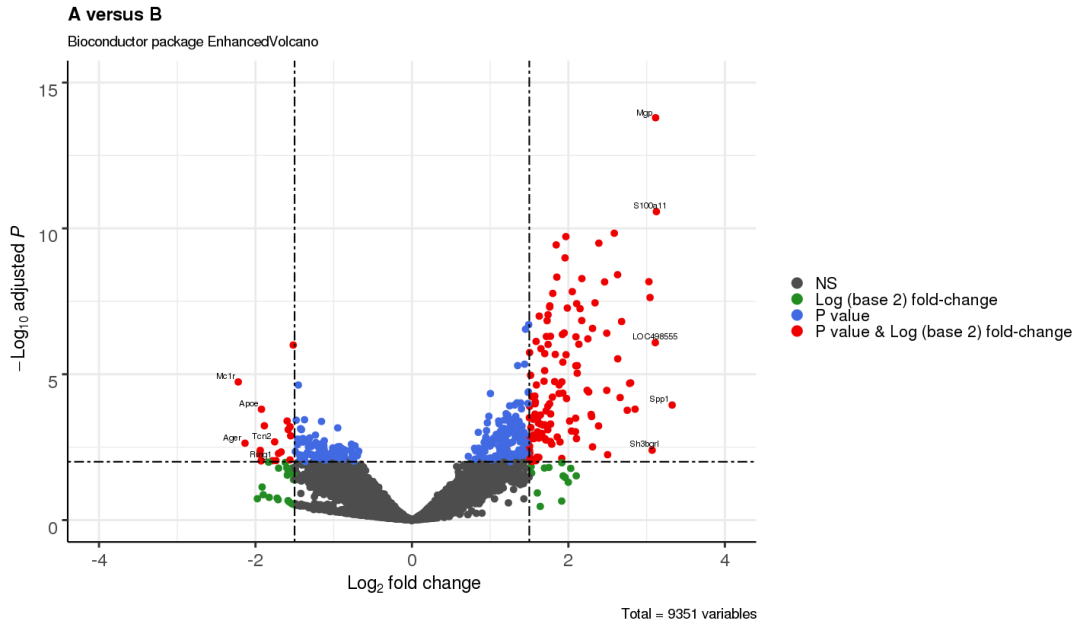
3.6 校正后的 p 值作图
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
xlab = bquote(~Log[2]~ 'fold change'),
ylab = bquote(~-Log[10]~adjusted~italic(P)),
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
legendLabSize = 16,
legendIconSize = 5.0)
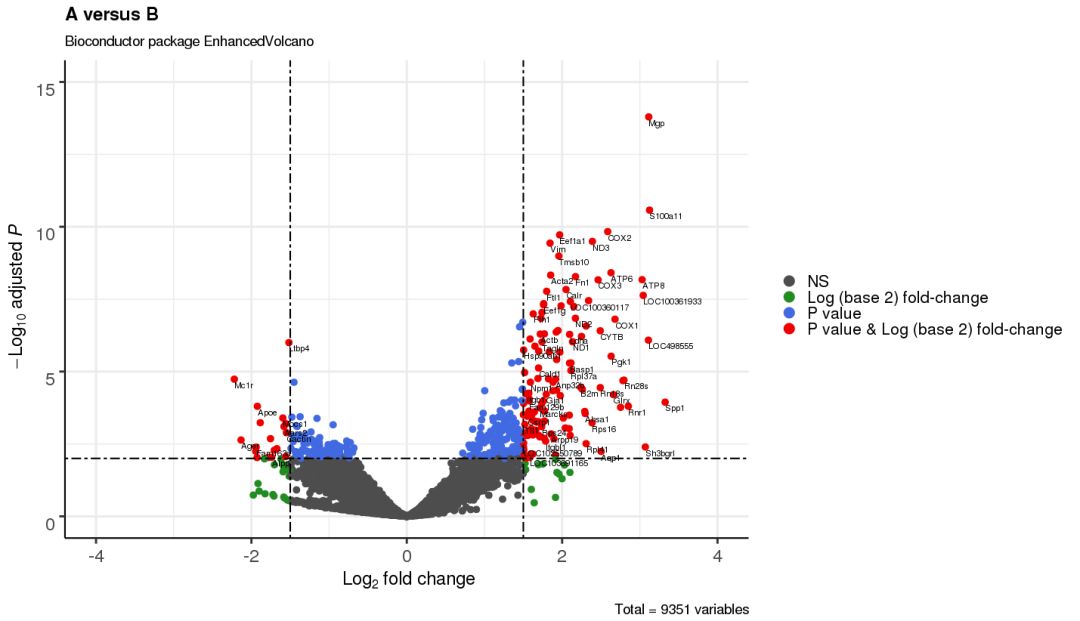
3.7 添加连线用于展示更多标注
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
xlab = bquote(~Log[2]~ 'fold change'),
ylab = bquote(~-Log[10]~adjusted~italic(P)),
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
drawConnectors = TRUE,
legendLabSize = 16,
legendIconSize = 5.0)
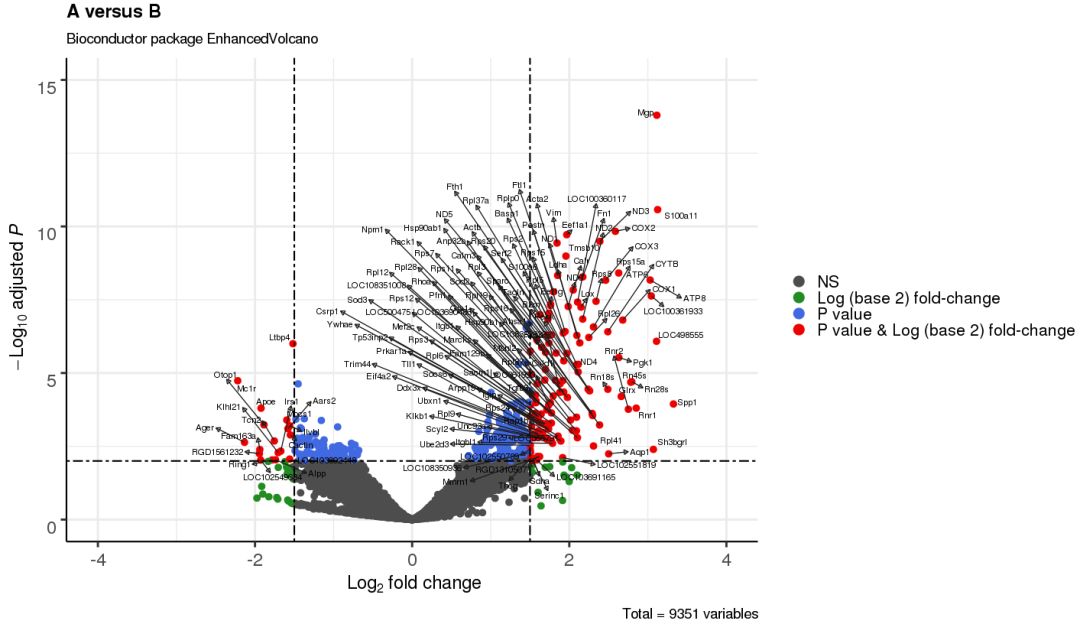
3.8 只标注重要变量
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
xlab = bquote(~Log[2]~ 'fold change'),
ylab = bquote(~-Log[10]~adjusted~italic(P)),
selectLab = c('Spp1','S100a11','Mgp','LOC498555','Sh3bgrl',
'Ring1','Apoe','Tcn2','Ager','Mc1r'),
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
drawConnectors = TRUE,
legendLabSize = 16,
legendIconSize = 5.0)
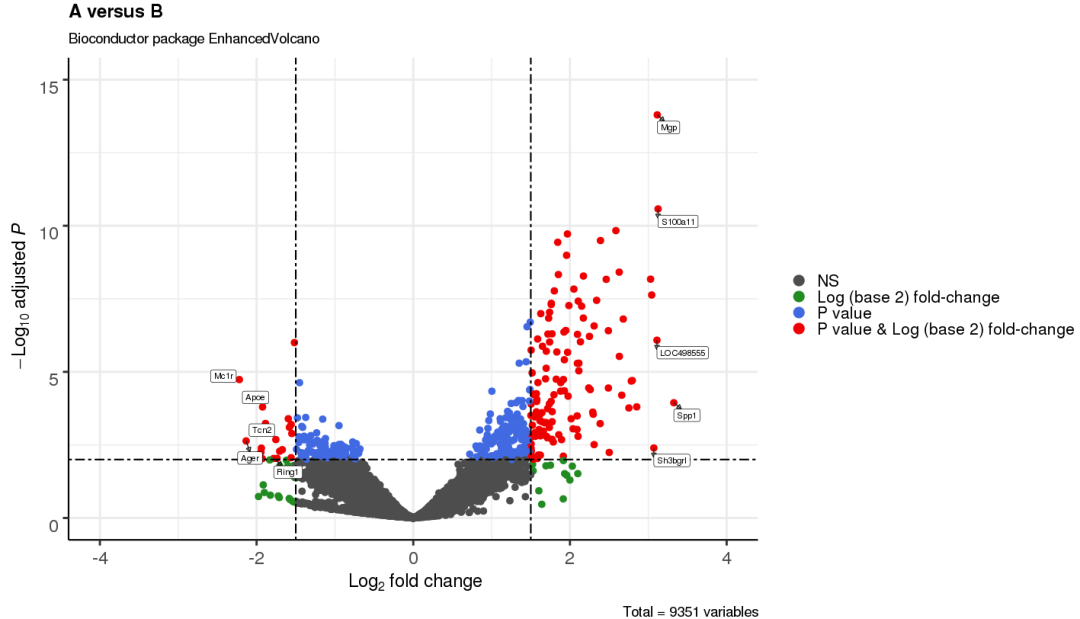
3.9 加框展示变量
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
xlab = bquote(~Log[2]~ 'fold change'),
ylab = bquote(~-Log[10]~adjusted~italic(P)),
selectLab = c('Spp1','S100a11','Mgp','LOC498555','Sh3bgrl',
'Ring1','Apoe','Tcn2','Ager','Mc1r'),
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
drawConnectors = TRUE,
boxedlabels = TRUE,
legendLabSize = 16,
legendIconSize = 5.0)
3.10 针对特殊点设置颜色
colCustom 功能可针对特定位点设置颜色,例如上下调基因设置不同颜色,参考代码如下:
keyvals <- rep('black', nrow(res))
# set the base name/label as 'Mid'
names(keyvals) <- rep('Mid', nrow(res))
# fold change > 1.5 & p-value < 0.0001 为高表达
keyvals[which(res$"log2(Fold_change)" > 1.5 & res$"p-value"<0.0001)] <- 'gold'
names(keyvals)[which(res$"log2(Fold_change)" > 1.5 & res$"p-value"<0.0001)] <- 'high'
# fold change < -1.5 & p-value < 0.0001为低表达
keyvals[which(res$"log2(Fold_change)" < -1.5 & res$"p-value"<0.0001)] <- 'royalblue'
names(keyvals)[which(res$"log2(Fold_change)" < -1.5 & res$"p-value"<0.0001)] <- 'low'
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = 'A versus B',
pCutoff = 10e-3,
FCcutoff = 1.5,
xlab = bquote(~Log[2]~ 'fold change'),
ylab = bquote(~-Log[10]~adjusted~italic(P)),
selectLab = rownames(res)[which(names(keyvals) %in% c('high', 'low'))],
transcriptPointSize = 3.0,
transcriptLabSize = 3.0,
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
colCustom = keyvals,
border = 'full',
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
drawConnectors = FALSE,
boxedlabels = FALSE,
legendLabSize = 16,
legendIconSize = 5.0)
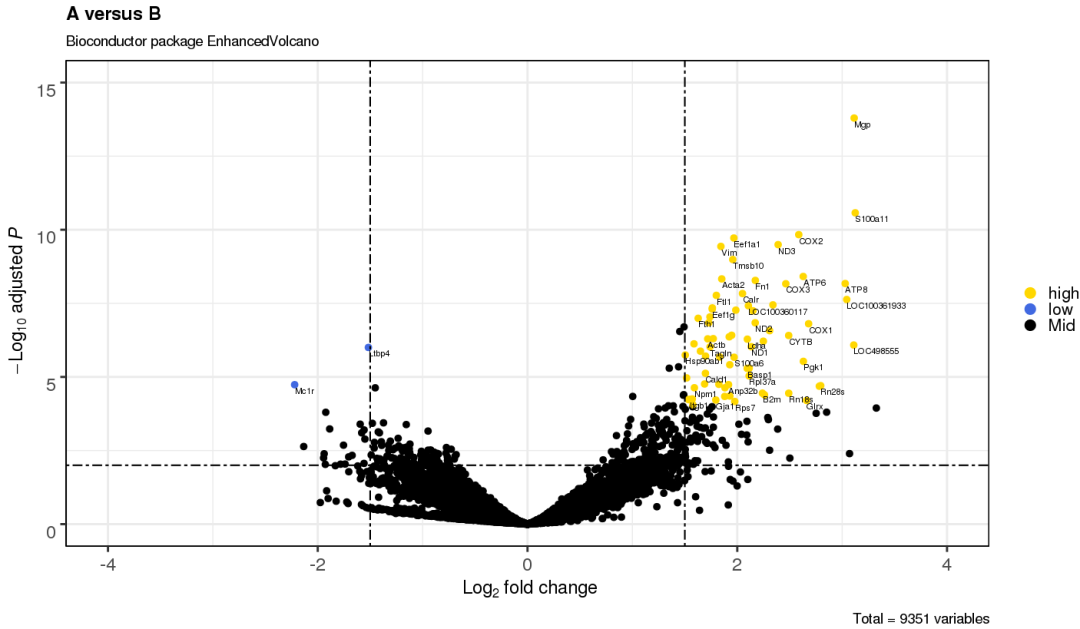
3.11 设置特定点的大小
p <- EnhancedVolcano(res,
lab = rownames(res),
x = 'log2(Fold_change)',
y = 'p-value',
xlim = c(-4, 4),
ylim = c(0,15),
title = '',
subtitle = '',
pCutoff = 10e-3,
FCcutoff = 1.5,
xlab = bquote(~Log[2]~ 'fold change'),
ylab = bquote(~-Log[10]~adjusted~italic(P)),
selectLab = rownames(res)[which(names(keyvals) %in% c('high', 'low'))],
transcriptLabSize = 3.0,
transcriptPointSize = c(ifelse((res$"log2(Fold_change)">2 |res$"log2(Fold_change)"< -2) & res$"p-value"<0.0001 , 3, 1)),
colAlpha = 1,
cutoffLineType = 'twodash',
cutoffLineWidth = 0.8,
colCustom = keyvals,
border = 'full',
legend=c('NS','Log (base 2) fold-change','P value',
'P value & Log (base 2) fold-change'),
legendPosition = 'right',
drawConnectors = FALSE,
boxedlabels = FALSE,
legendLabSize = 16,
legendIconSize = 5.0,
caption = "")
p
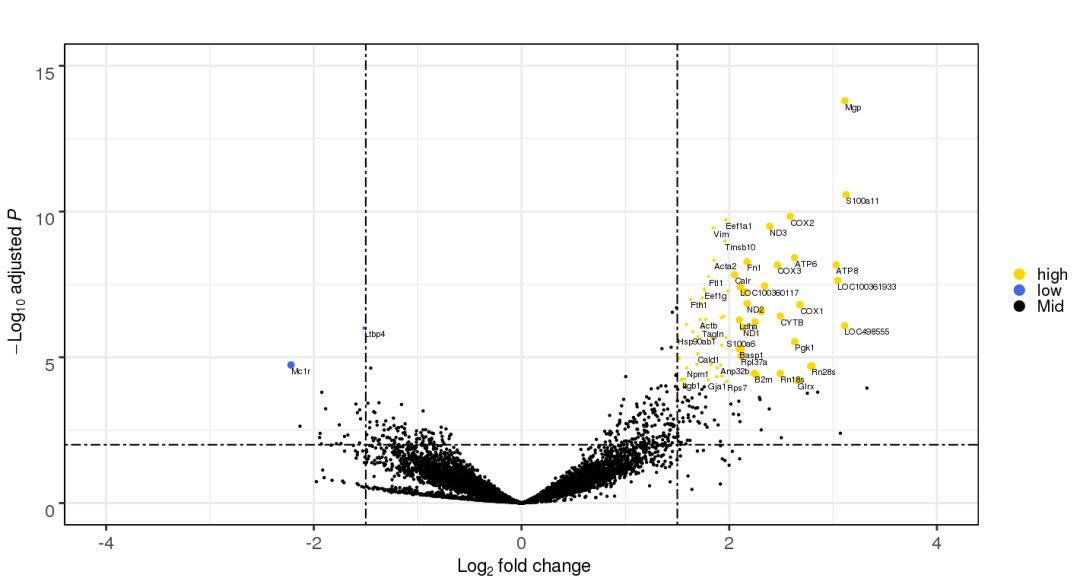
3.12 自定义刻度
p +
ggplot2::coord_cartesian(xlim=c(-6, 6)) +
ggplot2::scale_x_continuous(
breaks=seq(-6,6, 1))
EnhancedVolcano 包绘制火山图就先介绍到这里,如果对你有所帮助,请点个赞吧。

本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
R EnhancedVolcano 绘制火山图的更多相关文章
- R语言绘制花瓣图flower plot
R语言中有很多现成的R包,可以绘制venn图,但是最多支持5组,当组别数大于5时,venn图即使能够画出来,看上去也非常复杂,不够直观: 在实际的数据分析中,组别大于5的情况还是经常遇到的,这是就可以 ...
- R语言绘制QQ图
无论是直方图还是经验分布图,要从比较上鉴别样本是否处近似于某种类型的分布是困难的 QQ图可以帮我们鉴别样本的分布是否近似于某种类型的分布 R语言,代码如下: > qqnorm(w);qqline ...
- R语言绘制茎叶图
与直方图相比,茎叶图更能细致的看出数据分布情况! 代码: > x<-c(25, 45, 50, 54, 55, 61, 64, 68, 72, 75, 75,+ 78, 79, 81, 8 ...
- R语言——绘制半圆形图
好久没发点新的作品了.......也许...... Que sera, seraWhatever will be, will be
- R绘制韦恩图 | Venn图
解决方案有好几种: 网页版,无脑绘图,就是麻烦,没有写代码方便 极简版,gplots::venn 文艺版,venneuler,不好安装rJava,参见Y叔 酷炫版,VennDiagram 特别注意: ...
- GGPLOT2-plotly |让你的火山图“活”过来
火山图(Volcano Plot)常用于展示基因表达差异的分布,横坐标常为Fold change(倍数),越偏离中心差异倍数越大;纵坐标为P值(P值),值越大差异越显着.原因得名也许的英文因为查询查询 ...
- R绘图 第六篇:绘制线图(ggplot2)
线图是由折线构成的图形,线图是把散点从左向右用直线连接起来而构成的图形,在以时间序列为x轴的线图中,可以看到数据增长的趋势. geom_line(mapping = NULL, data = NULL ...
- R语言绘制相对性关系图
准备 第一步就是安装R语言环境以及RStudio 图绘制准备 首先安装库文件,敲入指令,回车 install.packages('corrplot') 然后安装excel导入的插件,点击右上角impo ...
- 一幅图解决R语言绘制图例的各种问题
一幅图解决R语言绘制图例的各种问题 用R语言画图的小伙伴们有木有这样的感受,"命令写的很完整,运行没有报错,可图例藏哪去了?""图画的很美,怎么总是图例不协调?" ...
- [R] 如何绘制各样本的pathway丰度热图?
前言 一般而言,我们做完pathway富集分析,就做下气泡图或bar图来进行展示,但它们实际上只考虑了富集因子和Pvalue.如果我们不关注这两个因素,而是在乎样本本身的pathway丰度呢? 对于K ...
随机推荐
- Xenomai 源码分析-Part I
Xenomai Edition v3.0.5 xenomai_init() static int __init xenomai_init(void) 源码分析 setup_init_state // ...
- 因为手哆嗦,发现了一个关于Python逗号的隐藏用法
python常规的用法,众多pythoner早已熟烂于心,如: 1.当一个元组只有一个元素时 a = (1, ) 2.当表示解包一个容器时 a = [('amo', 1), ('bmo', 1)] ...
- Linux报错:audit: backlog limit exceeded(审计:超出积压限制)
Linux报错:audit: backlog limit exceeded(审计:超出积压限制) 系统版本:CentOS Linux release 7.6.1810 (Core) 问题现象:一次巡检 ...
- CTF-RE-学习记录-汇编-2
汇编工具DTDebug 下载后设置好odd与插件路径,同时在属性中设置为管理员身份运行(无Administrator权限进入的时候一直在提示) 寄存器 存储数据: CPU>内存>硬盘 32 ...
- Spring(Bean详解)
GoF之工厂模式 GoF是指二十三种设计模式 GoF23种设计模式可分为三大类: 创建型(5个):解决对象创建问题. 单例模式 工厂方法模式 抽象工厂模式 建造者模式 原型模式 结构型(7个):一些类 ...
- 随机服务系统模拟—R实现(二)
M/M/1随机服务系统的模拟 M/M/1模型是一种出生-死亡过程,此随机过程中的每一个状态代表模型中人数的数目.因为模型的队列长度无限且参与人数亦无限,故此状态数目亦为无限.例如状态0表示模型闲置.状 ...
- 五月八号java基础知识点
1.对于容器中元素进行访问时,经常需要按照某种次序对容器中的每个元素访问且仅访问 一次,这就是遍历,也称为迭代.2.遍历是指从容器中获得当前元素的后续元素.对元素的遍历有很多种:第一种就是foreac ...
- 粘包,自定义协议,struct模块,粘包解决终极大招
粘包: 1.粘包问题出现的原因: (udp不会出现粘包问题) 1.1.tcp是流式协议,数据像水流一样黏在一起,没有任何边界区分 1.2.收数据没收干净,有残留,就会下一次结果混淆在一起去(客户端接受 ...
- 多态 polymorphic
多态是同一个行为具有多个不同表现形式,多态实现需要以下条件 继承父类并重写方法 父类引用指向子类对象:Animal a = new Cat(); 多态实例: //测试类 public class Te ...
- handler+looper+messagequeue源码解析
https://www.jianshu.com/p/b4d745c7ff7ahandler机制源码1.handler机制的作用在多线程的场景中,将子线程中需要更新UI的操作信息传递到UI主线程.多个线 ...
