项目笔记《DeepLung:Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification》(二)(上)模型设计
我只讲讲检测部分的模型,后面两样性分类的试验我没有做,这篇论文采用了很多肺结节检测论文都采用的u-net结构,准确地说是具有DPN结构的3D版本的u-net,直接上图。
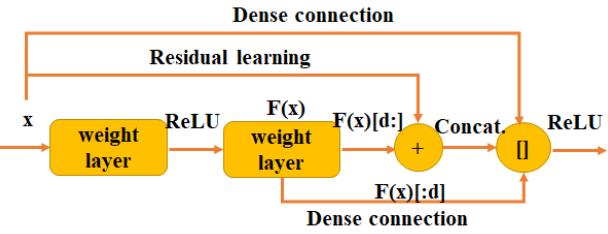
DPN是颜水成老师团队的成果,简单讲就是dense 与 residual的结合,如上图,输入特征图一部分通过residual与输出相加,另一部分与residual的结果再串联,个人觉得这个网络好乱,不简洁的网络都不是好网络,恰好文章中还给出了只采用residual的版本,所以我其实要讲的是这个只有residual的u-net,上图。
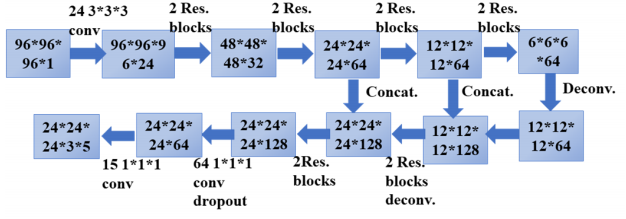
可以看到,输入是96*96*96的立方体,里面包含标记的结节,经过24个3*3*3的卷积核,通道数变为24,然后经过4个stage,尺寸缩减为1/16,接下来是分辨率放大阶段,采用反卷积实现,连续两个阶段都是反卷积后与低层特征串联,然后经过两个卷积操作,通道数变为15,图示中以3*5显示,是为了更清楚地表明,最后输出的proposal中,每个位置有三个,分别采用三种尺寸,设置的三个anchor尺寸是[5,10,20],每个位置预测z,y,x,d,p分别是结节的三维坐标以及直径,置信度。
下面看一下源码,采用pytorch框架。
首先是residual block的设计,位于layers.py文件
class PostRes(nn.Module):
def __init__(self, n_in, n_out, stride = 1):
super(PostRes, self).__init__()
self.conv1 = nn.Conv3d(n_in, n_out, kernel_size = 3, stride = stride, padding = 1)
self.bn1 = nn.BatchNorm3d(n_out)
self.relu = nn.ReLU(inplace = True)
self.conv2 = nn.Conv3d(n_out, n_out, kernel_size = 3, padding = 1)
self.bn2 = nn.BatchNorm3d(n_out) if stride != 1 or n_out != n_in:
self.shortcut = nn.Sequential(
nn.Conv3d(n_in, n_out, kernel_size = 1, stride = stride),
nn.BatchNorm3d(n_out))
else:
self.shortcut = None def forward(self, x):
residual = x
if self.shortcut is not None:
residual = self.shortcut(x)
out = self.conv1(x)
out = self.bn1(out)
out = self.relu(out)
out = self.conv2(out)
out = self.bn2(out) out += residual
out = self.relu(out)
return out
可以看到采用结构与2D的residual基本一致,采用的都是conv-bn-relu,根据步长和输入输出的尺寸,采用identity或1*1卷积作为skip connection。
然后就是网络,位于res18.py文件
class Net(nn.Module):
def __init__(self):
super(Net, self).__init__()
# The first few layers consumes the most memory, so use simple convolution to save memory.
# Call these layers preBlock, i.e., before the residual blocks of later layers.
self.preBlock = nn.Sequential(
nn.Conv3d(1, 24, kernel_size = 3, padding = 1),
nn.BatchNorm3d(24),
nn.ReLU(inplace = True),
nn.Conv3d(24, 24, kernel_size = 3, padding = 1),
nn.BatchNorm3d(24),
nn.ReLU(inplace = True)) # 3 poolings, each pooling downsamples the feature map by a factor 2.
# 3 groups of blocks. The first block of each group has one pooling.
num_blocks_forw = [2,2,3,3]
num_blocks_back = [3,3] self.featureNum_forw = [24,32,64,64,64]
self.featureNum_back = [128,64,64]
for i in range(len(num_blocks_forw)):
blocks = []
for j in range(num_blocks_forw[i]):
if j == 0:
blocks.append(PostRes(self.featureNum_forw[i], self.featureNum_forw[i+1]))
else:
blocks.append(PostRes(self.featureNum_forw[i+1], self.featureNum_forw[i+1]))
setattr(self, 'forw' + str(i + 1), nn.Sequential(*blocks)) for i in range(len(num_blocks_back)):
blocks = []
for j in range(num_blocks_back[i]):
if j == 0:
if i==0:
addition = 3
else:
addition = 0
blocks.append(PostRes(self.featureNum_back[i+1]+self.featureNum_forw[i+2]+addition, self.featureNum_back[i]))
else:
blocks.append(PostRes(self.featureNum_back[i], self.featureNum_back[i]))
setattr(self, 'back' + str(i + 2), nn.Sequential(*blocks)) self.maxpool1 = nn.MaxPool3d(kernel_size=2,stride=2,return_indices =True)
self.maxpool2 = nn.MaxPool3d(kernel_size=2,stride=2,return_indices =True)
self.maxpool3 = nn.MaxPool3d(kernel_size=2,stride=2,return_indices =True)
self.maxpool4 = nn.MaxPool3d(kernel_size=2,stride=2,return_indices =True)
self.unmaxpool1 = nn.MaxUnpool3d(kernel_size=2,stride=2)
self.unmaxpool2 = nn.MaxUnpool3d(kernel_size=2,stride=2) self.path1 = nn.Sequential(
nn.ConvTranspose3d(64, 64, kernel_size = 2, stride = 2),
nn.BatchNorm3d(64),
nn.ReLU(inplace = True))
self.path2 = nn.Sequential(
nn.ConvTranspose3d(64, 64, kernel_size = 2, stride = 2),
nn.BatchNorm3d(64*k),
nn.ReLU(inplace = True))
self.drop = nn.Dropout3d(p = 0.5, inplace = False)
self.output = nn.Sequential(nn.Conv3d(self.featureNum_back[0], 64, kernel_size = 1),
nn.ReLU(),
#nn.Dropout3d(p = 0.3),
nn.Conv3d(64, 5 * len(config['anchors']), kernel_size = 1)) def forward(self, x, coord):
out = self.preBlock(x)#
out_pool,indices0 = self.maxpool1(out)
out1 = self.forw1(out_pool)#
out1_pool,indices1 = self.maxpool2(out1)
out2 = self.forw2(out1_pool)#
#out2 = self.drop(out2)
out2_pool,indices2 = self.maxpool3(out2)
out3 = self.forw3(out2_pool)#
out3_pool,indices3 = self.maxpool4(out3)
out4 = self.forw4(out3_pool)#
#out4 = self.drop(out4) rev3 = self.path1(out4)
comb3 = self.back3(torch.cat((rev3, out3), 1))#64+64
#comb3 = self.drop(comb3)
rev2 = self.path2(comb3) comb2 = self.back2(torch.cat((rev2, out2,coord), 1))#
comb2 = self.drop(comb2)
out = self.output(comb2)
size = out.size()
out = out.view(out.size(0), out.size(1), -1)
#out = out.transpose(1, 4).transpose(1, 2).transpose(2, 3).contiguous()
out = out.transpose(1, 2).contiguous().view(size[0], size[2], size[3], size[4], len(config['anchors']), 5)
#out = out.view(-1, 5)
return out
看代码的时候有个地方比较绕,就是forw模块和back模块的迭代实现,个人觉得还不如直接一个模块一个模块地写出来,虽然多点代码,但比较清晰。还有就是path模块,其实就是反卷积模块。
网络结构就是这些,其实难点在loss的定义,以及标签的映射,下面来看一下loss的定义,标签映射以及数据增强部分待到(中)(下)部再讲。
loss的定义采用的也是pytorch网络的定义,位于layers.py文件。
上代码。
class Loss(nn.Module):
def __init__(self, num_hard = 0):
super(Loss, self).__init__()
self.sigmoid = nn.Sigmoid()
self.classify_loss = nn.BCELoss() #二分类交叉熵损失
self.regress_loss = nn.SmoothL1Loss() #平滑L1损失
self.num_hard = num_hard #hardming 数目 def forward(self, output, labels, train = True):
batch_size = labels.size(0) #标签的第0维度,样本数
output = output.view(-1, 5) #将输出维度调整,以anchor为第二维度
labels = labels.view(-1, 5) #将标签维度对应调整,同上 pos_idcs = labels[:, 0] > 0.5 #对标签进行筛选,输出为索引,示例[1,2,5]
pos_idcs = pos_idcs.unsqueeze(1).expand(pos_idcs.size(0), 5) #对索引维度扩展,重复5次,示例[[1,1,1,1,1],[2,2,2,2,2],[5,5,5,5,5]]
pos_output = output[pos_idcs].view(-1, 5) #筛选出与正标签对应的输出
pos_labels = labels[pos_idcs].view(-1, 5) #筛选出正标签 neg_idcs = labels[:, 0] < -0.5 #同上,筛选负标签索引,此处为负值
neg_output = output[:, 0][neg_idcs] #注意,此处与上面不同,负标签只考虑置信度即可,因为位置及直径不计入损失,没有意义
neg_labels = labels[:, 0][neg_idcs] if self.num_hard > 0 and train:#判断是否定义了,hardmining
neg_output, neg_labels = hard_mining(neg_output, neg_labels, self.num_hard * batch_size) #只选择置信度较高的负样本作计算,对于易于分类的负样本,都是虾兵蟹将,不足虑
neg_prob = self.sigmoid(neg_output)#对负样本输出进行sigmoid处理,生成0~1之间的值,符合置信度的范围,可能大家要问输出不就是0~1吗,这里网络最后没有用sigmoid激活函数,所以最后输出应该是没有范围的,
#这里我也比较不解,直接在网络中加入sigmoid不就行了
#classify_loss = self.classify_loss(
# torch.cat((pos_prob, neg_prob), 0),
# torch.cat((pos_labels[:, 0], neg_labels + 1), 0))
if len(pos_output)>0:
pos_prob = self.sigmoid(pos_output[:, 0]) #对正样本进行sigmoid处理
pz, ph, pw, pd = pos_output[:, 1], pos_output[:, 2], pos_output[:, 3], pos_output[:, 4] #依次输出z,h,w,d以便与标签结合求损失
lz, lh, lw, ld = pos_labels[:, 1], pos_labels[:, 2], pos_labels[:, 3], pos_labels[:, 4] #依次输出z,h,w,d以便与输出结合求损失 regress_losses = [ #回归损失
self.regress_loss(pz, lz),
self.regress_loss(ph, lh),
self.regress_loss(pw, lw),
self.regress_loss(pd, ld)]
regress_losses_data = [l.data[0] for l in regress_losses]
classify_loss = 0.5 * self.classify_loss( #对正样本和负样本分别求分类损失
pos_prob, pos_labels[:, 0]) + 0.5 * self.classify_loss(
neg_prob, neg_labels + 1)
pos_correct = (pos_prob.data >= 0.5).sum() #那些输出确实大于0.5的正样本是正确预测的正样本
pos_total = len(pos_prob) #正样本总数 else: #如果没有正标签,由于负标签又不用计算回归损失,于是回归损失就置零了,分类损失只计算负标签的分类损失
regress_losses = [0,0,0,0]
classify_loss = 0.5 * self.classify_loss(
neg_prob, neg_labels + 1)
pos_correct = 0 #此时没有正样本或正标签
pos_total = 0 #总数也为0
regress_losses_data = [0,0,0,0]
classify_loss_data = classify_loss.data[0] #loss = classify_loss#pytorch 0.4
loss = classify_loss.clone()
for regress_loss in regress_losses: #将回归损失与分类损失相加,求出总损失(标量)
loss += regress_loss neg_correct = (neg_prob.data < 0.5).sum() #那些输出确实低于0.5的负样本是正确预测的负样本
neg_total = len(neg_prob) #负样本总数 return [loss, classify_loss_data] + regress_losses_data + [pos_correct, pos_total, neg_correct, neg_total]
对于损失的解释都在代码旁边的注释了,只是有一点不大明白,求负样本损失的时候为何要把置信度加1?,应该是负标签在打标签的时候置为-1了,由此又想到一个问题,那些既非正也非负的样本的置信度是如何设置的,应该不是随机设置的,难道设为0了?
在(中)里面,我想把标签映射以及数据增强,讲一下,奈何自己还不太懂,等等吧,如果(中)完成,在(下)里简单说一说训练以及验证,以及测试,这些都完成,那么deeplung笔记三部曲连在一起就完整了。
项目笔记《DeepLung:Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification》(二)(上)模型设计的更多相关文章
- 项目笔记《DeepLung:Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification》(三)(下)结果评估
在(上)中讲了如何得到csv文件并调用noduleCADEvaluationLUNA16.py求取froc值,这里就讲一讲froc值是如何求取的. annotations_filename = './ ...
- 项目笔记《DeepLung:Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification》(三)(上)结果评估
在(一)中,我将肺结节检测项目总结为三阶段,这里我要讲讲这个项目的第三阶段,至于第二阶段,由于数据增强部分的代码我始终看不大懂,先不讲. 结果评估的程序在evaluationScript文件夹下,这个 ...
- 项目笔记《DeepLung:Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification》(一)预处理
最近一个月都在做肺结节的检测,学到了不少东西,运行的项目主要是基于这篇论文,在github上可以查到项目代码. 我个人总结的肺结节检测可以分为三个阶段,数据预处理,网络搭建及训练,结果评估. 这篇博客 ...
- Django商城项目笔记No.2项目准备工作
Django商城项目笔记No.2项目准备工作 接着上篇开始,创建好工程之后,随之而来的是怎么配置工程,这篇文章记录如何进行相关的配置 1.pycharm打开工程,进行相关的配置 通过pycharm打开 ...
- Django项目笔记:sessions处理以及复杂对象序列化
前言:一点题外话 我发现,不更新博客的时候,不是非常忙,就是效率非常低.最近没怎么更新博客,原因是第二种= =.惭愧惭愧. 今天效率出奇的高,一天时间把PassNote后端的接口全部写完了,Djang ...
- Dual Path Networks(DPN)——一种结合了ResNet和DenseNet优势的新型卷积网络结构。深度残差网络通过残差旁支通路再利用特征,但残差通道不善于探索新特征。密集连接网络通过密集连接通路探索新特征,但有高冗余度。
如何评价Dual Path Networks(DPN)? 论文链接:https://arxiv.org/pdf/1707.01629v1.pdf在ImagNet-1k数据集上,浅DPN超过了最好的Re ...
- 《BI项目笔记》——微软BI项目笔记连载
本系列文章主要是结合实际项目,加上自己的总结,整理出来的一系列项目笔记,涉及微软SQL Server2008中商务智能开发中的SSAS.SSIS模块: 准备工作: <BI项目笔记>基于雪 ...
- A Statistical View of Deep Learning (IV): Recurrent Nets and Dynamical Systems
A Statistical View of Deep Learning (IV): Recurrent Nets and Dynamical Systems Recurrent neural netw ...
- 项目笔记---CSharp图片处理
原文:项目笔记---CSharp图片处理 项目笔记---CSharp图片处理 最近由于项目上需要对图片进行二值化处理,就学习了相关的图片处理上的知识,从开始的二值化的意义到动态阀值检测二值化等等,并用 ...
随机推荐
- SqlServer——游标
参考:http://www.cnblogs.com/94cool/archive/2010/04/20/1715951.html http://www.cnblogs.com/moss_tan_ju ...
- tomcat中间件配置说明
因为Tomcat 技术先进.性能稳定,而且免费,因而深受Java 爱好者的喜爱并得到了部分软件开发商的认可,成为目前比较流行的Web 应用服务器.目前最新版本是8.0. 方法/步骤 一.tomca ...
- DAY10-MYSQL存储引擎
一 什么是存储引擎 mysql中建立的库===>文件夹 库中建立的表===>文件 现实生活中我们用来存储数据的文件有不同的类型,每种文件类型对应各自不同的处理机制:比如处理文本用txt类型 ...
- eclipse中maven的配置与使用
以eclipse Juno版本为例 1.插件安装 eclipse==>help====>install new software===>add name :m2e location: ...
- 定位程序问题出现的原因工具-jstack
jstack还可以生成线程快照 如何使用jstack: 1.打开命令行,输入jstack 在任务管理器中就可看到对应进程id 2.在命令行中输入 jstack -l 进程id 这样就可得到进程中所有的 ...
- 在PHP中PDO解决中文乱码问题的一些补充
我的环境是appsver包, 在网上最常出现的解决中文乱码显示的代码是: 第一种:PDO::__construct($dsn, $user, $pass, array (PDO::MYSQL_ATTR ...
- session和cookie个字消除的方法
session消除的方法就是: session_destroy(); cookie消除的方法就是setcookie()函数的时间设为当前时间即可 if(isset($_COOKIE['adminId' ...
- Angular18 RXJS
1 RX 全称是 Reactive Extensions,它是微软开发并维护的基于 Reactive Programming 范式实现的一套工具库集合:RX结合了观察者模式.迭代器模式.函数式编程来管 ...
- 02 mybatis环境搭建 【spring + mybatis】
1 导包 <project xmlns="http://maven.apache.org/POM/4.0.0" xmlns:xsi="http://www.w3.o ...
- 中华人民共和国建筑工业行业标准—IFC详细解读 第五篇
