NCBI News
NCBI淘汰序列GI - 使用Accession.Version代替!
截至2016年9月,被称为“GI”的整数序列标识符将不再包括在NCBI支持的序列记录的GenBank,GenPept和FASTA格式中。FASTA标题将进一步简化,以便仅报告国际序列数据库协作(INSDC)和NCBI参考序列(RefSeq)项目管理的登录的序列登录号和记录标题。 当NCBI进行此转换时,我们鼓励具有依赖于GI的工作流的任何用户开始计划使用accession.version标识符。2016年9月之后,任何完全依赖于地理标志的过程将不再像预期的那样发挥作用。
GI号自GenBank版本81.0(1994年2月)以来一直在使用,作为登录号的附加标识符,以稳定地参考序列记录的特定版本。版本跟踪在1997年作为整数后缀添加到登记号中,该后缀随着对记录中的序列数据的每次更新而增加。例如,“ AC020606.7 ”表示记录的序列内容自第一次发布以来已更新六次。因此,已经以冗余方式通过GI和登录版本提供序列版本化信息。在过去十年中,NCBI继续以快速增长的速度接收新的或更新的序列的提交。为了回应这一点,我们不得不开发使用accession.version信息的新数据存储解决方案,而不是GI信息,以跟踪更新。缺乏GI的序列的当前实例包括WGS和TSA项目中的未注释重叠群。这导致我们传送版本信息不一致的情况。
考虑到数据提交数量的持续增加和记录提交的日益不一致,现在是我们采取下一步,删除旧的冗余GI标识符并保留序列版本的单一标识符的时候了,可读的accession.version。这种改变将简化跟踪序列的过程,而没有任何功能的损失。这种变化还将通过促进使用accession.version作为优选序列标识符来简化科学通信。因此,在接下来的几个月中,我们将不再将GI分配给越来越多的新序列。具有现有GI的序列记录将以某些表示格式保留它们,例如ASN.1和5列特征表格格式,但GI值将不再以其他表示格式显示,包括GenBank平面文件和FASTA格式。将继续支持接受GI作为输入的NCBI服务,并且NCBI将向当前不支持它们的所有服务添加对accession.version标识符的支持。
这种转换到停止分配和报告GI首先在2013年12月的GenBank 199.0版本说明中描述,并在最近的GenBank更新中描述。有关背景信息,请参见当前GenBank发行说明的第1.4.1节:ftp : //ftp.ncbi.nih.gov/genbank/gbrel.txt
由INSDC和所有NCBI RefSeq记录交换的所有序列记录的FASTA显示也将更改为仅报告accession.version和记录标题。这将提高与NCBI提供的其他文件类型(包括GFF3,Gene和dbSNP下载文件)的兼容性。基于用户请求,已经对来自重新设计的基因组FTP位点的数据进行了该FASTA格式改变,以具有GFF3和FASTA格式的单一一致的序列标识符。请参阅此更改的以前公告:http://www.ncbi.nlm.nih.gov/news/08-26-2014-new-genomes-FTP-live/ ..此时,我们计划继续提供来自非INSDC和非RefSeq源的序列的FASTA显示中的数据库源信息,包括:SwissProt,PDB结构,PIR和专利序列。
2016年9月之后,这些更改将开始出现在平面文件和FASTA格式序列数据,NCBI编程实用程序结果以及GenBank和RefSeq全面FTP版本的NCBI Web视图中。
实施例1:INSDC核苷酸记录 - 在下面的样品记录中,核苷酸序列AF123456被分配GI为6633795,并且从其编码区特征翻译的蛋白质被分配GI为6633796:
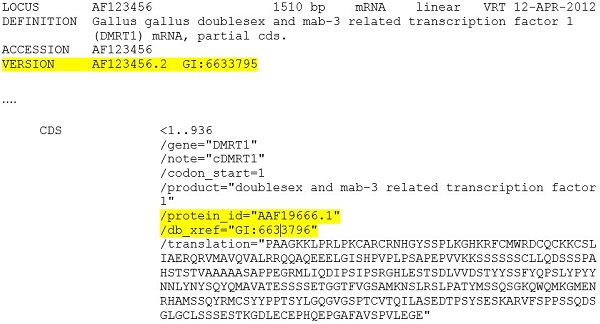
2016年9月之后,accession.version将是序列版本的唯一指示符。VERSION行上的GI值和编码区特征的GI / db_xref限定符将不再可见。
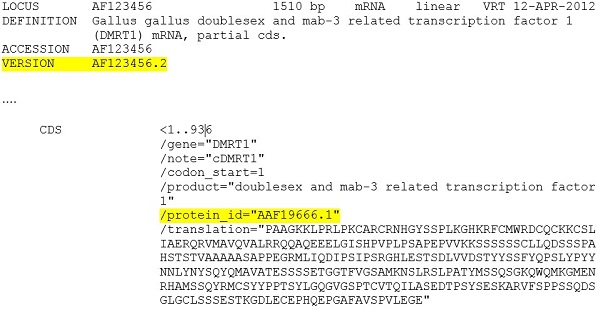
实施例2:GenPept蛋白质记录 - 当前记录显示包括VERSION行中的GI。注意,GenPept格式的编码区特征从未包括GI值的显示。

2016年9月后,VERSION行将不包含GI值:
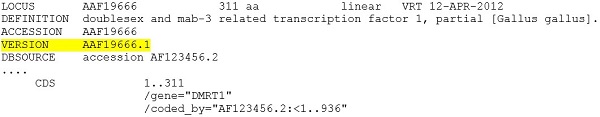
示例3:对FASTA格式的更改:GI和数据库源值将从FASTA头中删除 - 在大多数资源中,当前的FASTA显示当前包括GI和数据库源信息(例如,GenBank的'gb'), '。下游分析工具通常需要首先处理FASTA标题行以将序列标识符部分简化为登录版本或GI。复杂的FASTA序列标识符以黄色突出显示:

2016年9月之后,将在FASTA标题中提供一个简单的序列ID用于核苷酸和蛋白质记录

NCBI News的更多相关文章
- download ncRNA sequences form NCBI
#!/bin/bash usage() { echo;echo "Usage: ./`basename $0` [gi number list] [number of cpu]"; ...
- NCBI database download
ascp -T -l 200M -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh --host=ftp-private.ncbi.nlm.nih.go ...
- 生物信息大数据&数据库(NCBI、EBI、UCSC、TCGA)
想系统的学习生信数据库可以先看一下北大的公开课,有一章专门讲的数据库与软件: -生物信息学:导论与方法 北大\ 生物信息数据库及软件资源 一个优秀的生信开发者能够解决如下问题: 如何鉴定一个重要的且没 ...
- NCBI原始数据下载by Aspera Connect
主要参考这篇文章: http://mp.weixin.qq.com/s?__biz=MzA5NjU5NjQ4MA==&mid=2651154488&idx=1&sn=e693a ...
- NCBI下载sra数据(新)
今天要上NCBI下载sra数据发现没有下载的链接,网上查发现都是老的方法,NCBI页面已经变更,于是看了NCBI的help,并且记录下来新版的sra数据下载方法,要用NCBI的工具SRA Tool ...
- NCBI通过氨基酸位置查看相邻SNP
进入NCBI网站 在SNP的搜索框中输入SNP位点,比如“rs52811957” 在弹出的对话框中选择“Gene View” 进入以后会显示该变异相邻SNP.原始氨基酸.变异后的氨基酸.positio ...
- 【只要有ENA千万别用NCBI】拆分SRA文件,通过SRAtoolkits
只要有ENA千万别用NCBI!!!! 最近开始分析网上Download的数据,一开始用人家现成的GWAS数据,后来觉得反正自己的数据到手该做的也是要做的,出来混早晚是要还的,所以就开始从头分析一些SR ...
- 从 NCBI 批量下载基因组的方法
先下载 assembly summary files The assembly_summary files report metadata for the genome assemblies on t ...
- NCBI上查看SNP位点在哪个基因座上(locus)
首先,进入NCBI的主页网站:https://www.ncbi.nlm.nih.gov/variation/view/ 进入后,在下图红色框框位置输入目的SNP,比如rs608139 输完后,出现如下 ...
- 上传RNA-seq数据到NCBI GEO数据库
SRA - NCBI example - NCBI 要发文章了,审稿时编辑肯定会要求你上传NGS测序数据. 一般数据都是放在集群,不可能放在个人电脑上,因为有的数据大的吓人(几个T). 所以我们就建一 ...
随机推荐
- 互联网进行限流策略的Semaphore信号量使用
在Semaphore信号量非常适合高并发访问,新系统在上线之前,要对系统的访问量进行评估,当然这个值肯定不是随便拍拍脑袋就能想出来的,是经过以往的经验.数据.历年的访问量,已经推广力度进行一个合理的评 ...
- angular小技巧随笔
1. 重新刷新页面 同页面切换状态: $state.go('tab.index', {inviteId:inviteId}); self.location.reload();
- python:django
====启动django==== python manager.py runserver --host 0.0.0.0 --port 9008 python manager.py runserver ...
- f5基本介绍
1.信息查看 1)登录: https://10.160.100.10 f5有2台,做HA IP地址分别为10.160.100.3和10.160.100.2 10.160.100.10为虚拟地址 2)基 ...
- bbs项目实现点赞和评论的功能
一.点赞功能 思路是这样的: 1.对点赞和踩都设置一个相同的class,然后对这个class绑定点击事件 2.点击触发ajax请求,我们对赞的标签设置了一个class属性,对踩的标签没有设置这个cla ...
- Vmware黑屏解决方法
此原因是显卡性能差,显示选项开启了3D加速导致的,具体修改步骤: 英文路径:VM->Settings->Hardware->Display 在右面的内容栏中将 Accelerate ...
- 迭代器、生成器 day13
一 迭代器 迭代器的由来pythone2.2引进的,是一种序列(也是一种数据类型),也是为类对象提供一个序列的入口. for 循环str list tuple dict set 文件句柄可迭代: s ...
- windows上安装RabbitMQ
windows下 安装 rabbitMQ 及操作常用命令 rabbitMQ是一个在AMQP协议标准基础上完整的,可服用的企业消息系统.它遵循Mozilla Public License开源协议,采用 ...
- widget jquery 理解
jquery ui 的所有组件都是基于一个简单,可重用的widget. 这个widget是jquery ui的核心部分,实用它能实现一致的API,创建有状态的插件,而无需关心插件的内部转换. $.wi ...
- BZOJ1925或洛谷2467 [SDOI2010]地精部落
BZOJ原题链接 洛谷原题链接 先讲下关于波动数列的\(3\)个性质. 性质\(1\):对于数列中的每一对\(i\)和\(i + 1\),若它们不相邻,那么交换这两个数形成的依旧是一个波动数列. 性质 ...
