R 包 pathview 代谢通路可视化
pathview R 包是一个集成 pathway 通路数据与可视化的工具集。它用于把用户的数据映射并渲染到相关的 pathway 通路图上,用户只需要提供基因或者化合物数据(gene or compound data)并指定目标通路(specify the target pathway)即可。
pathview 会产生 native KEGG view 和 Graphviz view 两种 pathway 查看方式,前者以 native KEGG graph (.png) 进行渲染,后者则使用 graphviz layout engine (.pdf)。pathview 作为主程序提供了 downloader, parser, mapper 以及 viewer 四部分功能:自动下载通路图表数据,解析并映射用户数据,最后把 mapped 的数据渲染到通路图上。
Pathview automatically downloads the pathway graph data, parses the data file, maps user data to the pathway, and renders pathway graph with the mapped data.
pathview 安装
在 R 命令行下 pathview 安装:
# pathview 依赖包
> source( "http://bioconductor.org/biocLite.R" )
> biocLite(c("Rgraphviz", "png", "KEGGgraph", "org.Hs.eg.db"))
# pathview 安装
> biocLite("pathview")
我们也可以通过 R-forge 的方式安装:
> install.packages("pathview", repos="http://R-Forge.R-project.org")
或者通过下载 pathview 的源码包进行安装,这里不介绍。
pathview 使用
利用 pathview 自带的 example 数据(data(package="pathview" 可查看 pathview 包所有的 example 数据)绘制人 hsa04110 通路图:
> library(pathview)
> data(gse16873.d)
> pv.out <- pathview(gene.data = gse16873.d[, 1], pathway.id = "04110", species = "hsa", out.suffix = "gse16873")
当前目录得到 hsa04110.gse16873.png 通路图:
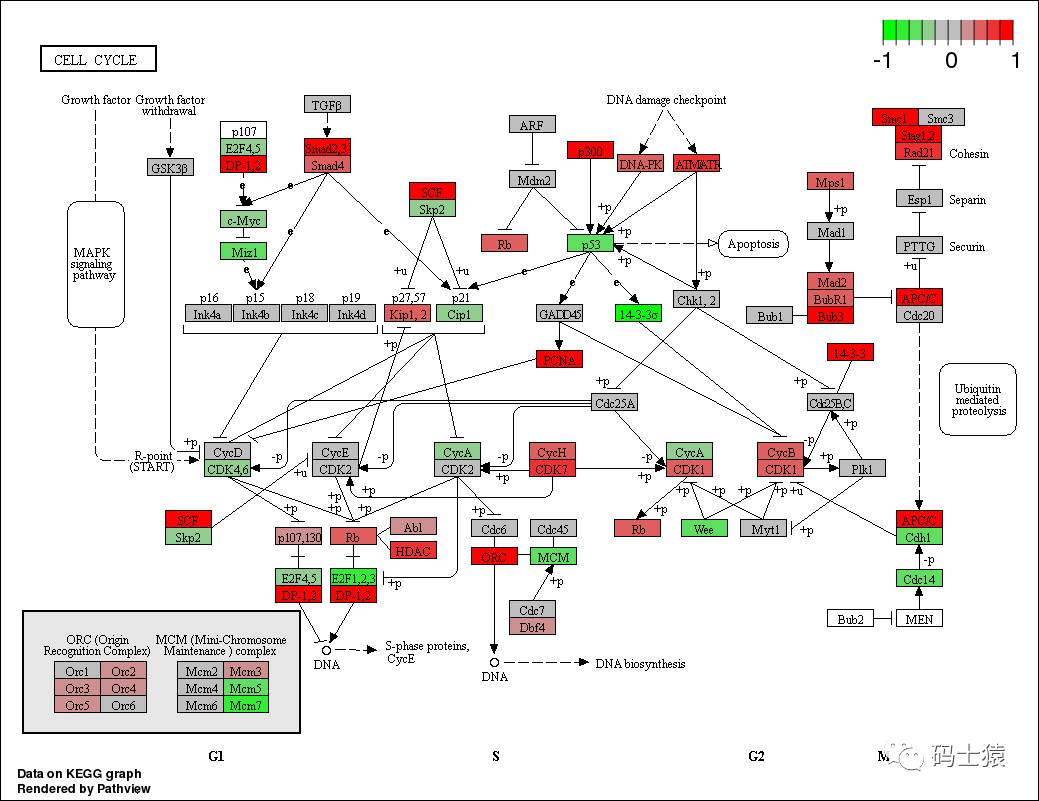
其中 gene.data 接收的是矩阵(或向量)的基因数据,这些数据既可以是数值型(like log2 fold change or absolute expression levels)也可以是基因 id 数据(默认为 entrez 的 gene id,gene.idtype = "entrez"),取决于我们想要得到什么样的可视化结果。
使用 gene IDs 的数据,得到的 hsa04110.geneid.png 如下:
> pv.out <- pathview(gene.data = c("1029"), pathway.id = "04110", species = "hsa", out.suffix = "geneid")
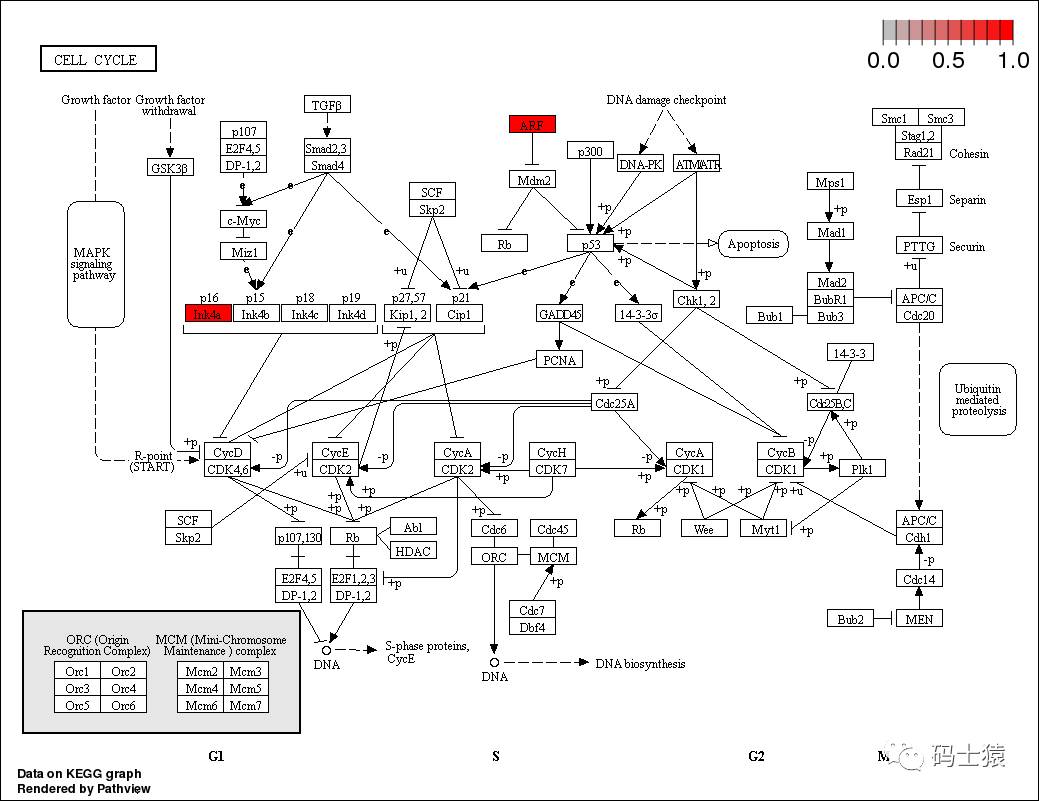
pathview 是一款功能强大的工具集,除了可以展示规范信号通路图外,还支持代谢通路图。利用 pathview 的化合物、基因内置数据,绘制代谢通路图如下:
> data(demo.paths)
> sim.cpd.data = sim.mol.data(mol.type = "cpd", nmol = 3000)
> i <- 3
> print(demo.paths$sel.paths[i])
[1] "00640"
> pv.out <- pathview(gene.data = gse16873.d[, 1], cpd.data = sim.cpd.data, pathway.id = demo.paths$sel.paths[i], species = "hsa", out.suffix = "gse16873.cpd",keys.align = "y", kegg.native = T, key.pos = demo.paths$kpos1[i])
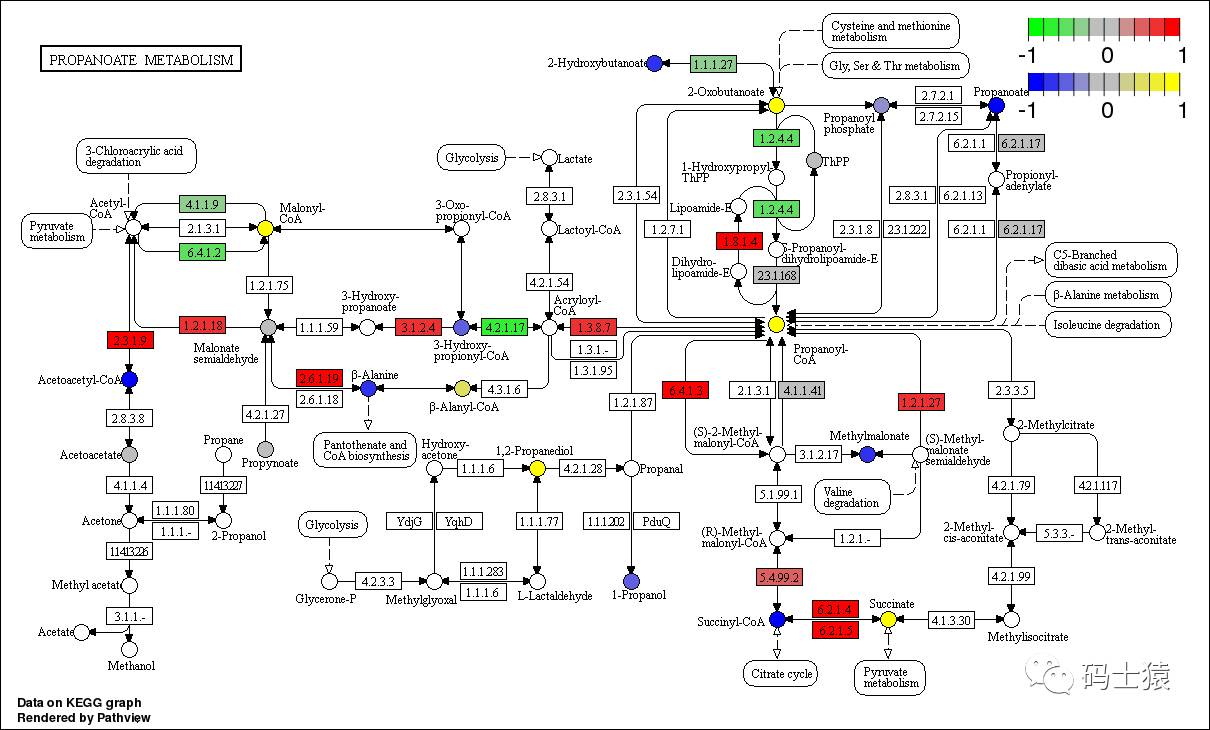
其中,cpd.data(与 gene.data 一样)为 KEGG 的化合物 IDs(KEGG compound IDs),CHEMBL 数据库中超过 20 种 ID 都可以用在这里。gene.data 与 cpd.data 不能同时为空。
ok,就先介绍到这里,更加详细的使用请参考:
http://pathview.r-forge.r-project.org/
https://www.rdocumentation.org/packages/pathview/versions/1.12.0/topics/pathview
本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
R 包 pathview 代谢通路可视化的更多相关文章
- 多组学分析及可视化R包
最近打算开始写一个多组学(包括宏基因组/16S/转录组/蛋白组/代谢组)关联分析的R包,避免重复造轮子,在开始之前随便在网上调研了下目前已有的R包工具,部分罗列如下: 1. mixOmics 应该是在 ...
- 《R包的分类介绍》
R分析空间数据(Spatial Data) R机器学习包(Machine Learning) R多元统计包(Multivariate Statistics) R药物(代谢)动力学数据分析包 R计算计量 ...
- R包MetaboAnalystR安装指南(Linux环境非root)
前言 这是代谢组学数据分析的一个R包,包括用于代谢组学数据分析.可视化和功能注释等众多功能.最近有同事在集群中搭建蛋白和代谢流程,安装这个包出现了问题,于是我折腾了一上午. 这个包的介绍在:https ...
- 利用R语言进行交互数据可视化(转)
上周在中国R语言大会北京会场上,给大家分享了如何利用R语言交互数据可视化.现场同学对这块内容颇有兴趣,故今天把一些常用的交互可视化的R包搬出来与大家分享. rCharts包 说起R语言的交互包,第一个 ...
- R 包
[下面列出每个步骤最有用的一些R包] .数据导入 以下R包主要用于数据导入和保存数据: feather:一种快速,轻量级的文件格式:在R和python上都可使用 readr:实现表格数据的快速导入 r ...
- 开发自己的R包(转)
R不必说,数据统计分析可视化的必备语言,R包开发的门槛比较低,所以现在随便一篇文章都会发表一个自己的R包,这样有好处(各种需求早有人帮你解决了)也有坏处(R包太多,混乱,新手上手较难).作为生信工程师 ...
- GO 和 KEGG 的区别 | GO KEGG数据库用法 | 基因集功能注释 | 代谢通路富集
一直都搞不清楚这两者的具体区别. 其实初学者搞不清楚很正常,因为它们的本质是相通的,都是对基因进行归类注释的数据库. 建议初学者自己使用一下这两个数据库,应该很快就能明白其中的区别. (抱歉之前没讲清 ...
- 如何制作自己的R包?
摘自 方匡南 等编著<R数据分析-方法与案例详解>.电子工业出版社 R包简介 R包提供了一个加载所需代码.数据和文件的集合.R软件自身就包含大约30种不同功能的包,这些基本包提供了R软件的 ...
- 如何制作自己的R包
如何制作自己的R包? 摘自 方匡南 等编著<R数据分析-方法与案例详解>.电子工业出版社 R包简介 R包提供了一个加载所需代码.数据和文件的集合.R软件自身就包含大约30种不同功能的包,这 ...
- R包对植物进行GO,KEGG注释
1.安装,加载所用到到R包 用BiocManager安装,可同时加载依赖包 source("https://bioconductor.org/biocLite.R") BiocMa ...
随机推荐
- XAML 设计器已意外退出。(退出代码: e0434352)
一.前言 开门见山,这个问题我遇到过两次,第一次因为项目刚开始不长时间,我查了很长时间都没解决,然后就直接重写了,几乎一样的写法,但问题没复现了,但程序员思维告诉我,一定还是有比较关键的地方出现了问题 ...
- window身上的方法 弹出框/打开和关闭
window身上的方法内置函数 alert() parseInt() parseFloat() setInterval(); setTimeout(); clearTimeout(); clearIn ...
- vivo 手机云服务建设之路-平台产品系列04
作者:vivo 互联网平台产品研发团队 - He Zhichuang.Han Lei 手机云服务目前作为每家手机厂商必备的一项基础服务,其服务能力和服务质量对用户来说可以说是非常重要.用户将自己大量的 ...
- 关于Docker compose值IP与域名的映射 之 extra_host
公司的所有项目都是采用Docker容器化部署,最近有一个项目需要使用定时任务调用第三方Api,正式web环境服务器的网络与第三方网络是通畅的,但是当将代码发布到正式环境,调用接口却显示 System. ...
- JVM 监控和故障处理总结
JDK命令工具 jps (JVM Process Status):类似 UNIX 的 ps 命令.用户查看所有 Java 进程的启动类.传入参数和 Java 虚拟机参数等信息 jstat (JVM S ...
- 系统评价——主成分分析PCA的R语言实现(六)
主成分分析(Principal Component Analysis,PCA),是将多个变量通过线性变换以选出较少个数重要变量的一种多元统计分析方法,起到数据约减和集成的作用.在许多领域的研究与应用中 ...
- day29:计算机网络概念
目录 1.网络开发的两大架构 2.网络概念 3.OSI七层模型 4.ARP协议 5.TCP三次握手和四次挥手 1.网络开发的两大架构 1.没有网络的时候,文件是如何传输的? 早期没有网络 a.py - ...
- Kubuesphere部署Ruoyi(三):持久化存储配置
按照如下教程配置NFS 先服务器: https://kubesphere.io/zh/docs/v3.3/reference/storage-system-installation/nfs-serve ...
- Network Science: 巴拉巴西网络科学阅读笔记
前言: 最小生成树中Kruskal算法对应了统计物理中的著名模型invasion percolation.由此写了一篇文章:invasion percolation and global optimi ...
- [C++提高编程] 3.1 string容器
文章目录 3.1 string容器 3.1.1 string基本概念 3.1.2 string构造函数 3.1.3 string赋值操作 3.1.4 string字符串拼接 3.1.5 string查 ...
