吴裕雄--天生自然 R语言开发学习:图形初阶





















# ----------------------------------------------------#
# R in Action (2nd ed): Chapter 3 #
# Getting started with graphs #
# requires that the Hmisc and RColorBrewer packages #
# have been installed #
# install.packages(c("Hmisc", "RColorBrewer")) #
#-----------------------------------------------------# par(ask=TRUE)
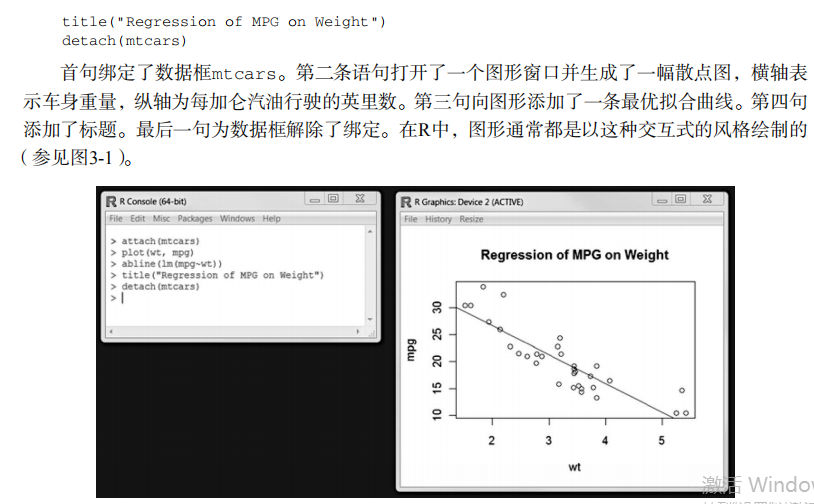
opar <- par(no.readonly=TRUE) # make a copy of current settings attach(mtcars) # be sure to execute this line plot(wt, mpg)
abline(lm(mpg~wt))
title("Regression of MPG on Weight")
# Input data for drug example
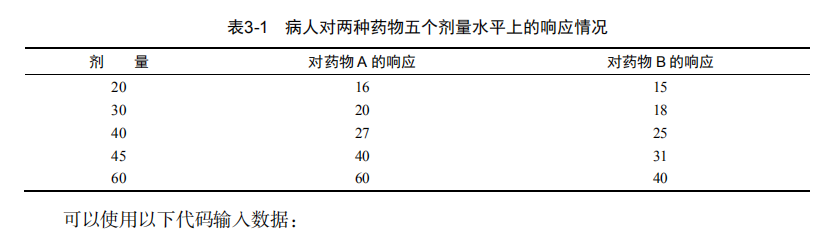
dose <- c(20, 30, 40, 45, 60)
drugA <- c(16, 20, 27, 40, 60)
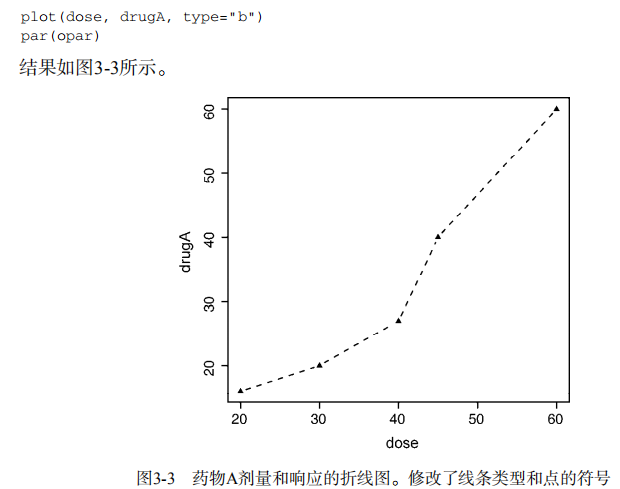
drugB <- c(15, 18, 25, 31, 40) plot(dose, drugA, type="b") opar <- par(no.readonly=TRUE) # make a copy of current settings
par(lty=2, pch=17) # change line type and symbol
plot(dose, drugA, type="b") # generate a plot
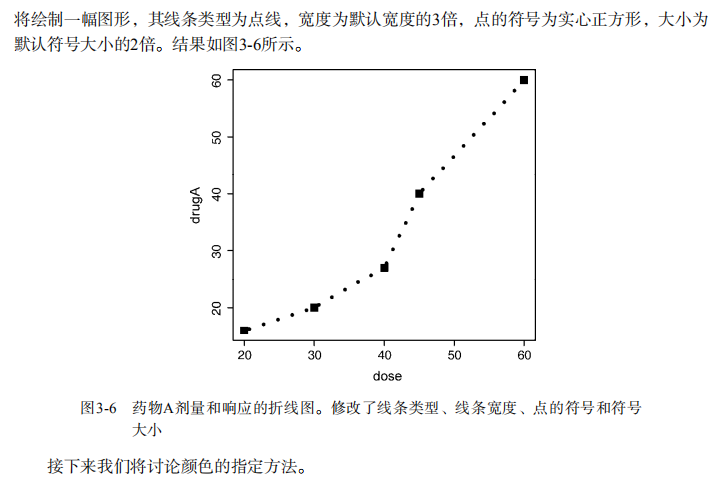
par(opar) # restore the original settings plot(dose, drugA, type="b", lty=3, lwd=3, pch=15, cex=2) # choosing colors
library(RColorBrewer)
n <- 7
mycolors <- brewer.pal(n, "Set1")
barplot(rep(1,n), col=mycolors) n <- 10
mycolors <- rainbow(n)
pie(rep(1, n), labels=mycolors, col=mycolors)
mygrays <- gray(0:n/n)
pie(rep(1, n), labels=mygrays, col=mygrays) # Listing 3.1 - Using graphical parameters to control graph appearance
dose <- c(20, 30, 40, 45, 60)
drugA <- c(16, 20, 27, 40, 60)
drugB <- c(15, 18, 25, 31, 40)
opar <- par(no.readonly=TRUE)
par(pin=c(2, 3))
par(lwd=2, cex=1.5)
par(cex.axis=.75, font.axis=3)
plot(dose, drugA, type="b", pch=19, lty=2, col="red")
plot(dose, drugB, type="b", pch=23, lty=6, col="blue", bg="green")
par(opar) # Adding text, lines, and symbols
plot(dose, drugA, type="b",
col="red", lty=2, pch=2, lwd=2,
main="Clinical Trials for Drug A",
sub="This is hypothetical data",
xlab="Dosage", ylab="Drug Response",
xlim=c(0, 60), ylim=c(0, 70)) # Listing 3.2 - An Example of Custom Axes
x <- c(1:10)
y <- x
z <- 10/x
opar <- par(no.readonly=TRUE)
par(mar=c(5, 4, 4, 8) + 0.1)
plot(x, y, type="b",
pch=21, col="red",
yaxt="n", lty=3, ann=FALSE)
lines(x, z, type="b", pch=22, col="blue", lty=2)
axis(2, at=x, labels=x, col.axis="red", las=2)
axis(4, at=z, labels=round(z, digits=2),
col.axis="blue", las=2, cex.axis=0.7, tck=-.01)
mtext("y=1/x", side=4, line=3, cex.lab=1, las=2, col="blue")
title("An Example of Creative Axes",
xlab="X values",
ylab="Y=X")
par(opar) # Listing 3.3 - Comparing Drug A and Drug B response by dose
dose <- c(20, 30, 40, 45, 60)
drugA <- c(16, 20, 27, 40, 60)
drugB <- c(15, 18, 25, 31, 40)
opar <- par(no.readonly=TRUE)
par(lwd=2, cex=1.5, font.lab=2)
plot(dose, drugA, type="b",
pch=15, lty=1, col="red", ylim=c(0, 60),
main="Drug A vs. Drug B",
xlab="Drug Dosage", ylab="Drug Response")
lines(dose, drugB, type="b",
pch=17, lty=2, col="blue")
abline(h=c(30), lwd=1.5, lty=2, col="gray")
library(Hmisc)
minor.tick(nx=3, ny=3, tick.ratio=0.5)
legend("topleft", inset=.05, title="Drug Type", c("A","B"),
lty=c(1, 2), pch=c(15, 17), col=c("red", "blue"))
par(opar) # Example of labeling points
attach(mtcars)
plot(wt, mpg,
main="Mileage vs. Car Weight",
xlab="Weight", ylab="Mileage",
pch=18, col="blue")
text(wt, mpg,
row.names(mtcars),
cex=0.6, pos=4, col="red")
detach(mtcars) # View font families
opar <- par(no.readonly=TRUE)
par(cex=1.5)
plot(1:7,1:7,type="n")
text(3,3,"Example of default text")
text(4,4,family="mono","Example of mono-spaced text")
text(5,5,family="serif","Example of serif text")
par(opar) # Combining graphs
attach(mtcars)
opar <- par(no.readonly=TRUE)
par(mfrow=c(2,2))
plot(wt,mpg, main="Scatterplot of wt vs. mpg")
plot(wt,disp, main="Scatterplot of wt vs. disp")
hist(wt, main="Histogram of wt")
boxplot(wt, main="Boxplot of wt")
par(opar)
detach(mtcars) attach(mtcars)
opar <- par(no.readonly=TRUE)
par(mfrow=c(3,1))
hist(wt)
hist(mpg)
hist(disp)
par(opar)
detach(mtcars) attach(mtcars)
layout(matrix(c(1,1,2,3), 2, 2, byrow = TRUE))
hist(wt)
hist(mpg)
hist(disp)
detach(mtcars) attach(mtcars)
layout(matrix(c(1, 1, 2, 3), 2, 2, byrow = TRUE),
widths=c(3, 1), heights=c(1, 2))
hist(wt)
hist(mpg)
hist(disp)
detach(mtcars) # Listing 3.4 - Fine placement of figures in a graph
opar <- par(no.readonly=TRUE)
par(fig=c(0, 0.8, 0, 0.8))
plot(mtcars$mpg, mtcars$wt,
xlab="Miles Per Gallon",
ylab="Car Weight")
par(fig=c(0, 0.8, 0.55, 1), new=TRUE)
boxplot(mtcars$mpg, horizontal=TRUE, axes=FALSE)
par(fig=c(0.65, 1, 0, 0.8), new=TRUE)
boxplot(mtcars$wt, axes=FALSE)
mtext("Enhanced Scatterplot", side=3, outer=TRUE, line=-3)
par(opar)
吴裕雄--天生自然 R语言开发学习:图形初阶的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:基础知识
1.基础数据结构 1.1 向量 # 创建向量a a <- c(1,2,3) print(a) 1.2 矩阵 #创建矩阵 mymat <- matrix(c(1:10), nrow=2, n ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- Python 学习笔记:根据输入年月区间,返回期间所有的月份
目的: 给定一个年月区间,比如:2019.01 至 2019.05,要求返回一个包含期间所有的月份的列表,比如:['2019.01', '2019.02', '2019.03', '2019.04', ...
- swoole使用异步redis
1.lnmp安装redis拓展 wget http://download.redis.io/releases/redis-4.0.9.tar.gz chmod 755 redis-4.0.9.tar. ...
- dubbo的超时处理和配置覆盖
提供者的设置方式 消费者的设置方式 配置原则 dubbo推荐在Provider上尽量多配置Consumer端属性: 1.作服务的提供者,比服务使用方更清楚服务性能参数,如调用的超时时间,合理的重试次数 ...
- 11)PHP,单选框和复选框的post提交方式处理
就是一个表单中会有input的checkbox形式,那么怎么处理,就有了问题,一般采用二维数组来处理 代码展示: <!DOCTYPE html PUBLIC "-//W3C//DTD ...
- 吴裕雄--天生自然Linux操作系统:Linux常用命令大全
系统信息 arch 显示机器的处理器架构 uname -m 显示机器的处理器架构 uname -r 显示正在使用的内核版本 dmidecode -q 显示硬件系统部件 - (SMBIOS / DMI) ...
- [思路笔记]WEB安全之漏洞挖掘
记录自己在实际渗透测试以及漏洞挖掘中会用到的思路和方法.不断完善,尽量以系统的方式展现程序化式的漏洞挖掘.由于各种原因,不便公开. 通用策略 1.信息搜集 : 数据挖掘.业务挖掘 数据: 邮箱.手机号 ...
- android中的适配器模式
原文: https://blog.csdn.net/beyond0525/article/details/22814129 类适配模式.对象适配模式.接口适配模式
- 与http协作的web服务器
1.虚拟主机 一台web服务器可以搭建多个独立域名的web网站,也可以作为通信线路上的中转服务器提升传输效率.比如web托管服务供应商可以用一台服务器为多个不同域名的客户提供服务,这是利用了虚拟主机( ...
- Linux虚拟机添加硬盘
任务:添加1块硬盘,并且分1个区,挂载到/bak 第一.插上硬盘 第二.分区 第三.格式化(定义:文件系统的类型) FAT,FAT32,NTFS,ext1,ext2,ext3,ext4,.... 第 ...
- SQLServer 安装提示需要重启计算机的解决方案
处理方法:在开始-程序-运行中(或者直接windows+R件同时),输入regedit回车,在注册表中找到HKEY_LOCAL_MACHINE\SYSTEM\CurrentControlSet\Con ...
