为什么 Biopython 的在线 BLAST 这么慢?
用过网页版本 BLAST 的童鞋都会发现,提交的序列比对往往在几分钟,甚至几十秒就可以得到比对的结果;而通过调用 API 却要花费几十分钟或者更长的时间!这到底是为什么呢?

NCBIWWW 基本用法
首先,我们来看一下提供了基于 API 在线比对的 Biopython 模块。
Biopython 中的 BLAST 提供了 over the Internet 和 locally 两种选择:Bio.Blast.NCBIWWW 主要是基于 NCBI BLAST API 用于在线比对;Bio.Blast.Applications 模块则是调用本地安装好的 BLAST 程序以及数据库执行比对。在这里我们来重点看一下 Bio.Blast.NCBIWWW 。
Bio.Blast.NCBIWWW 模块中主要是通过 qblast() 函数来调用 BLAST 的在线版本。它具有三个非可选参数:
第一个参数是用于搜索的 blast 程序,为小写字符串。目前,
qblast(biopython==1.7.4)仅适用于 blastn,blastp,blastx,tblast 和 tblastx。第二个参数指定要搜索的数据库。关于这个选项,在 NCBI Guide to BLAST 上有详细的描述。
第三个参数是包含查询序列的字符串。这可以是序列本身,也可以是 fasta 格式的序列,或者是诸如 GI 号之类的标识符。
qblast 函数还接受许多其他选项参数,这些参数基本上类似于我们可以在 BLAST 网页上设置的不同参数。我们在这里只重点介绍其中一些:
参数
url_base是设置用于在 Internet 上运行 BLAST 的基本 URL。默认情况下,它连接到 NCBI(即 url_base='https://blast.ncbi.nlm.nih.gov/Blast.cgi'),但是可以使用它连接到云端运行的 NCBI BLAST 实例。更多详细信息请参阅 qblast 功能的文档。qblast 函数可以返回各种格式的 BLAST 结果,您可以使用可选的
format_type关键字进行选择:“HTML”,“Text”,"ASN.1” 或 "XML"。默认值为 “XML”,因为这是解析器期望的格式。参数
expect用于设置期望值或 e-value 阈值。
有关可选的 BLAST 参数的更多信息,请参考 NCBI 自己的文档或 Biopython 内置的文档:
>>> from Bio.Blast import NCBIWWW
>>> help(NCBIWWW.qblast)
请注意,NCBI BLAST 网站上的默认设置与 qblast 上的默认设置不太相同。如果获得不同的结果,则需要检查参数(例如,e-value 值和 gap 值)。
例如,如果您要使用 BLASTN 在核苷酸数据库(nt)中搜索核苷酸序列,并且知道查询序列的 GI 号,则可以使用:
>>> from Bio.Blast import NCBIWWW
>>> result_handle = NCBIWWW.qblast("blastn", "nt", "8332116")
另外,如果我们的查询序列已经存在于 FASTA 格式的文件中,则只需打开文件并以字符串形式读取此记录,然后将其用作查询参数:
>>> from Bio.Blast import NCBIWWW
>>> fasta_string = open("m_cold.fasta").read()
>>> result_handle = NCBIWWW.qblast("blastn", "nt", fasta_string)
我们还可以将 FASTA 文件作为
SeqRecord 对象进行读取,然后仅提供序列本身进行比对:
>>> from Bio.Blast import NCBIWWW
>>> from Bio import SeqIO
>>> record = SeqIO.read("m_cold.fasta", format="fasta")
>>> result_handle = NCBIWWW.qblast("blastn", "nt", record.seq)
仅提供序列意味着 BLAST 将自动为您的序列分配一个标识符。您可能更喜欢使用
SeqRecord 对象的
format 方法来制作 FASTA 字符串(其中将包含现有标识符):
>>> from Bio.Blast import NCBIWWW
>>> from Bio import SeqIO
>>> record = SeqIO.read("m_cold.fasta", format="fasta")
>>> result_handle = NCBIWWW.qblast("blastn", "nt", record.format("fasta"))
无论给 qblast() 函数提供什么参数,都应在 handle 对象(默认为 XML 格式)中返回结果。下一步是将 XML 输出解析为表示搜索结果的 Python 对象,但是您可能想先保存输出文件的本地副本。在调试从 BLAST 结果中提取信息的代码时,我发现这特别有用(因为重新运行在线搜索速度很慢,并且浪费了 NCBI 计算机时间)。
我们需要小心一点,因为我们只能使用
result_handle.read() 读取一次 BLAST 输出——再次调用
result_handle.read() 会返回一个空字符串。
>>> with open("my_blast.xml", "w") as out_handle:
... out_handle.write(result_handle.read())
...
>>> result_handle.close()
完成上面的操作后,结果将保存在文件 my_blast.xml 中,并且原始句柄已提取了所有数据(因此我们将其关闭了)。但是,BLAST 解析器的解析功能采用了类似于文件句柄的对象,因此我们可以打开保存的文件进行输入:
>>> result_handle = open("my_blast.xml")
现在我们已经将 BLAST 结果重新放回了句柄中,下一步,如果我们准备对它们进行处理,我们可以参考 Biopython 中 Parsing BLAST output 部分的内容,这里不再说明。
NCBIWWW 实现
在了解 NCBIWWW 的实现前,我们先来看一下 NCBI BLAST 对于 API 使用的一些说明:
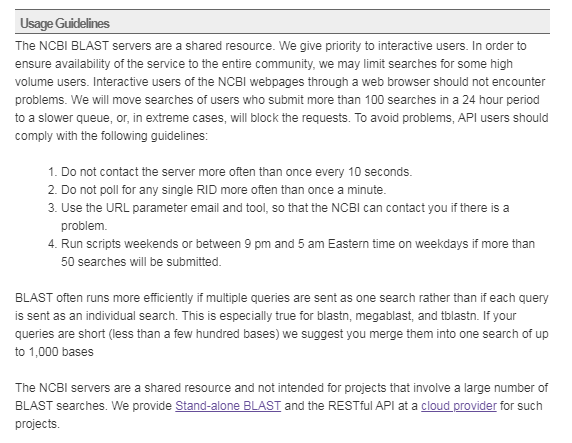
NCBI BLAST 服务器是共享资源。为了确保整个社区都能使用该服务,他们可能会限制某些高流量用户的搜索。
他们会将在 24 小时内提交 100 次以上搜索的用户的搜索移到较慢的队列中,或者在极端情况下将阻止请求。
NCBI BLAST 优先考虑互动的用户,通过网络浏览器的 NCBI 网页的交互式用户不会遇到以上的问题。
对于 API 的使用准则:
与服务器联系的频率不要超过每 10 秒一次。
不要轮询每一个 RID(Request ID) 多于一分钟一次。
使用 URL 参数电子邮件和工具,以便 NCBI 在出现问题时可以与您联系。
如果将提交超过 50 个搜索,则在周末或东部时间东部时间晚上 9 点至凌晨 5 点之间运行脚本。
我们再来看一下 NCBIWWW 在源码层面的处理:
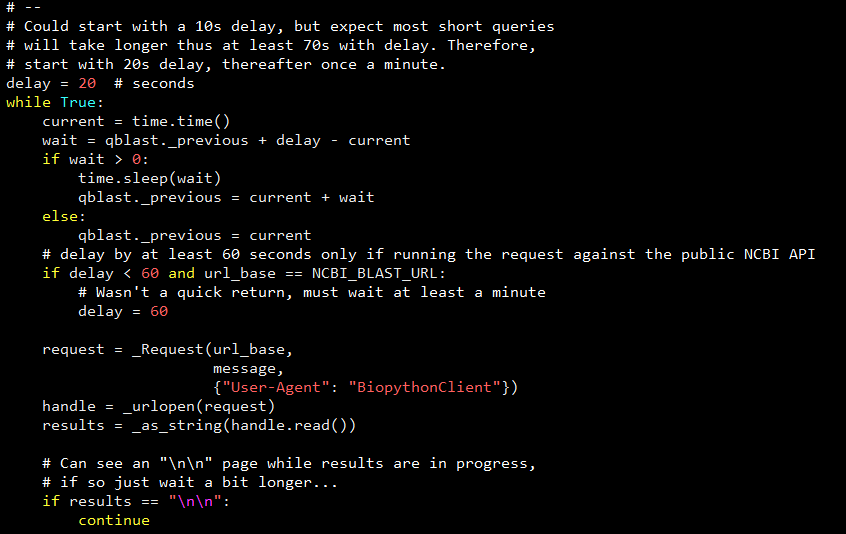
可以看到 NCBIWWW 从 20 秒的延迟开始,然后开始每隔一分钟执行一次 request 轮询,直至任务完成或者任务出现异常。
所以,总的来说,NCBI BLAST API 的使用准则,加上 NCBI BLAST 对用户请求的任务队列处理,甚至 NCBI BLAST 服务器共享资源的限制,以及总用户请求数,这些都可能成为 NCBIWWW.qblast() 异常耗时的原因,这其中还不算个人服务器的网络影响。综上种种原因,如果考虑使用 NCBIWWW.qblast() 执行频繁的序列在线批处理,或许不是一个好的解决方案。
最后,基于 Python 的 NCBI BLAST 在线批处理,如果你有更好的方法,欢迎留言交流。

本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
为什么 Biopython 的在线 BLAST 这么慢?的更多相关文章
- 4、在线blast比对结果解析(保守结构域)
转载:http://www.bio1000.com/experiment/fenzi/237846.html 标签: NCBI Blast LASTP 摘要 : NCBI BLAST比对结果报告分析: ...
- blast 数据库说明
Peptide Sequence Databases蛋白序列的数据库 nrAll non-redundant GenBank CDS translations + RefSeq Proteins + ...
- Biopython常用功能模块
Biopython项目是旨在减少计算生物学中代码重复的开源项目之一,由国际开发人员协会创建. 它包含表示生物序列和序列注释的类,并且能够读取和写入各种文件格式(FASTA,FASTQ,GenBank和 ...
- BLAST在Windows系统中本地化
简介 NCBI除了提供在线的Web BLAST序列比对服务外,还提供FTP方式下载序列比对工具.这允许在本地平台上针对从NCBI下载或本地创建的数据库执行BLAST搜索.这些实用程序没有图形用户界面, ...
- 从直播编程到直播教育:LiveEdu.tv开启多元化的在线学习直播时代
2015年9月,一个叫Livecoding.tv的网站在互联网上引起了编程界的注意.缘于Pingwest品玩的一位编辑在上网时无意中发现了这个网站,并写了一篇文章<一个比直播睡觉更奇怪的网站:直 ...
- Scrapy框架爬虫初探——中关村在线手机参数数据爬取
关于Scrapy如何安装部署的文章已经相当多了,但是网上实战的例子还不是很多,近来正好在学习该爬虫框架,就简单写了个Spider Demo来实践.作为硬件数码控,我选择了经常光顾的中关村在线的手机页面 ...
- 基于本地存储的kvm虚拟机在线迁移
基于本地存储的kvm虚拟机在线迁移 kvm虚拟机迁移分为4种(1)热迁移基于共享存储(2)热迁移基于本地存储(3)冷迁移基于共享存储(4)冷迁移基于本地存储 这里介绍的是基于本地存储的热迁移 动态块迁 ...
- 关于开启.NET在线提升教育培训的通知! - 可在此页面观看在线直播!
年前在线公开课程通知: 近期在开启VIP课程,隔天讲一次,年前其它时间插空讲公开课,主题:设计模式系列 1:培训 - 大概不会讲的内容: 1:不讲系列. 2:不讲入门. 3:不讲我不懂的! 2:培训 ...
- 在ASP.NET Core中使用百度在线编辑器UEditor
在ASP.NET Core中使用百度在线编辑器UEditor 0x00 起因 最近需要一个在线编辑器,之前听人说过百度的UEditor不错,去官网下了一个.不过服务端只有ASP.NET版的,如果是为了 ...
- 在线浏览PDF之PDF.JS (附demo)
平台之大势何人能挡? 带着你的Net飞奔吧!:http://www.cnblogs.com/dunitian/p/4822808.html#skill 下载地址:http://mozilla.gith ...
随机推荐
- StyleGAN 调整面部表情,让虚拟人脸更生动
目录 人脸表情 调整步骤 调整结果 人脸表情 通过上一篇文章 StyleGAN 生成的人脸:https://www.cnblogs.com/tinygeeker/p/17236264.html 人脸图 ...
- Github Copilot 比在座各位更会写代码。jpg
之前大佬和我安利过 Copilot, 作为一个能用就行的践行者, 我一贯对这些东西都不太感兴趣. 就如我多年VS Code写各种编程语言, jetbrains 全家桶我都懒得搞~ 不过最近看到过Cha ...
- CSAPP-Architecture Lab
Part A 前置准备 gcc -Wall -O1 -g -c yis.c gcc -Wall -O1 -g -c isa.c gcc -Wall -O1 -g yis.o isa.o -o yis ...
- 机器学习基础05DAY
分类算法之k-近邻 k-近邻算法采用测量不同特征值之间的距离来进行分类 优点:精度高.对异常值不敏感.无数据输入假定 缺点:计算复杂度高.空间复杂度高 使用数据范围:数值型和标称型 一个例子弄懂k-近 ...
- ChatGPT 开源了第一款插件,都来学习一下源码吧!
3 月 23 日,OpenAI 又投出了一枚重磅炸弹:为 ChatGPT 推出插件系统! 此举意味着 ChatGPT 将迎来"APP Store"时刻,也就是围绕它的能力,形成一个 ...
- PVE虚拟系统的安装
上期有说到,会出一些具体的安装过程以及掉进去的坑.这集我就说一说PVE系统的安装. As mentioned in the previous issue, there will be some ...
- 联邦学习FATE框架安装搭建
联邦学习 FATE (Federated AI Technology Enabler) 是微众银行AI部门发起的开源项目,为联邦学习生态系统提供了可靠的安全计算框架.FATE项目使用多方安全计算 (M ...
- 内网搭建DNS服务器
DNS:Domain Name Service,域名解析服务 监听端口:udp/53,tcp/53 应用程序:bind 根域:. 一级域: 组织域:.com, .org, .net, .mil, .e ...
- 自建kms2种方法,亲测有效
你还在用网上那种坑爹的Ghost盗版系统吗?内置各种辣鸡.流氓软件,新系统?纯净版Ghost系统?不存在的~ 在开始自建KMS服务器之前,我觉得很有必要给大家分享一个神站,没错就是MSDN! 地址:h ...
- pysimplegui之进度表one_line_progress_meter
我们的代码中都有循环.'等待,看着文本窗口中滚动过去的计数器不是很快乐吗?一行代码如何获得一个进度表,其中包含有关您的代码的统计信息? one_line_progress_meter(title, c ...
