6Sampling Configuration Space: 6.3 Using Accelerated Molecular Dynamics (aMD) to enhance sampling
6.3 Using Accelerated Molecular Dynamics (aMD) to enhance sampling
英文官网:http://ambermd.org/tutorials/advanced/tutorial22/index.php
另外可参考 https://blog.chembiosim.com/aMD_in_Amber/
传统的分子动力学允许人们访问数十到数百纳秒的时间尺度; 然而,许多感兴趣的生物过程发生在长达几毫秒或更长时间的较长时间尺度上。
加速动力学是对势能的一种修改——在模拟时减少局域barriers的高度,允许计算得更快。aMD是一个有趣的选择,因为它只需要单个副本的进化,并且它不需要任何关于势能面的先前知识。
aMD 有两种主要版本,一种提高山谷,另一种是降低障碍(山坡)。 后者最近才被引入,它的可用性和好处尚未得到充分研究。 这种方法通常被称为加窗 aMD (w-aMD)。 在本教程中,我们将使用原始的 aMD 公式,但是这两个版本现在都可以在 AMBER 上使用。基础aMD引擎对势能的修改被以下方程定义:
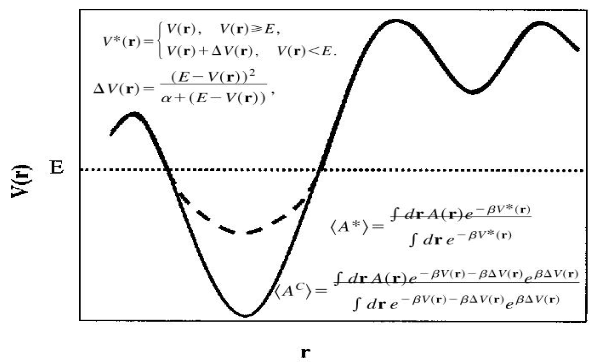
当aMD在AMBER中的实施既可以lowering barriers,又可以raising minima。aMD可以在AMBER的所有MD引擎(sander,pmemd和pmemd.cuda)中实施。The implementation includes the possibility of boosting independently only the torsional terms of the potential (iamd=2) or the whole potential at once (iamd=1). It also allows the possibility to boost the whole potential with an extra boost to the torsions(iamd=3). (目前没搞懂这句的意思)。在aMD中唯一额外需要用户给出的参数是:
a) EthreshP. Average total potential energy threshold. 平均总势能阈值
b) alphaP. Inverse strength boost factor for the total potential energy.总势能的逆强度提升因子
c) EthreshD. Average dihedral energy threshold. 平均二面角能量阈值
d) alphaD. Inverse strength boost factor for the dihedral energy. 二面角能量的逆强度提升因子
本教程将介绍使用aMD的raising minima的采样能力来研究BPTI在微秒时间尺度上发生的构象转变,与毫秒的传统分子动力学模拟进行比较。
Section 1
1) Generating and Relaxing the Initial Structure
普通的MD,将体系平衡。
- Minimize only the water, restraining the protein (20000 cycles)
- Let water move (NTP, 300K), restraining the protein
- Minimize water and protein (20000 cycles)
- Heat the system, restraining the protein (NVT 0 to 300K)
- Relax the system, restraining the protein heavy atoms (NPT, 300K, 0.5ns)
- Relax the system (NPT, 300K, 5ns)
2) Running the aMD calculations and data collection.
有了一个平衡的初始结构后,首先需要运行一个简短的常规MD模拟以收集必要的信息来设置必要的aMD参数。
NSTEP = 39000 TIME(PS) = 6078.000 TEMP(K) = 299.91 PRESS = 94.7
Etot = -39262.0762 EKtot = 7879.4131 EPtot = -47141.4893
BOND = 183.1676 ANGLE = 426.5374 DIHED = 594.3544
1-4 NB = 204.0621 1-4 EEL = 1794.3799 VDWAALS = 7733.9917
EELEC = -58077.9825 EHBOND = 0.0000 RESTRAINT = 0.0000
EKCMT = 3596.2487 VIRIAL = 3332.2057 VOLUME = 129191.4706
Density = 1.0200上述结果只是展示某一帧的能量结果。比如,我们从5ns的MD中获得了 -47,128 kcal/mol 的平均总势能(EPtot)和 595 kcal/mol 的平均二面角(DIHED)能量。 使用这些信息,并考虑到 BPTI 体系有 58 个残基,整个系统 18,226 个原子,我们可以计算 aMD 参数如下:
a) EthreshP: E(tot)= -47128 kcal mol-1 + (0.16kcal*mol-1 atom-1 * 18,226 atoms) = -44212 kcal mol-1
b) alphaP: Alpha(tot)= (0.16kcal mol-1 atom-1 * 18,226 atoms) = 2916 kcal mol-1
c) EthreshD: E(dih)=595 kcal mol-1 + (4kcal mol-1 residue-1 * 58 solute residues) = 827 kcal mol-1
d) alphaD: Alpha(dih)=(1/5)*(4kcal mol-1 residues-1 * 58 solute residues) = 46.4 kcal mol-1
我们现在可以运行完整的 500 ns 模拟,如下所示:
500 ps NVT production
NVT
&cntrl
imin=0,irest=1,ntx=5,
nstlim=250000000,dt=0.002,
ntc=2,ntf=2,ig=-1,
cut=10.0, ntb=1, ntp=0,
ntpr=1000, ntwx=1000,
ntt=3, gamma_ln=2.0,
temp0=300.0,ioutfm=1,iwrap=1,
iamd=3,
ethreshd=827, alphad=46.4,
ethreshp=-44212, alphap=2916,
/
pmemd.cuda -O -i amd.in -o amd.out -p ../*.prmtop -c ../eq.rst -r amd.rst -x prod.nc
3) Analyzing the aMD results
未详细看……
4) Reweighting the aMD results
未详细看……
可以重新加权在模拟中获得的分布,以获得未受干扰的分布。
6Sampling Configuration Space: 6.3 Using Accelerated Molecular Dynamics (aMD) to enhance sampling的更多相关文章
- 机器人学 —— 轨迹规划(Configuration Space)
之前的轨迹规划中,我们只考虑了质点,没有考虑机器人的外形与结构.直接在obstacle map 中进行轨迹规划,然而世纪情况中,机器人有固定外形,可能会和障碍物发生碰撞.此情况下,我们针对机器人自由度 ...
- 机器人运动规划中的构形空间(Configuration Space)
A key concept in motion planning is configuration space, or C-space for short. Every point in the C- ...
- Molecular Dynamics
First]前处理 Second]运行MD Third]后处理 一.获得结构文件-来自实验数据或者某些化学软件工具 1]第一步: 获取并处理pdb文件 从Protein Data Bank下载小肽的p ...
- PCI & PCIE Configuration Register Space
1.PCI-Compatible Configurationbits [1:0] are hard-wired, read-only and must return zeros when read.b ...
- [中英对照]Device Drivers in User Space: A Case for Network Device Driver | 用户态设备驱动: 以网卡驱动为例
前文初步介绍了Linux用户态设备驱动,本文将介绍一个典型的案例.Again, 如对Linux用户态设备驱动程序开发感兴趣,请阅读本文,否则请飘过. Device Drivers in User Sp ...
- Spring源码解析之@Configuration
@Configuration简介 用于标识一个类为配置类,与xml配置效果类似 用法简介 public class TestApplication { public static void main( ...
- Monitoring and Tuning the Linux Networking Stack: Receiving Data
http://blog.packagecloud.io/eng/2016/06/22/monitoring-tuning-linux-networking-stack-receiving-data/ ...
- zhihu spark集群,书籍,论文
spark集群中的节点可以只处理自身独立数据库里的数据,然后汇总吗? 修改 我将spark搭建在两台机器上,其中一台既是master又是slave,另一台是slave,两台机器上均装有独立的mongo ...
- 《Monitoring and Tuning the Linux Networking Stack: Receiving Data》翻译
Overview 从宏观的角度来看,一个packet从网卡到socket接收缓冲区的路径如下所示: 驱动加载并初始化 packet到达网卡 packet通过DMA被拷贝到内核中的一个ring buff ...
- NVIDIA---CUDA
http://en.wikipedia.org/wiki/CUDA CUDA From Wikipedia, the free encyclopedia CUDA Developer(s) N ...
随机推荐
- 7. 交换排序的阈值(swapThreshold)
1 <!DOCTYPE html> 2 <html lang="zh"> 3 <head> 4 <meta charset="U ...
- -bash: ./mlnxofedinstall: /usr/bin/perl: bad interpreter: No such file or directory
-bash: ./mlnxofedinstall: /usr/bin/perl: bad interpreter: No such file or directory 解决办法:安装相关的环境即可 输 ...
- mqtt安装和使用
linux下 下载: wget https://www.emqx.io/downloads/broker/v3.2.1/emqx-centos7-v3.2.1.zip 解压:unzip emqx-ce ...
- python 发送 application/x-www-form-urlencoded 类型的数据包
在网上百度了很多,坑的要死,基本不咋好用,自己多尝试了几遍,成功实现了. data 的构建方式 data = f"page={page}&sign={sign}&t={t}& ...
- C#定时任务(Timer)
新建Timer类 using BaseAsset.Data.Infrastructure; using BaseAsset.Data.Repositories; using BaseAsset.Ent ...
- Drone自动部署配置文件
.drone.yml 点击查看代码 kind: pipeline # 定义对象类型,还有secret和signature两种类型 type: docker # 定义流水线类型,还有kubernetes ...
- create-react-app卸载与升级
用create-react-app 创建项目发现报错 You are running `create-react-app` 4.0.3, which is behind the latest rele ...
- [478] C2 Age Of Splendor Opcodz
[478] C2 Age Of Splendor Client 00 SendProtocolVersion 01 MoveBackwardToLocation 02 Say 03 RequestEn ...
- win常用的dos命令
常用的dos命令 开启dos终端的两种方式: win+r后输入cmd指令 在资源管理器上方的路径窗口直接输入cmd,即可开启指定路径下的cmd终端,省去了cd/d这一步 注:在dos终端里ctrl+v ...
- pytest之conftest.py
一.conftest.py的特点 1.可以跨.py文件调用,有多个.py文件调用时,可让conftest.py只调用了一次fixture,或调用多次fixture 2.conftest.py与运行的用 ...
