Journal of Proteome Research | Improving Silkworm Genome Annotation Using a Proteogenomics Approach (分享人:张霞)
题目:Improving Silkworm Genome Annotation Using a Proteogenomics Approach
期刊:Journal of Proteome Research
发表时间:June 28, 2019
DOI:10.1021/acs.jproteome.8b00965
分享人:张霞
内容与观点:
1、 文章研究概述
桑蚕是一种重要的经济昆虫,并作为鳞翅目模式昆虫用于基础研究。作者通过以往的研究发现,目前对于桑蚕的基因测序和组装研究已基本解决,在基因注释研究方面,基于ESTs和基因序列鉴定了277可变剪接类型;基于RNA-seq鉴定了1923可变剪接,58反式剪接;基于转录蛋白组学分析,发现3247 基因可能存在可变剪接,可见对基因的注释并不完全,同时有研究表明注释误差较大,误差主要发生在阅读框架,外显子界线的确定,可变剪接额类型,使得基因模型难于预测。同时,目前的分析预测软件也存在一定的错误率。作者认为,基于质谱的蛋白数据可以为蛋白编码基因提供直接证据,所以本文欲以蛋白基因组学方法试图对桑蚕的基因进行注释研究,并辅以转录蛋白质组学进行加以验证。本文以桑蚕不同发育时期的不同组织进行质谱检测,获得980万张谱图,通过与数据库比对,经由软件分析,共鉴定了1838新转录本和1593可变剪接基因;证实了14个不确定基因和10个跳跃外显子;校正了5074个现有基因和2581非转录区。
2、 实验流程
2.1 MS 数据
选用培养和野生桑蚕不同发育阶段的不同组织(后部丝腺PSG,中部丝腺,卵巢,睾丸,头部,大脑,前胸腺,食管下神经节,血淋巴,脂肪和胚胎)进行bottom-up分析,先进行SDS-PAGE,对蛋白进行分离,接着胶内酶解,用0.1%甲酸重悬用于LC-MS/MS上机,使用iTRAQ对PSG组织进行标记定量。
2.2 数据库构建和肽段鉴定
从Silk Genome Database Website 上下载蛋白数据库Silk PDB(14623),从NCBI上下载BmPDB(7859),构建数据库SilkGDB(B.mori genome的六帧翻译数据库)和SilkASDB(外显子-外显子连接肽段数据库)以及混合数据库(Silk PDB+SilkGDB和Silk PDB+SilkASDB),质谱得到的数据通过以上数据库进行搜索鉴定。参数设置时注意以下五点:①trypsin酶切设置:允许一个missed cleavage。② 肽段谱图错误容忍度为20ppm。③片段谱图中iTRAQ容忍度为0.02Da,其他为0.6Da。④固定修饰为半胱氨酸的脲基化;可变修饰为N端乙酰化,甲硫氨酸的氧化,谷氨酰胺和天冬酰胺的脱酰胺。⑤通过decoy库比对,以FDR为1% 过滤肽段。
2.3 基因注释工作流程
通过数据库搜索,定义GSSP为仅存在于SilkGDB数据库,而不存在于Silk PDB数据库。以GSSP用来修正基因注释;以Silk PDB数据库和SilkASDB数据库搜索的肽段鉴定新的可变剪接。使用TBLASTN软件以GSSP获得相对应的ORF,若ORF与现有基因模型重叠,为固有基因;若ORF与基因间区域重叠,则为新基因。为确保结果可靠,ORF序列与EST序列进行比对鉴定单一基因,用BLAST搜索同源蛋白,以GSSP,单一基因和同源蛋白共同质控固有和新基因的鉴定。
2.4 RNA-seq分析
对桑蚕不同发育阶段的PSG进行RNA提取并测序,得9600万读数,与参考基因进行比对,鉴定可变剪接位点,校正基因模型,鉴定新基因。
2.5 GO和KEGG路径分析
以桑蚕全基因作为背景,用agriGO和Blast2GO软件对可变剪接和全基因进行GO注释。用线上KEGG进行路径注释。
3、 实验结果
3.1 传统蛋白组学
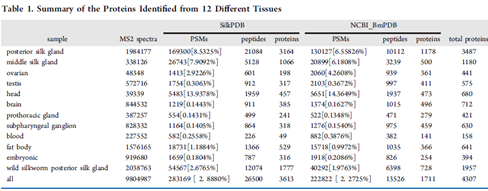
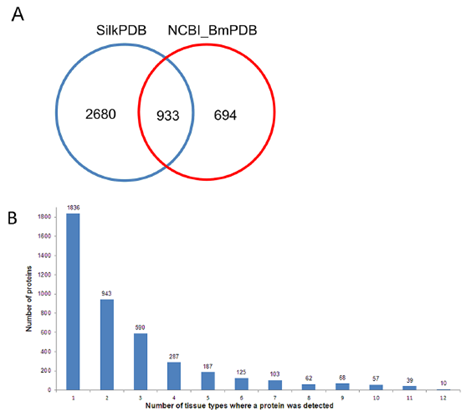
通过MS/MS谱图与参考蛋白数据库(Silk PDB和BmPDB)比对,去掉多余数据后,共鉴定4307蛋白,933个蛋白是两个数据库共有,不同的组织蛋白也不同。PSG为桑蚕中合成蛋白的主要器官,对比培养组和野生组,分别鉴定了3487和1957个蛋白,共享蛋白为1823个。结果如下表1和下图2所示。
3.2 GSSPs完善基因注释
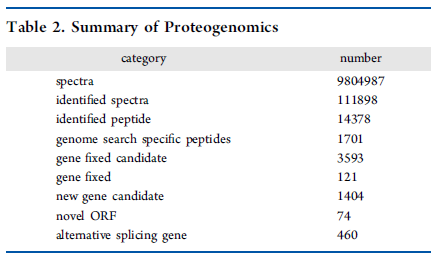
质谱图通过与SilkGDB和SilkPDB的混合数据库的匹配对比,共鉴定14378肽段序列,其中1701个为新肽段。GSSP与基因相比对,找到了74个新ORF位置,根据上述流程中介绍,结合肽段,同源蛋白,单一基因的证据,当ORF与注释基因模式重叠时为固有基因,共鉴定3593个;与注释基因不重叠的为新基因,共1404个。结果如下表2所示。
3.3 新基因
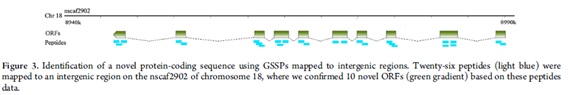
74个新ORF中,53个有同源蛋白为支撑证据,1个有单一基因为支撑证据,20个除了至少有两个GSSP外没有其他支撑证据。很多新的ORF被注释为相同预测蛋白。例如,在18号染色体上大约50kbp基因内,在26个GSSPs中证实了10个新ORF。如下图3所示。
3.4 修正注释基因模型
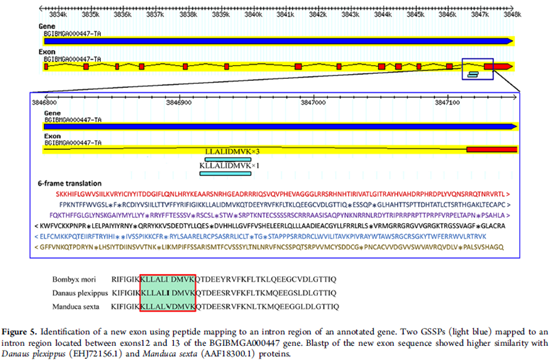
使用GSSPs与SilkPDB数据库中固有基因进行比对,以重叠结果校正了121个基因模型,这些基因中的大部分(68个基因)是外显子延伸,其中37个为第一个外显子的5‘端延伸。例如下图4所示,两个特殊新肽段比对到一个固有基因的上游;再如下图5所示,12和13外显子之间的内含子区域鉴定了2个特有GSSP,新外显子序列与Danaus plexippus (EHJ72156.1) and Manduca sexta (AAF18300.1) 蛋白具有高度相似性。
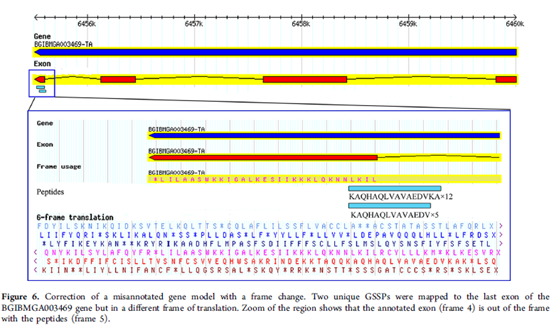
基于核苷酸的传统基因注释很难鉴定读写框,但基于肽段的方法能特异证实对应基因的转录读写框并能对错误注释进行校正。如下图6所示,两个特异GSSP比对到该基因的最后外显子上,但注释外显子frame与肽段的不同,可进行校正。

3.5 可变剪接
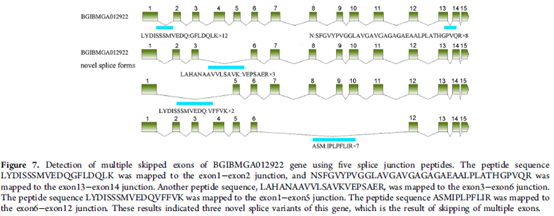
由于可变剪接的表达不高,所以很难判断, 且一般从mRNA判定,容易包含可变剪接前体序列;基于蛋白质水平的质谱数据,可通过鉴定连接肽段,进而鉴定剪接连接以及发现新的剪接类型。可构建外显子跳跃数据库SilkASDB(一个基因中有两个外显子),搜索连接肽段,通过与质谱图进行比对,找到1330个剪接交界跨肽,从527个基因中找到1105个剪接位点,从148个剪接交界跨肽中找到132个已知剪接连接。下图7所示为5个剪接连接肽段鉴定了基因的多个跳跃外显子并发现了新的剪接类型。
3.6 通过RNA-seq提高基因注释
结合RNA-seq方法以提高蛋白基因组学的可靠性。得到的样品RNA序列与参考基因数据库SilkTransDB比对后发现1704个新转录本,1325个序列数>1000bp,1007个仅有一个外显子。除此之外,还在注释基因的内含子区发现11368个新外显子,在3482个基因模型中具有一定作用;在2153个基因中发现2581个UTRs。共鉴定2017个可变剪接,可分为四种不同类型:①1498为保留内含子,②514为跳跃外显子,③4个为5‘和3‘可变剪接位点,④一个为可变5’外显子。
3.7 蛋白基因组学和转录蛋白组学分析的比较
由蛋白基因组学鉴定的74个新ORFs中有39个可以从转录数据库中鉴定到,这39个外显子大多被注释为外显子,但两种方法的外显子鉴定界限不同。蛋白基因组学和转录组学对许多未发现基因具有更精确的注释。当前注释误差的主要来源为传统注释途径的不准确以及可变剪接的存在,发现的新基因可能是保留内含子,也可能是注释错误,很难区分。由蛋白基因组学鉴定的外显子结构和转录组学鉴定的外显子结构有很大的不同。由蛋白基因组学和转录组学共同鉴定1838个新转录本,1593个可变剪接;校正了5074个固有基因。
3.8 可变剪接和新转录本的GO和KEGG路径分析
用所有桑蚕基因作为背景,对1838个新转录本和1593个可变剪接基因做GO功能分类,1838个新转录本中仅350个在GO中被分类。在分子功能分类中,新转录本和全基因没有明显区别,值得注意的是,没有基因参与抗氧化过程,有一个新的转录本与蛋白辅助转导活性特异相关。大多数GO terms与细胞和代谢过程相关,Six GO terms 与多细胞有机体过程,生物调节,发育过程,色素沉着,细胞成分组织和位置相关。最终1593个可变剪接中有1074个被GO注释,大都包含于细胞,细胞器,结合,催化活性,细胞和代谢过程中。
KEGG的通路分析显示1024个可变剪接基因包含于285个KEGG通路中,46个可变剪接基因参与RNA传输通路,40个可变剪接基因参与于"剪接体通路"。共有40个可变剪接基因与内质蛋白处理有关,38个可变剪接基因参与核糖体,表明可变剪接基因的蛋白合成活性。
4、 结论
最终,共鉴定1838个新转录本,1593个可变剪接基因;使用蛋白基因组学和转录组学分析校正了5074个固有基因。
5、创新点
本文通过使用蛋白基因组学的方法对基因进行注释,扩大了鉴定范围并提高了注释准确度。
Journal of Proteome Research | Improving Silkworm Genome Annotation Using a Proteogenomics Approach (分享人:张霞)的更多相关文章
- Journal of Proteome Research | SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants for proteogenomic interpretation | SAAV的识别、功能注释和检索 | (解读人:徐洪凯)
文献名:SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants fo ...
- Journal of Proteome Research | Prediction of an Upper Limit for the Fraction of Interprotein Cross-Links in Large-Scale In Vivo Cross-Linking Studies (分享人:张宇星)
题目:Prediction of an Upper Limit for the Fraction of Interprotein Cross-Links in Large-Scale In Vivo ...
- Journal of Proteome Research | 人类牙槽骨蛋白的蛋白质组学和n端分析:改进的蛋白质提取方法和LysargiNase消化策略增加了蛋白质组的覆盖率和缺失蛋白的识别 | (解读人:卜繁宇)
文献名:Proteomic and N-Terminomic TAILS Analyses of Human Alveolar Bone Proteins: Improved Protein Extr ...
- Journal of Proteome Research | iHPDM: In Silico Human Proteome Digestion Map with Proteolytic Peptide Analysis and Graphical Visualizations(iHPDM: 人类蛋白质组理论酶解图谱的水解肽段分析和可视化展示)| (解读人:邓亚美)
文献名:iHPDM: In Silico Human Proteome Digestion Map with Proteolytic Peptide Analysis and Graphical Vi ...
- Journal of Proteome Research | Down-Regulation of a Male-Specific H3K4 Demethylase, KDM5D, Impairs Cardiomyocyte Differentiation (男性特有的H3K4脱甲基酶基因(KDM5D)下调会损伤心肌细胞分化) | (解读人:徐宁)
文献名:Down-Regulation of a Male-Specific H3K4 Demethylase, KDM5D, Impairs Cardiomyocyte Differentiatio ...
- Journal of Proteome Research | Quantitative Subcellular Proteomics of the Orbitofrontal Cortex of Schizophrenia Patients (精神分裂症病人眶额叶皮层亚细胞结构的定量蛋白质组学研究)(解读人:王聚)
期刊名:Journal of Proteome Research 发表时间:(2019年10月) IF:3.78 单位: 里约热内卢联邦大学 坎皮纳斯州立大学 坎皮纳斯州立大学神经生物学中心 卡拉博大 ...
- Journal of Proteome Research | Proteomic Profiling of Rhabdomyosarcoma-Derived Exosomes Yield Insights into Their Functional Role in Paracrine Signaling (解读人:孙国莹)
文献名:Proteomic Profiling of Rhabdomyosarcoma-Derived Exosomes Yield Insights into Their Functional Ro ...
- Journal of Proteome Research | Global Proteomic Analysis of Lysine Succinylation in Zebrafish (Danio rerio) (解读人:关姣)
文献名:Global Proteomic Analysis of Lysine Succinylation in Zebrafish (Danio rerio)(斑马鱼赖氨酸琥珀酰化的全球蛋白质组学分 ...
- Journal of Proteome Research | An automated ‘cells-to-peptides’ sample preparation workflow for high-throughput, quantitative proteomic assays of microbes (解读人:陈浩)
文献名:An automated ‘cells-to-peptides’ sample preparation workflow for high-throughput, quantitative p ...
随机推荐
- unittest(22)- p2p项目实战(3)-project_path
# 3. project_path.py # 用来读取文件的路径 import os # os.path.split(path)使用: # 1.path如果是具体到文件名,则返回最后层级的文件,和文件 ...
- python标准库:csv 模块
原文地址:http://www.bugingcode.com/blog/python_csv.html csv 模块被用来读取CSV格式(用逗号分割数值)的数据文件,CSV格式的文件经常在微软的Exc ...
- 添砖加瓦:[OpenCV]入门(一)
1.OpenCV安装 (1)下载: 本文采用的是源码的方式进行安装,源码可以从OpenCV官网下载.这里以3.4.1为例. (2)安装 这里下载到的文件为3.4.1.zip."unzip 3 ...
- 使用powerdesigner进行数据库设计
powerdesigner安装破解文件:链接:https://pan.baidu.com/s/1oKAdUqTKElQ9d86FV-SDTQ 密码:l4y5 基本操作参考:1.PowerDesigne ...
- 作为前端,你需要懂得javascript实现继承的方法
在ES6之前,javascript不跟其他语言一样,有直接继承的方法,它需要借助于构造函数+原型对象模拟实现继承.现在我们可以利用ES6的extends方法实现继承,如果想了解更多有关ES6实现的继承 ...
- Python——详解collections工具库
本文始发于个人公众号:TechFlow,原创不易,求个关注 今天为大家介绍Python当中一个很好用也是很基础的工具库,叫做collections. collection在英文当中有容器的意思,所以顾 ...
- Javascript学习笔记-基本概念-函数
ECMAScript 中的函数使用function 关键字来声明,后跟一组参数以及函数体.函数的基本语法如下所示: function functionName(arg0, arg1,...,argN) ...
- 6,HDFS HA
目录 HDFS HA 一.HA(High Availability)的使用原因 二.HA的同步 三.HA的自动容灾 HDFS HA 一.HA(High Availability)的使用原因 1.1 在 ...
- JavaScript实现栈结构(Stack)
JavaScript实现栈结构(Stack) 一.前言 1.1.什么是数据结构? 数据结构就是在计算机中,存储和组织数据的方式. 例如:图书管理,怎样摆放图书才能既能放很多书,也方便取? 主要需要考虑 ...
- Mysql8以上需要指定时区serverTimezone
JDBC连接Mysql8以下 com.mysql.jdbc.Driver url=jdbc:mysql://localhost:3306/test?useUnicode=true&charac ...
