HDU1560 DNA sequence(IDA*)题解
DNA sequence
For example, given "ACGT","ATGC","CGTT" and "CAGT", you can make a sequence in the following way. It is the shortest but may be not the only one.
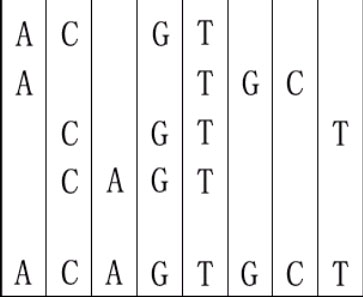
思路:
刚开始BFS就爆内存了。
新思路是给dfs加一个最小限制,超过限制就返回,然后不断加大限制直到符合,那么此时dfs的答案也是最小的。这里有几个要剪枝的地方:一是超过限制剪枝;二是预估值+当前值超过限制也要剪枝。
一开始看的那些题解都看不懂的我emmmm......orz
借鉴题解:链接
Code:
#include<cstdio>
#include<cstring>
#include<cstdlib>
#include<cctype>
#include<queue>
#include<cmath>
//#include<map>
#include<iostream>
#include<algorithm>
#define INF 0x3f3f3f3f
const int N=810;
using namespace std;
char str[10][10],dna[4]={'A','C','G','T'};
int n,deep,ans,len[10];
void dfs(int step,int pos[]){ //step为当前长度
if(step>deep) return; //超过深度返回
int maxdeep=0;
for(int i=0;i<n;i++){
maxdeep=max(len[i]-pos[i],maxdeep); //maxdeep为预估剩余深度
}
if(step+maxdeep>deep) return; //当前长度加预估剩余深度大于deep,剪枝
if(maxdeep==0){ //所有串都满足
ans=deep;
return;
}
int temp[10],flag;
for(int i=0;i<4;i++){
flag=0;
for(int j=0;j<n;j++){
if(str[j][pos[j]]==dna[i]){
temp[j]=pos[j]+1;
flag=1;
}
else temp[j]=pos[j];
}
if(flag){
dfs(step+1,temp);
}
if(ans) return;
}
}
int main(){
int t;
scanf("%d",&t);
while(t--){
deep=0;
scanf("%d",&n);
for(int i=0;i<n;i++){
scanf("%s",str[i]);
len[i]=strlen(str[i]);
deep=max(deep,len[i]); //找出最长的作为第一次深搜最小深度
}
ans=0;
int pos[10]; //表示第i组验证到第pos[i]个
memset(pos,0,sizeof(pos));
while(1){
dfs(0,pos);
if(ans) break;
deep++; //加深迭代深度,重新DFS
}
printf("%d\n",ans);
}
return 0;
}
HDU1560 DNA sequence(IDA*)题解的更多相关文章
- HDU1560 DNA sequence —— IDA*算法
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=1560 DNA sequence Time Limit: 15000/5000 MS (Java/Oth ...
- HDU1560 DNA sequence IDA* + 强力剪枝 [kuangbin带你飞]专题二
题意:给定一些DNA序列,求一个最短序列能够包含所有序列. 思路:记录第i个序列已经被匹配的长度p[i],以及第i序列的原始长度len[i].则有两个剪枝: 剪枝1:直接取最长待匹配长度.1900ms ...
- Hdu1560 DNA sequence(IDA*) 2017-01-20 18:53 50人阅读 评论(0) 收藏
DNA sequence Time Limit : 15000/5000ms (Java/Other) Memory Limit : 32768/32768K (Java/Other) Total ...
- HDU1560 DNA sequence
题目: The twenty-first century is a biology-technology developing century. We know that a gene is made ...
- HDU 1560 DNA sequence (IDA* 迭代加深 搜索)
题目地址:http://acm.hdu.edu.cn/showproblem.php?pid=1560 BFS题解:http://www.cnblogs.com/crazyapple/p/321810 ...
- HDU 1560 DNA sequence(IDA*)
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=1560 题目大意:给出n个字符串,让你找一个字符串使得这n个字符串都是它的子串,求最小长度. 解题思路: ...
- HDU 1560 DNA sequence(DNA序列)
HDU 1560 DNA sequence(DNA序列) Time Limit: 15000/5000 MS (Java/Others) Memory Limit: 32768/32768 K ...
- hdu 1560 DNA sequence(迭代加深搜索)
DNA sequence Time Limit : 15000/5000ms (Java/Other) Memory Limit : 32768/32768K (Java/Other) Total ...
- POJ2278 DNA Sequence —— AC自动机 + 矩阵优化
题目链接:https://vjudge.net/problem/POJ-2778 DNA Sequence Time Limit: 1000MS Memory Limit: 65536K Tota ...
随机推荐
- 一致性 hash 算法(转)
add by zhj:介绍了什么是一致性hash,以及实现一致性hash的一种算法. 原文:http://my.oschina.net/u/195065/blog/193614 目录[-] 一致性 h ...
- Loadrunner之脚本的思考时间(固定/随机)设置、调试、保存、测试服务器监控等(六)
一.思考时间的设置 1)设置固定思考时间: Action(){ // … your code lr_think_time(3); //固定设置此处思考时间3s // … more of your co ...
- (1.2)mysql 索引概念
索引的存储分类:mysql目前提供了以下4种索引 [1]B-Tree索引:最常见的索引类型,大部分引擎都支持B树索引 [2]HASH索引:只有Memory引擎支持,使用场景简单 [3]R-Tree索引 ...
- windows dos命令大全
windows dos命令大全 一.windows DOS命令概述 Windows的dos命令又是Windows的CMD命令.而cmd命令又是原来MS-DOS系统保留下来. DOS来源路径与配置对应的 ...
- 【剑指offer】输入一个链表,输出该链表中倒数第k个结点。
一.题目: 输入一个链表,输出该链表中倒数第k个结点. 二.思路: 用两个指针p1和p2,p2先跑k步,然后p1和p2同时跑,p2跑到头p1所在的位置就是倒数第k个节点.很简单吧?简单你也想不到,想到 ...
- phpQuery中文手册(更新中)
示例 phpQuery::newDocumentFileXHTML('my-xhtml.html')->find('p'); $ul = pq('ul'); 载入文档 phpQuery::new ...
- spring boot读取配置文件
一.springboot配置文件 核心配置文件和自定义配置文件.核心配置文件是指在resources根目录下的application.properties或application.yml配置文 ...
- [LintCode] 395. Coins in a Line 2_Medium tag: Dynamic Programming, 博弈
Description There are n coins with different value in a line. Two players take turns to take one or ...
- c/c++的预处理定义 Stringizing Operator (#) Charizing Operator (#@) Token-Pasting Operator (##)
c/c++的预处理定义:一.Stringizing Operator (#)在c和c++中数字标志符#被赋予了新的意义,即字符串化操作符.其作用是:将宏定义中的传入参数名转换成用一对双引号括起来参数名 ...
- mongodb mongotemplate聚合
1.group by并且计算总数 @Test public void insertTest() { //测试数据 //insertTestData(); Aggregation agg = Aggre ...
