使用 Docker 分析高通量测序数据
端午节假期,先祝各位 Bio IT 的爱好者们,节日快乐!
做生信的童鞋想要学习 Docker,或者使用 Docker+Pipeline 封装自己的一套数据分析流程,相信一定不能错过胡博强老师在2017年写这篇《[Docker]使用阿里云 + Docker 分析高通量测序数据——RNA-Seq 与 ChIP-Seq. - Boqiang Hu》教程,这个教程同时也以推文的方式发布在了 2017-03-21 生信技能树公众号上,感兴趣的同学可以自己去翻一下。
根据教程+tangEpiNGSInstall 仓库提供的原始测试数据,本人这两天测试跑了一下,发现了一点点小问题。
$ git clone https://github.com/shenweiyan/tangEpiNGSInstall.git
$ tree
.
└── tangEpiNGSInstall
├── Dockerfile
├── README.md
├── settings
│ ├── run_chipseq.py
│ ├── run_chipseq.sh
│ ├── run_mRNA.py
│ ├── run_mRNA.sh
│ ├── scripts_chipseq.py
│ └── scripts_mRNA.py
├── src
│ └── run_sample.sh
├── test_fq
│ ├── H3K4me3
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ ├── Input
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ └── sample.tab.xls
└── test_fq_RNA
├── SampleA1
│ ├── test.1.fastq.gz
│ └── test.2.fastq.gz
└── sample.tab.xls
8 directories, 17 files
$ mkdir -p results database_ChIP/mm10
$ chmod 777 results database_ChIP/mm10 # avoiding Permission issue
$ tree
.
├── database_ChIP
│ └── mm10
├── results
└── tangEpiNGSInstall
├── Dockerfile
├── README.md
├── settings
│ ├── run_chipseq.py
│ ├── run_chipseq.sh
│ ├── run_mRNA.py
│ ├── run_mRNA.sh
│ ├── scripts_chipseq.py
│ └── scripts_mRNA.py
├── src
│ └── run_sample.sh
├── test_fq
│ ├── H3K4me3
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ ├── Input
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ └── sample.tab.xls
└── test_fq_RNA
├── SampleA1
│ ├── test.1.fastq.gz
│ └── test.2.fastq.gz
└── sample.tab.xls
11 directories, 17 files
$ docker pull hubq/tanginstall:latest
$ docker run -v /data/docker/train/tangEpiNGSInstall/test_fq:/fastq -v /data/docker/train/results:/home/analyzer/project -v /data/docker/train/database_ChIP/mm10:/home/analyzer/database_ChIP/mm10 -v /data/docker/train/tangEpiNGSInstall/settings/:/settings/ --env ref=mm10 --env type=ChIP hubq/tanginstall:latest
INFO @ 2021-06-10 03:29:48,154: Begin checking input files.
INFO @ 2021-06-10 03:29:48,154: Input database files were all put in /home/analyzer/database_ChIP/mm10.
INFO @ 2021-06-10 03:29:48,154: Input fasta /home/analyzer/database_ChIP/mm10/mm10.fa not find. Now download from UCSC
INFO @ 2021-06-10 03:45:01,604: /home/analyzer/database_ChIP/mm10/mm10.fa generation done!
INFO @ 2021-06-10 03:45:01,605: Fasta were not indexed.
INFO @ 2021-06-10 03:45:02,105: Now build index using bwa.
INFO @ 2021-06-10 03:48:20,683: Building index done!
INFO @ 2021-06-10 03:48:20,683: Genome GTF file were not found.
INFO @ 2021-06-10 03:48:21,184: Now download refGene file from UCSC.
INFO @ 2021-06-10 05:03:38,768: Generate refGene done!
INFO @ 2021-06-10 05:03:38,768: RepeatMask file were not found.
INFO @ 2021-06-10 05:03:39,269: Now download rmsk file from UCSC.
INFO @ 2021-06-10 05:06:03,592: Generate RepeatMask done!
Traceback (most recent call last):
File "/home/analyzer/module/ChIP/run_chipseq.py", line 148, in <module>
main()
File "/home/analyzer/module/ChIP/run_chipseq.py", line 126, in main
samp_peak.get_idr_stat()
File "/home/analyzer/module/ChIP/frame/module02_call_peaks.py", line 244, in get_idr_stat
mod_Stat.IDR_Stat()
File "/home/analyzer/module/ChIP/frame/module00_StatInfo.py", line 113, in IDR_Stat
f_idr_out = open(file_idr_out,"w")
IOError: [Errno 2] No such file or directory: '/home/analyzer/project/ChIP_test/StatInfo/IDR_result./home/analyzer/project/ChIP_test/sample.tab.xls'
cp: cannot stat `03.2.Peak_mrg/*/*_treat_minus_control.sort.norm.bw': No such file or directory
cp: cannot stat `03.3.Peak_idr/*/*.conservative.regionPeak.gz*': No such file or directory
cp: cannot stat `StatInfo/*': No such file or directory

出于学习和折腾,针对这个问题,个人在
hubq/tanginstall:latest 的镜像基础上做了一点小调整,并重新打包成一个名为
shenweiyan/tanginstall:latest 的新镜像 push 到了 Docker Hub,抛砖引玉,供大家学习参考。
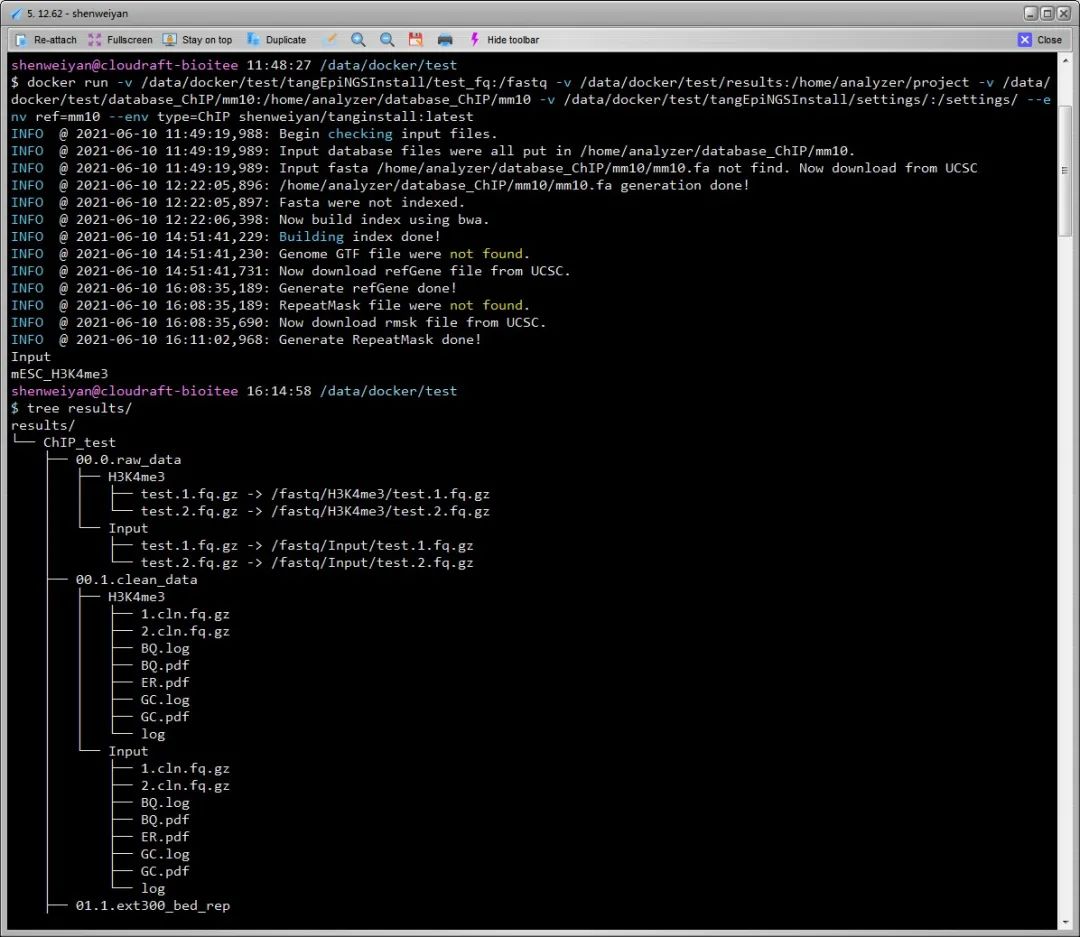
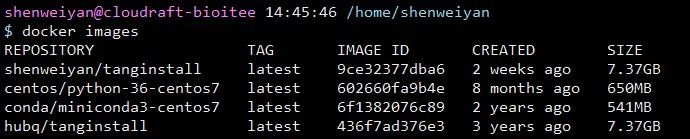
简单说一下这个镜像的几点细节。
整个镜像体积比较大,总共约 7.37GB,pull 下来可能比较慢。
- 如果没有 ref(hg19/hg38 or mm9/mm10),镜像执行过程中会首先执行下载,然后拆分合并,建立 index。
db01.DownloadRef.sh
$ cat db01.DownloadRef.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
dir_path=/home/analyzer/module/ChIP
cd $dir_database
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/bigZips/chromFa.tar.gz
tar -zxvf $dir_database/chromFa.tar.gz
for i in {1..22} X Y M
do
cat $dir_database/chr$i.fa
done >$dir_database/${ref}.fa && rm $dir_database/chr*fa
db02.RefIndex.sh
$ cat db02.RefIndex.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
bwa_exe=/software/install_packages/bwa-0.7.5a/bwa
samtools_exe=/software/install_packages/samtools-0.1.18/samtools
div_bins_exe=/home/analyzer/module/ChIP/bin/div_bins/bed_read
$samtools_exe faidx $dir_database/${ref}.fa
$bwa_exe index $dir_database/${ref}.fa
$dix_bins_exe -b 100 $dir_database/${ref}.fa.fai $dir_database/columns.100.bed
$dix_bins_exe -b 1000 $dir_database/${ref}.fa.fai $dir_database/columns.1kb.bed
cut -f 1-2 $dir_database/${ref}.fa.fai >$dir_database/${ref}.fa.len
db03.RefGene.sh
$ cat db03.RefGene.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
bedtools_exe=/software/install_packages/bedtools2/bin/bedtools
ucsc_dir=/software/install_packages/UCSC
bin=/home/analyzer/module/ChIP/bin
dir_path=/home/analyzer/module/ChIP
cd $dir_database
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/refGene.txt.gz
### remove chromosome fragments(unassembled).
for i in {1..22} X Y M
do
zcat $dir_database/refGene.txt.gz | grep -w chr$i
done >$dir_database/tmp
mv $dir_database/tmp $dir_database/refGene.txt
# refGene.bed
cat $dir_database/refGene.txt |\
awk '{
tag="noncoding";
if($4~/^NM/){tag="protein_coding"};
OFS="\t";
print $3,$5,$6,$2,$4,$10,$11,tag,$13
}' /dev/stdin |\
python $bin/s03_genePred2bed.py /dev/stdin |\
$bedtools_exe sort -i /dev/stdin >$dir_database/refGene.bed &&\
# region.Intragenic.bed
# For novo lncRNA detection
$bin/find_ExonIntronIntergenic/find_ExonIntronIntergenic \
$dir_database/refGene.bed \
$dir_database/${ref}.fa.fai >$dir_database/pos.bed &&\
grep -v "Intergenic" $dir_database/pos.bed |\
awk '{OFS=" ";print $1,$2,$3,"Intragenic"}' /dev/stdin \
>$dir_database/region.Intragenic.bed &&\
# refGene.gtf
# For mapping
zcat $dir_database/refGene.txt.gz |\
cut -f 2- |\
$ucsc_dir/genePredToGtf file stdin /dev/stdout |\
grep -w exon |\
$bedtools_exe sort -i /dev/stdin >$dir_database/refGene.gtf &&\
cat $dir_path/database/ERCC.gtf >>$dir_database/refGene.gtf
db04.rmsk.sh
$ cat db04.rmsk.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
bedtools_exe=/software/install_packages/bedtools2/bin/bedtools
ucsc_dir=/software/install_packages/UCSC
bin=/home/analyzer/module/ChIP/bin
dir_path=/home/analyzer/module/ChIP
cd $dir_database
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/rmsk.txt.gz
zcat $dir_database/rmsk.txt.gz |\
awk '{
OFS="\t";
print $6,$7,$8,$2,".",".",".","("$9")",$10,$11,$12 "/" $13,$14,$15,$16,$17
}' /dev/stdin |\
tail -n +2 /dev/stdin >$dir_database/chrom.bed
for i in {1..22} X Y M
do
grep -w chr$i $dir_database/chrom.bed
done >$dir_database/tmp
mv $dir_database/tmp $dir_database/chrom.bed
$bedtools_exe sort -i $dir_database/chrom.bed >$dir_database/chrom.sort.bed
为节省下载时间,建议事先准备好 ${ref}.fa,如果没有,也可以先下载好以下文件。
# db01.DownloadRef.sh:
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/bigZips/chromFa.tar.gz
# db03.RefGene.sh:
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/refGene.txt.gz
#db04.rmsk.sh:
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/rmsk.txt.gz
bwa index(db02.RefIndex.sh)非常耗时,个人一个4核16G配置的服务器也跑了大约2.5小时。


生信服务器 | 更改 CentOS/RHEL 6/7 中的时区





本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
使用 Docker 分析高通量测序数据的更多相关文章
- NGS基础 - 高通量测序原理
NGS基础 - 高通量测序原理 原创: 赑屃 生信宝典 2017-07-23 NGS系列文章包括NGS基础.转录组分析.ChIP-seq分析.DNA甲基化分析.重测序分析五部分内容. NGS基础系列文 ...
- 【转录组入门】3:了解fastq测序数据
操作:需要用安装好的sratoolkit把sra文件转换为fastq格式的测序文件,并且用fastqc软件测试测序文件的质量 作业:理解测序reads,GC含量,质量值,接头,index,fastqc ...
- fastx_toolkit去除测序数据中的接头和低质量的reads
高通量测序数据下机后得到了fastq的raw_data,通常测序公司在将数据返还给客户之前会做"clean"处理,即得到clean_data.然而,这些clean_data是否真的 ...
- Next generation sequencing (NGS)二代测序数据预处理与分析
二代测序原理: 1.DNA待测文库构建. 超声波把DNA打断成小片段,一般200--500bp,两端加上不同的接头2.Flowcell.一个flowcell,8个channel,很多接头3.桥式PCR ...
- 单细胞转录组测序数据的可变剪接(alternative splicing)分析方法总结
可变剪接(alternative splicing),在真核生物中是一种非常基本的生物学事件.即基因转录后,先产生初始RNA或称作RNA前体,然后再通过可变剪接方式,选择性的把不同的外显子进行重连,从 ...
- linux驱动由浅入深系列:高通sensor架构实例分析之三(adsp上报数据详解、校准流程详解)【转】
本文转载自:https://blog.csdn.net/radianceblau/article/details/76180915 本系列导航: linux驱动由浅入深系列:高通sensor架构实例分 ...
- GEO(Gene Expression Omnibus):高通量基因表达数据库
Gene Expression Omnibus(GEO)是一个公共存储库,可以存档和自由分发由科学界提交的全套微阵列,新一代测序和其他形式的高通量功能基因组数据. 除数据存储外,还提供一系列基于Web ...
- 测序数据质控-FastQC
通常我们下机得到的数据是raw reads,但是公司通常会质控一份给我们,所以到很多人手上就是clean data了.我们再次使用fastqc来进行测序数据质量查看以及结果分析. fastqc的操作: ...
- 弗雷塞斯 从生物学到生物信息学到机器学习 转录组入门(3):了解fastq测序数据
sra文件转换为fastq格式 1 fastq-dump -h --split-3 也就是说如果SRA文件中只有一个文件,那么这个参数就会被忽略.如果原文件中有两个文件,那么它就会把成对的文件按*_1 ...
- 转录组入门(3):了解fastq测序数据
sra文件转换为fastq格式 fastq-dump -h --split-3 也就是说如果SRA文件中只有一个文件,那么这个参数就会被忽略.如果原文件中有两个文件,那么它就会把成对的文件按*_1.f ...
随机推荐
- Django笔记五之字段类型
这篇笔记介绍字段的类型 Field Type. Django 的model 下的 field 对应的是 MySQL 中的表字段,而我们定义的 field 的类型则对应 MySQL 中的字段类型. 本次 ...
- Spring Data Solr 对 Solr 的增删改查实例
Spring Data Solr 就是为了方便 solr 的开发研制的一个框架,其底层是对 SolrJ(官方 API)的封装 一.环境搭建 第一步:创建 Maven 项目(springdatasolr ...
- mysql根据多列去除重复数据bing 并保留id最小的一项
首先我的数剧是这样的: 我想根据name1.relation和name2判断如果重复的话删除重复的数据且保留id最小的一条数据,于是写了下面的sql语句 DELETE FROM star_relati ...
- go微服务框架kratos学习笔记二(kratos demo 结构)
目录 api cmd configs dao di model server service 上篇文章go微服务框架kratos学习笔记一(kratos demo)跑了kratos demo 本章来看 ...
- [网络/Java EE/Web]Tomcat/Nginx中配置全局的安全响应头(header)——X-Frame-Options / X-XSS-Protection / X-Content-Options
Step1 配置Tomcat step1.1 查看是否已配置目标的HTTP网络安全头 方式1 – Tomcat / conf/web.xml cat /opt/myTomcat/conf/web.xm ...
- [Linux]常用命令之【cat/echo/iconv/vi/grep/find/head/tail】
cat cat [选项] [文件].. # 一次显示整个文件或从键盘创建一个文件或将几个文件合并成一个文件 cat -n file1 # 编号文件内容再输出 echo -n 输出后不换行 -e 遇到转 ...
- React+Antd在使用form表单提交DatePicker日期框的时候会出现少八小时的情况
在使用antd做form表单提交的时候,突然发现了一个很有意思的bug.就是在使用datepicker组件日期框的时候会出现提交后少一天的问题 我在网上搜索了许多解决办法,也是困扰了我一天的时间,下面 ...
- day17:内置方法&math模块&random模块&pickle模块
内置方法 1.round:四舍五入 正常遵守四舍五入,但在n.5结构中,n为偶数则舍去,n为奇数则进一. res1 = round(4.51) # 5 res2 = round(4.5) # 4 re ...
- Proxy 与 Object.defineProperty 优劣对比?
Proxy的优势如下 1.Proxy 可以直接监听对象而不是属性(Object.defineProperty一次只能监视一个属性,如果要监视一个对象,那么需要遍历这个对象),可以直接监听数组的变化(O ...
- springCloud项目搭建版本选择
1.查看spring cloud的版本 https://spring.io/projects/spring-cloud#learn 选择spring boot版本 https://mvnreposit ...
