bam文件softclip , hardclip ,markduplicate的探究
测序产生的bam文件,有一些reads在cigar值里显示存在softclip,有一些存在hardclip,究竟softclip和hardclip是怎么判断出来的,还有是怎么标记duplicate的reads的,我怀着这些问题进行了探究。
测试步骤
- 编辑两个bed文件,分别含有我们需要的read1和read2位置,这里每个文件包含两条read1或者两条read2,read1、read2一对作为原始的reads(序列名primer_pri),另一对作为截取的材料(这里取序列名为other)
- 使用bedtools getfasta,从参考基因组获得reads的序列,将read2反向互补。将原始reads放入两个文件,一个test_R1.fa,一个test_R2.fa
在test_R1.fa中添加其它修改过的原始reads,并在test_R2.fa中也添加相应的read2,不过read2不修改
read1名称如下

- primer_pri:原始read
- pimer_duplicate1:primer_pri的重复,一模一样
- pimer_duplicate2:read1 primer_pri去掉5‘两个碱基
- pimer_duplicate3:read1 primer_pri去掉5’两个碱基,再去掉3'两个碱基
- pimer_changeR2Termi5base:read1修改了read2 5‘端的碱基
- primer_halfother:read1截掉后面reads,用other的5‘部分reads补全
- pimer_change3Termi5base_change5Termi5base_sametwo:read1和read2一样,并且5'端和3‘端都改变了5个碱基
pimer_change3Termi5base_change5Termi5base:read1 5'端和3‘端都改变了5个碱基,但是read2保留primer_pri的read2
结果
softclip和hardclip
其中
- primer_halfother read1 82M65S,有SA tag,SA:Z:chr12,5378700,+,79S68M,60,0
- pimer_change3Termi5base_change5Termi5base_sametwo 两条reads均为5S137M5S
- pimer_change3Termi5base_change5Termi5base read1 5S137M5S
- primer_halfother read1 79H68M ,有SA tag,SA:Z:chr12,5378502,+,82M65S,60,0
结论(部分分析参考SAMv1.pdf文件)
- 对于map到一个位置的read,两端map不上的叫做clip ,map到一个位置的情况下以softclip显示(比如 pimer_change3Termi5base_change5Termi5base_sametwo和 pimer_change3Termi5base_change5Termi5base read1)
- 对于嵌合比对的read(可以map到多个区域,并且这比对上的区域很大部分非overlap),比如primer_halfother read1比对上两个位置,一个比对到chr12 : 5378502,一个比对到chr12:5378700,并且两次hit的位置的碱基overlap少,产生的这种情况是因为read前一部分比对到了前者,而后一部分又可以比对到了后者,因此无论比对到任何一个位置都这条read都是部分match(这种叫做Chimeric alignment/嵌合比对)。
- 嵌合比对的read,有一条是最优的read,因为我们map的时候设置了-M参数,因此认为较短的split的reads断定为优,这里是的62 clip 的hit断定为优。因此65个比对不上的显示为softclip,而另外一个hit,79 clip显示为hard clip,序列中不显示,并且存入0x800(supplementary alignment flag)
- 为什么82M65S对应的是79H68M呢,理论上应该是82H65M才对,这是因为这里两个比对有三个碱基的overlap,所以前面有65+3个match,后面有79+3个match(制造reads的时候碰巧截取的primer read 3'端三个碱基和截取的other read 5‘部分read 三个碱基一样)
- 这种嵌合比对的reads含有SA tag
duplicate
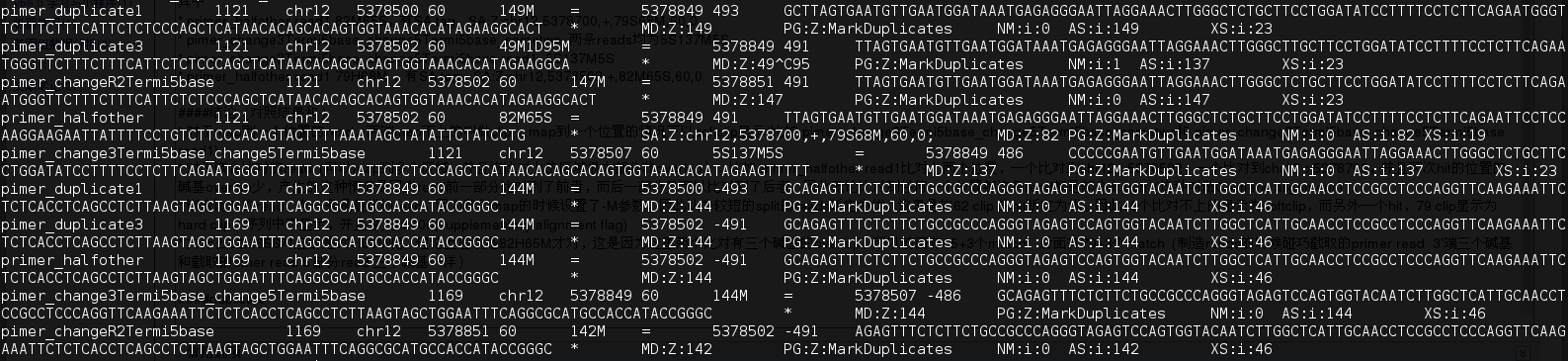
其中mark为duplicate 的reads 对(duplicate 是按fragment算) 有 primer duplicate1,primer_duplicate3,pimer_changeR2Termi5base,primer_halfother(82M65S,144M(未改的read2)),pimer_change3Termi5base_change5Termi5base
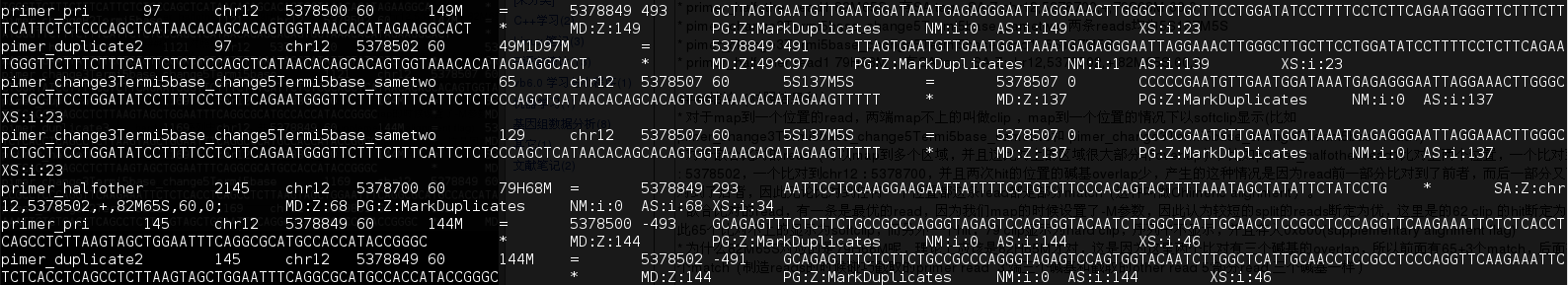
不属于duplicate的有
primer_pri,pimer_duplicate2,primer_halfother(79H68M,一条),pimer_change3Termi5basechange5Termi5base_sametwo
结论
- fragment的start和end一样(read1和read2(因为read2是测对链的,reads的5‘端都是fragment的末端)的5’的位置都相同)判断为duplicate,只取最优的不标记为duplicate
- primer_pri的duplicate是 primer duplicate1, primer_halfother
- pimer_duplicate2的duplicate是primer_duplicate3,pimer_changeR2Termi5base,pimer_change3Termi5base_change5Termi5base
- 没有duplicate的是primer_halfother(79H68M,一条),pimer_change3Termi5basechange5Termi5base_sametwo
- pimer_change3Termi5basechange5Termi5base_sametwo 5'有5 softclip,map的位置从M的碱基算起(见图),所以它没有duplicate
bam文件softclip , hardclip ,markduplicate的探究的更多相关文章
- SAMTOOLS使用 SAM BAM文件处理
[怪毛匠子 整理] samtools学习及使用范例,以及官方文档详解 #第一步:把sam文件转换成bam文件,我们得到map.bam文件 system"samtools view -bS m ...
- SAM/BAM文件处理
当测序得到的fastq文件map到基因组之后,我们通常会得到一个sam或者bam为扩展名的文件.SAM的全称是sequence alignment/map format.而BAM就是SAM的二进制文件 ...
- C++使用htslib库读入和写出bam文件
有时候我们需要使用C++处理bam文件,比如取出read1或者read2等符合特定条件的序列,根据cigar值对序列指定位置的碱基进行统计或者对序列进行处理并输出等,这时我们可以使用htslib库 ...
- Vivado约束文件(XDC)的探究(2)
Vivado约束文件(XDC)的探究(2)
- Vivado约束文件(XDC)的探究(1)
Vivado约束文件(XDC)的探究(1) 工程建好之后会出现xdc文件: 注意:active 和 target 生成的约束文件如下:
- bam文件测序深度统计-bamdst
最近接触的数据都是靶向测序,或者全外测序的数据.对数据的覆盖深度及靶向捕获效率的评估成为了数据质量监控中必不可少的一环. 以前都是用samtools depth 算出单碱基的深度后,用perl来进行深 ...
- 文件格式——Sam&bam文件
Sam&bam文件 SAM是一种序列比对格式标准, 由sanger制定,是以TAB为分割符的文本格式.主要应用于测序序列mapping到基因组上的结果表示,当然也可以表示任意的多重比对结果.当 ...
- Pysam 处理bam文件
Pysam可用来处理bam文件 安装: 用 pip 或者 conda即可 使用: Pysam的函数有很多,主要的读取函数有: AlignmentFile:读取BAM/CRAM/SAM文件 Varian ...
- 推荐一个SAM文件或者bam文件中flag含义解释工具
SAM是Sequence Alignment/Map 的缩写.像bwa等软件序列比对结果都会输出这样的文件.samtools网站上有专门的文档介绍SAM文件.具体地址:http://samtools. ...
随机推荐
- Laravel 日志查看器 导入log-viewer扩展
1.修改配置文件 config\app.php中 'log'=>'daily' 日志文件是按天生成的 2.在项目目录中composer命令安装扩展:composer require arcan ...
- struts2.0的工作原理?
struts2并不是一个陌生的web框架,它是以Webwork的设计思想为核心,吸收struts1的优点,可以说 struts2是struts1和Webwork结合的产物. struts2 的工作原理 ...
- (转)完整java开发中JDBC连接数据库代码和步骤
JDBC连接数据库 •创建一个以JDBC连接数据库的程序,包含7个步骤: 1.加载JDBC驱动程序: 在连接数据库之前,首先要加载想要连接的数据库的驱动到JVM(Java虚拟机), 这通过java.l ...
- 【JAVASCRIPT】React学习-巧用 props 的 children 属性实现内容填充
背景 平常写组件,经常遇到需要获取内容放入组件内部的情形. 实现方法 我们有两种实现方式 1. 自定义 props render 的时候通过获取 this.props.content 填充到组件内部 ...
- 【CSS】less 学习小结
1. less 使用 less 可直接使用浏览器解析 or 使用node 的grunt/gulp 解析成传统css . 推荐开发环境直接使用less 文件调试, 生产环境部署解析好的css 2. l ...
- Ubuntu中使用iptables
(一) 设置开机启动iptables # sysv-rc-conf --level 2345 iptables on (二) iptables的基本命令 1. 列出当前iptables的策略和规则 # ...
- Android学习笔记-ImageView(图像视图)
本节引言: 本节介绍的UI基础控件是:ImageView(图像视图),见名知意,就是用来显示图像的一个View或者说控件! 官方API:ImageView;本节讲解的内容如下: ImageView的s ...
- 深入浅出数据结构C语言班(11)——简要介绍算法时间复杂度
在接下来的数据结构博文中,我们将会开始接触到一些算法,也就是"解决某个问题的方法",而解决同一个问题总是会存在不同的算法,所以我们需要在不同的算法之中做出抉择,而做出抉择的根据往往 ...
- java笔录---反射机制(1)
引言 为了方便记忆java的反射机制,在这里仔细的总结了一下.主要是怕以后忘记了,这样也方便回忆.因为最近利用空余时间深入的了解spring和Mybatis框架, 像spring中核心模块IO ...
- jvm系列 (二) ---垃圾收集器与内存分配策略
垃圾收集器与内存分配策略 前言:本文基于<深入java虚拟机>再加上个人的理解以及其他相关资料,对内容进行整理浓缩总结.本文中的图来自网络,感谢图的作者.如果有不正确的地方,欢迎指出. 目 ...
