edgeR使用学习【转载】
转自:http://yangl.net/2016/09/27/edger_usage/
1.Quick start

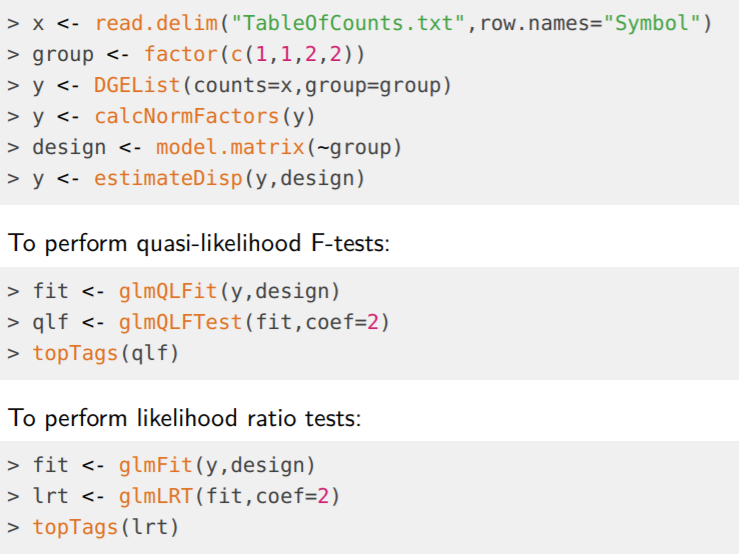
2. 利用edgeR分析RNA-seq鉴别差异表达基因:
#加载软件包
library("edgeR",verbose=0); # 1. 载入数据 读取read count数
data <- read.delim("pnas_expression.txt", row.names=1, stringsAsFactors=FALSE);
head(data); #输出
# lane1 lane2 lane3 lane4 lane5 lane6 lane8 len
# ENSG00000215696 0 0 0 0 0 0 0 330
# ENSG00000215700 0 0 0 0 0 0 0 2370
# ENSG00000215699 0 0 0 0 0 0 0 1842
# ENSG00000215784 0 0 0 0 0 0 0 2393
# ENSG00000212914 0 0 0 0 0 0 0 384
# ENSG00000212042 0 0 0 0 0 0 0 92 dim(data);
# [1] 37435 8 #2. 构建分组变量
#分为 Control组和DHT组 分别为4个和3个重复
targets <- data.frame(Lane = c(1:6,8), Treatment = c(rep("Control",4),rep("DHT",3)),
Label = c(paste("Con", 1:4, sep=""), paste("DHT", 1:3, sep=""))); targets
#输出
# Lane Treatement Label
# 1 1 Control Con1
# 2 2 Control Con2
# 3 3 Control Con3
# 4 4 Control Con4
# 5 5 DHT DHT1
# 6 6 DHT DHT2
# 7 8 DHT DHT3 #3. 创建基因表达列表 进行标准化因子计算
y <- DGEList(counts=data[,1:7], group=targets$Treatment, genes=data.frame(Length=data[,8]));
colnames(y) <- targets$Label;
dim(y);
# [1] 37435 7 #过滤表达量偏低的基因 !!!
#基因在至少3个样本中得count per million(cpm)要大于1
keep <- rowSums(cpm(y)>1) >= 3;
y <- y[keep,];
dim(y)
# [1] 16494 7
#重新计算库大小
y$samples$lib.size <- colSums(y$counts); #3. 进行标准化因子计算 默认使用TMM方法
y <- calcNormFactors(y);
y #输出
# An object of class "DGEList"
# $counts
# Con1 Con2 Con3 Con4 DHT1 DHT2 DHT3
# ENSG00000124208 478 619 628 744 483 716 240
# ENSG00000182463 27 20 27 26 48 55 24
# ENSG00000124201 180 218 293 275 373 301 88
# ENSG00000124207 76 80 85 97 80 81 37
# ENSG00000125835 132 200 200 228 280 204 52
# 16489 more rows ...
#
# $samples
# group lib.size norm.factors
# Con1 1 976847 1.0296636
# Con2 1 1154746 1.0372521
# Con3 1 1439393 1.0362662
# Con4 1 1482652 1.0378383
# DHT1 1 1820628 0.9537095
# DHT2 1 1831553 0.9525624
# DHT3 1 680798 0.9583181
#
# $genes
# [1] 2131 5453 4060 3264 945
# 16489 more rows ... #这里主要是通过图形的方式来展示实验组与对照组样本是否能明显的分开
#以及同一组内样本是否能聚的比较近 即重复样本是否具有一致性
plotMDS(y); #4. 估计离散度
y <- estimateCommonDisp(y, verbose=TRUE)
# Disp = 0.02002 , BCV = 0.1415
y <- estimateTagwiseDisp(y); plotBCV(y); #5. 通过检验来鉴别差异表达基因
et <- exactTest(y);
top <- topTags(et);
top #输出
# Comparison of groups: DHT-Control
# Length logFC logCPM PValue FDR
# ENSG00000151503 5605 5.816156 9.716866 0.000000e+00 0.000000e+00
# ENSG00000096060 4093 5.004454 9.950606 0.000000e+00 0.000000e+00
# ENSG00000166451 1556 4.683425 8.850401 2.297717e-249 1.263285e-245
# ENSG00000127954 3919 8.120955 7.216393 1.534440e-229 6.327264e-226
# ENSG00000162772 1377 3.316701 9.743797 7.975243e-216 2.630873e-212
# ENSG00000115648 2920 2.598440 11.474677 6.984860e-180 1.920138e-176
# ENSG00000116133 4286 3.244446 8.791930 1.290432e-174 3.040627e-171
# ENSG00000113594 10078 4.111120 8.055613 3.115276e-161 6.422921e-158
# ENSG00000130066 868 2.609899 9.989778 6.009018e-155 1.101253e-151
# ENSG00000116285 3076 4.201846 7.361640 6.299060e-149 1.038967e-145 #6. 定义差异表达基因与基本统计
summary(de <- decideTestsDGE(et)); # 默认选取FDR = 0.05为阈值 #输出
# [,1]
# -1 2094 #显著下调
# 0 12060 #没有显著差异
# 1 2340 #显著上调 #图形展示检验结果
detags <- rownames(y)[as.logical(de)];
plotSmear(et, de.tags=detags);
abline(h=c(-1, 1), col="blue");
//这个是分为 Control组和DHT组,检验这两组的差异表达基因。
//中间又一步是去除表达量过低的基因。
- 读取read count数
- 构建分组变量
- 创建基因表达列表 进行标准化因子计算 ,过滤表达量偏低的基因,进行标准化因子计算 默认使用TMM方法
- 估计离散度
- 通过检验来鉴别差异表达基因
- 定义差异表达基因与基本统计
edgeR使用学习【转载】的更多相关文章
- Java多线程学习(转载)
Java多线程学习(转载) 时间:2015-03-14 13:53:14 阅读:137413 评论:4 收藏:3 [点我收藏+] 转载 :http://blog ...
- Windows Services 学习(转载)
转载:http://blog.csdn.net/fakine/article/details/42107571 一.学习点滴 1.本机服务查看:services.msc /s2.服务手动安装(使用sc ...
- 【学习转载】MyBatis源码解析——日志记录
声明:转载自前辈:开心的鱼a1 一 .概述 MyBatis没有提供日志的实现类,需要接入第三方的日志组件,但第三方日志组件都有各自的Log级别,且各不相同,但MyBatis统一提供了trace.deb ...
- JVM的相关知识整理和学习--(转载)
JVM是虚拟机,也是一种规范,他遵循着冯·诺依曼体系结构的设计原理.冯·诺依曼体系结构中,指出计算机处理的数据和指令都是二进制数,采用存储程序方式不加区分的存储在同一个存储器里,并且顺序执行,指令由操 ...
- Jqgrid学习(转载)
jqGrid API 全 JQGrid是一个在jquery基础上做的一个表格控件,以ajax的方式和服务器端通信. JQGrid Demo 是一个在线的演示项目.在这里,可以知道jqgrid可以做 ...
- R中双表操作学习[转载]
转自:https://www.jianshu.com/p/a7af4f6e50c3 1.原始数据 以上是原有的一个,再生成一个新的: > gene_exp_tidy2 <- data.fr ...
- Java核心编程快速学习(转载)
http://www.cnblogs.com/wanliwang01/p/java_core.html Java核心编程部分的基础学习内容就不一一介绍了,本文的重点是JAVA中相对复杂的一些概念,主体 ...
- FPGA/SOPC学习转载
转自小時不識月http://www.cnblogs.com/yuphone/archive/2010/08/27/docs_plan.html 新网址为:http://andrewz.cn [连载计划 ...
- OpenGL入门学习(转载)
说起编程作图,大概还有很多人想起TC的#include <graphics.h>吧? 但是各位是否想过,那些画面绚丽的PC游戏是如何编写出来的?就靠TC那可怜的640*480分辨率.16色 ...
随机推荐
- 【转载】经典.net面试题目【为了笔试。。。。。】
. 简述 private. protected. public. internal 修饰符的访问权限. 答 . private : 私有成员, 在类的内部才可以访问. protected : 保护成员 ...
- Linux(Ubuntu)下也能用搜狗输入法了!!!
Ubuntu原生的中文输入法是不是总有点别扭? 不用再别扭了. 告诉你一个好消息:Linux(Ubuntu)下也能用搜狗输入法了!!! 下载地址:http://pinyin.sogou.com/lin ...
- spring整合websocket通信
1. maven依赖 <?xml version="1.0" encoding="UTF-8"?> <project xmlns=" ...
- Storm启动流程分析
1. 客户端运行storm nimbus时,会调用storm的python脚本,该脚本中为每个命令编写一个方法,每个方法都可以生成一条相应的java命令. 命令格式如下:java -server xx ...
- oAuth 认证和授权原理
什么是OAuth授权? 一.什么是OAuth协议 OAuth(开放授权)是一个开放标准. 允许第三方网站在用户授权的前提下访问在用户在服务商那里存储的各种信息. 而这种授权无需将用户提供用户名和密 ...
- 应用程序创建自己的奔溃转储(crash dump)文件
1.注册自定义的UnhandledExceptionFilter,C/C++ Runtime Library下需要注意自定义handler被移除(hook kernel32.dll的SetUnhand ...
- Ios8 Xcode6 设置Launch Image 启动图片
http://blog.sina.com.cn/s/blog_6c97abf10102voui.html Http://Www.woowen.com/Swift/2014/12/12/Ios8设置La ...
- 7.22 python面试题
2018-7-22 16:32:24 把面试题敲完了,,好强悍! Python 10期考试题 1.常用字符串格式化有那些?并说明他们的区别 # format 直接调用函数 # %s 语法塘 # %r ...
- 计蒜客 30994 - AC Challenge - [状压DP][2018ICPC南京网络预赛E题]
题目链接:https://nanti.jisuanke.com/t/30994 样例输入: 5 5 6 0 4 5 1 1 3 4 1 2 2 3 1 3 1 2 1 4 样例输出: 55 样例输入: ...
- ZOJ 2760 - How Many Shortest Path - [spfa最短路][最大流建图]
人老了就比较懒,故意挑了到看起来很和蔼的题目做,然后套个spfa和dinic的模板WA了5发,人老了,可能不适合这种刺激的竞技运动了…… 题目链接:http://acm.zju.edu.cn/onli ...
