DNA sequence HDU - 1560
DNA sequence
Time Limit: 15000/5000 MS (Java/Others) Memory Limit: 32768/32768 K (Java/Others)
Total Submission(s): 4217 Accepted Submission(s): 2020
For example, given "ACGT","ATGC","CGTT" and "CAGT", you can make a sequence in the following way. It is the shortest but may be not the only one.
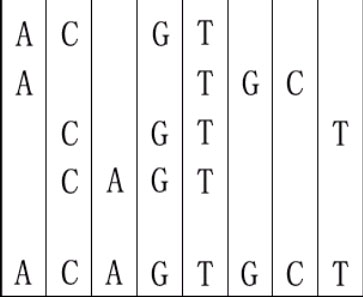
4
ACGT
ATGC
CGTT
CAGT
#include<iostream>
#include<cstdio>
#include<cstdlib>
#include<sstream>
#include<cstring>
#include<string>
#include<vector>
#include<set>
#include<stack>
#include<queue>
#include<map>
#include<cmath>
#include<algorithm>
using namespace std;
#define inf 0x3f3f3f3f
#define ll long long
#define MAX_N 1000005
#define gcd(a,b) __gcd(a,b)
#define mem(a,x) memset(a,x,sizeof(a))
#define mid(a,b) a+b/2
#define stol(a) atoi(a.c_str())//string to long
string temp[];
string c = "ACGT";
int pos[];
int len[];
int deep;
int n;
int ans(){
int maxn = ;
for(int i = ; i < n; i++){
maxn = max(maxn,len[i]-pos[i]);
}
return maxn;
}
int dfs(int step){
if(step + ans() > deep)
return ;
if(!ans())//满足构造序列包含所有序列
return ;
int temp1[];
for(int i = ; i < ; i++){ int flag = ;
for(int j = ; j < n; j++)
temp1[j] = pos[j]; for(int j = ; j < n; j++){
if(temp[j][pos[j]] == c[i]){
flag = ;
pos[j]++;
}
}
if(flag){
if(dfs(step+))
return ;
for(int j = ; j < n; j++)
pos[j] = temp1[j];
}
}
return ;
}
int main(){
// freopen("D:\\in.txt","r",stdin);
int T;
scanf("%d",&T);
while(T--){
scanf("%d",&n);
deep = ;
for(int i = ; i < n; i++){
cin>>temp[i];
len[i] = temp[i].length();
pos[i] = ;
deep = max(deep,len[i]);
}
while(){
if(dfs())
break;
++deep;
}
printf("%d\n",deep);
}
return ;
}
DNA sequence HDU - 1560的更多相关文章
- DNA sequence HDU - 1560(IDA*,迭代加深搜索)
题目大意:有n个DNA序列,构造一个新的序列,使得这n个DNA序列都是它的子序列,然后输出最小长度. 题解:第一次接触IDA*算法,感觉~~好暴力!!思路:维护一个数组pos[i],表示第i个串该匹配 ...
- IDA*、剪枝、较难搜索、扫描——DNA sequence HDU - 1560
万恶之源 翻译 题意就是给出N个DNA序列,要求出一个包含这n个序列的最短序列是多长 这是一道搜索题,为什么呢?从样例可以感受到,我们应该从左往右"扫描",从n个DNA序列中取出某 ...
- G - DNA sequence HDU - 1560
题目链接: https://vjudge.net/contest/254151#problem/G AC代码: #include<iostream> #include<cstring ...
- hdu 1560 DNA sequence(搜索)
http://acm.hdu.edu.cn/showproblem.php?pid=1560 DNA sequence Time Limit: 15000/5000 MS (Java/Others) ...
- HDU 1560 DNA sequence(DNA序列)
HDU 1560 DNA sequence(DNA序列) Time Limit: 15000/5000 MS (Java/Others) Memory Limit: 32768/32768 K ...
- hdu 1560 DNA sequence(迭代加深搜索)
DNA sequence Time Limit : 15000/5000ms (Java/Other) Memory Limit : 32768/32768K (Java/Other) Total ...
- 【HDU - 1560】DNA sequence (dfs+回溯)
DNA sequence 直接中文了 题目描述 21世纪是生物科技飞速发展的时代.我们都知道基因是由DNA组成的,而DNA的基本组成单位是A,C,G,T.在现代生物分子计算中,如何找到DNA之间的最长 ...
- HDU1560 DNA sequence —— IDA*算法
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=1560 DNA sequence Time Limit: 15000/5000 MS (Java/Oth ...
- POJ 2778 DNA Sequence(AC自动机+矩阵加速)
DNA Sequence Time Limit: 1000MS Memory Limit: 65536K Total Submissions: 9899 Accepted: 3717 Desc ...
随机推荐
- python学习笔记(19)-切片
转自https://www.jianshu.com/p/15715d6f4dad 在利用python解决各种实际问题的过程中,经常会遇到从某个对象中抽取部分值的情况,切片操作正是专门用于完成这一操作的 ...
- Redhat6更改yum源 (转)
最近虚拟机中安装了redhat6.3企业版,自带的yum用不起来,软件都找不到. 网上搜了一下说是没付钱...,需要改下yum源.操作步骤如下: 1.切换到yum源存放目录[root@rhel6 ~] ...
- 《C 程序设计语言》练习1-4
#include<stdio.h> /*当celsius=0,1,...,100时,打印摄氏温度与华氏温度对照表; 浮点数版本*/ main () { float fahr,celsius ...
- AttributeError: 'bytes' object has no attribute 'hex'
python3.5之前bytes数据没有hex()属性 需要使用 ''.join(map(lambda x:('' if len(hex(x))>=4 else '/x0')+hex(x)[2: ...
- [LC] 103. Binary Tree Zigzag Level Order Traversal
Given a binary tree, return the zigzag level order traversal of its nodes' values. (ie, from left to ...
- 吴裕雄--天生自然python学习笔记:python文档操作批量替换 Word 文件中的文字
我们经常会遇到在不同的 Word 文件中的需要做相同的文字替换,若是一个一个 文件操作,会花费大量时间 . 本节案例可以找出指定目录中的所有 Word 文件(包含 子目录),并对每一个文件进行指定的文 ...
- 爬虫之使用requests爬取某条标签并生成词云
一.爬虫前准备 1.工具:pychram(python3.7) 2.库:random,requests,fake-useragent,json,re,bs4,matplotlib,worldcloud ...
- JavaScript学习总结(一)基础部分
转自:http://segmentfault.com/a/1190000000652749 基本概念 javascript是一门解释型的语言,浏览器充当解释器. js执行引擎并不是一行一行的执行,而是 ...
- 10X genomics|cell base|in-vivo based|model organisms|SBI|
生命组学-药物基因组学 精准医学的内容有个人全基因组测序,移动可穿戴设备,它可以实时监测,深度学习模型预测疾病,对疾病预测做到有效.安全和可控. 药物基因组学就是研究疾病.化合物和靶点之间的关系,关键 ...
- 瑞星发布Linux系统安全报告:Linux病毒或将大面积爆发
近半年来,由于中央推荐使用国产Linux操作系统,国产Linux操作系统开始受到政府机关及大型企事业机关单位的高度重视.很多人都认为,以Linux系统为基础的国产操作系统最符合国家.政府和企业信息安全 ...
