U-Net: Convolutional Networks for Biomedical Image Segmentation(理解+github代码)
github代码:https://github.com/Chet1996/pytorch-UNet
0 - Abstract
这篇文章是生物学会议ICMICCAI2015的文章,主要针对的是生物影像进行分割。由于普遍认为深度学习需要大量的样本进行训练,而生物医学领域上的数据量比较少,所以本文提出了一种网络和训练策略,依靠数据增强等技巧有效的利用了有限的标签信息。该体系结构包括捕捉上下文的收缩路径(contracting path)和实现精确定位的对称扩展路径(symmetric expanding path)。实验表明,该网络结构可以在非常少的图像数据集上进行端到端训练。
1 - Introduction & Network Architecture
Ciresan等人使用滑动窗口,提高围绕该像素的局部区域(补丁)作为输入来预测每个像素的类别标签。虽然该方法可以达到很好的精度,但是存在两个缺点:
- 速度非常慢。因为网络必须分别为每个补丁运行,并且由于补丁的重叠造成大量的冗余;
- 精确度和局部区域(补丁)大小的权衡。较大局部区域带来更多的信息但需要更多的缓冲层(例如最大池化层)来处理,较小局部区域使得上下文信息变少。
本文提出的网络,是全卷积网络,其中主要是想是通过逐层扩充来补充通常的收缩网络(下采样),其中pooling被unsampling操作代替(称之为上采样),这些层能够增加输出的分辨率。为了精准定位每一个像素,下采样和上采样路径中相同尺度的特征进行连接,整体架构如下图:
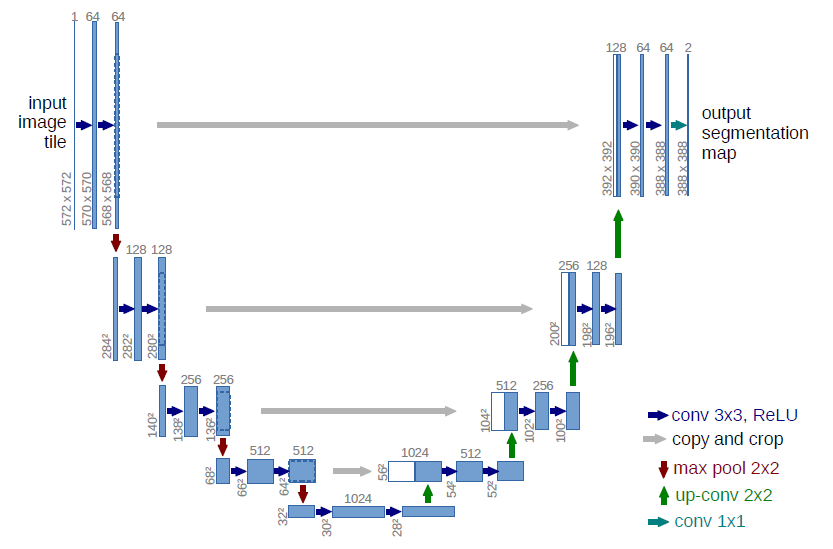
从上图可以看到,作者采用的3x3卷积的padding设置为0(unpadded的卷积),因此每一次卷积都会使得特征尺度在h和w上均减少2,从而使得,上采样路径得到的特征图尺度和下采样路径的特征图尺度不完全相同(下采样的特征图大于上采样),所以需要先对下采样特征图进行裁剪之后再和上采样特征图进行连接(即图中表述的copy and crop)。(我猜想,应该是当时文章发出的时候,计算力的限制,导致要求输出和输入具有同等大小的分辨率会牺牲很多的实效性,因此作者做了如此一个权衡。在实验过程和我的认识中,如果对于特征图进行裁剪,是会损失一些特征信息的,因此我在我的代码中并没有完全按照文章的做法复现,而是把输入和输出都统一到512x512的分辨率,并且3x3卷积的padding都设置为1)
2 - Training
- input/output:输入为572x572分辨率的图像,标签为388x388的分割图;
- batch size:为了最小化开销并最大限度地利用GPU内存,我们倾向于使用大的输入块而不是大的批处理大小,从而将批处理减少到单个图像,即batch_size设置为1;
- optimizer:SGD(随机梯度下降)优化器,其momentum(动量)设置为0.99,使得几乎所有之前训练的样本都能影响到当前训练样本的更新(我觉得就和batch size设置得比较大的效果应该是一样的);
- criterion:交叉熵损失函数(但我在我的代码实现中使用了sigmoid+BCELoss代替了交叉熵损失函数),作者通过预先计算每个真实分割的权重图,来补偿训练集中不同类别的不同频率,并迫使网络学习我们的触摸单元之间引入的小分离边界。分离边界使用形态学操作来计算,计算权重图通过公式$w(x)=w_c(x)+w_0*exp(-\frac{(d_1(x)+d_2(x))^2}{2\sigma^2})$,其中$w_c$是权重图用来平衡像素的频率,$d_1$表示最近单元边界的距离,$d_2$表示到第二进单元的边界的距离,文中设置$w_0= 10, \sigma\approx 5pixels$(涉及到形态学和边界的部分还没有搞懂,后续需要补充);
- initialize:文中提出使用标准偏差为$\sqrt{\frac{2}{N}}$的高斯分布来初始化卷积网络的kernel,其中N表示一个神经元输入节点的数量,例如3x3的64通道的卷积层的$N= 3*3*64= 576$;
- data augmentation:文中主要使用移位、旋转、变形、灰度值变化等数据增强方法,其中似乎是训练样本的随机弹性变形是训练具有很少标签的分割网络的关键。文中使用随机位移矢量在粗糙的3x3网络上生成平滑变形,位移从10像素便准偏差的高斯分布中采样,然后使用双三次插值计算每个像素位移。下采样路径末尾的dropout层执行进一步的隐式数据增强(这一点似乎网络结构图没有体现,按文中的意思应该是在下采样路径末尾加入了dropout层从而防止过拟合而达到相当于图像增强的效果)。
3 - My code
https://github.com/Chet1996/pytorch-UNet
我基于文中的思想和文中提到的EM segmentation challenge数据集大致复现了该网络(github代码)。其中为了代码的简洁方便,有几点和文中提出的有所不同:
- 将输入输出统一到512x512(文中输入为572x572,输出为388x388);
- 将输出的通道数改为1,而后接上sigmoid激活,再用BCELoss计算损失(文中输出通道为2,而后通过softmax激活,再用交叉熵损失函数计算损失);
- 只采用随机水平、垂直翻转作为数据增强(文中采用了移位、旋转、变形、灰度值变化等数据增强方法,并且似乎在下采样路径末尾加入了dropout层);
- 没有引入文中所提到的分离边界的技巧;
- 加入了batch normalize层。
我的训练参数如下:
- train/val:将前28张图片作为训练集,后2张图片作为验证集;
- data augmentation:随机水平、垂直翻转;
- input/output:1x512x512;
- optimizer:SGD优化器,其中lr设置为0.01,momentum设置为0.99,weight_decay设置为0.0005;
- criterion:BCELoss;
- epochs:60;
- batch size:1;
- lr decay:每30个epoch衰减为原来lr的0.1;
- initialize:文中提到的初始化方法;
- batch normalize:在每一层卷积层后面加入了bn层。
训练数据可视化如下图:

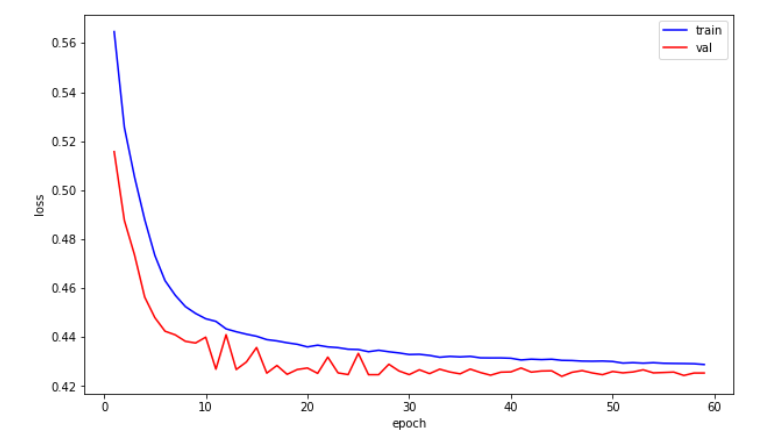
训练集和验证集的loss变化曲线如下图:
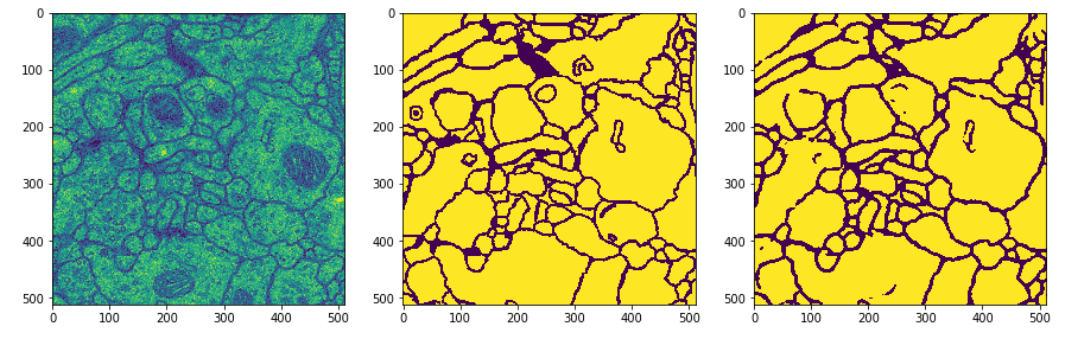
在验证集上的预测效果如下图(第一张图为输入图片,第二张图为标签,第三张图为网络预测结果):
 4 - 参考资料
4 - 参考资料
https://blog.csdn.net/u014451076/article/details/79424233
https://blog.csdn.net/shine19930820/article/details/80098091
https://github.com/Chet1996/pytorch-UNet
U-Net: Convolutional Networks for Biomedical Image Segmentation(理解+github代码)的更多相关文章
- 【Semantic Segmentation】U-Net: Convolutional Networks for Biomedical Image Segmentation 论文解析(转)
目录 0. 前言 1. 第一篇 2. 第二篇 3. 第三篇keras实现 4. 一篇关于U-Net的改进 0. 前言 今天读了U-Net觉得很不错,同时网上很多很好很详细的讲解,因此就不再自己写一 ...
- Paper | U-Net: Convolutional Networks for Biomedical Image Segmentation
目录 故事背景 U-Net 具体结构 损失 数据扩充 发表在2015 MICCAI.原本是一篇医学图像分割的论文,但由于U-Net杰出的网络设计,得到了8k+的引用. 摘要 There is larg ...
- 论文阅读笔记五:U-Net: Convolutional Networks for Biomedical Image Segmentation(CVPR2015)
前面介绍了两个文本检测的网络,分别为RRCNN和CTPN,接下来鄙人会介绍语义分割的一些经典网络,同样也是论文+代码实现的过程,这里记录一下自己学到的东西,首先从论文下手吧. 英文论文原文地址:htt ...
- Convolutional Networks for Image Semantic Segmentation
本系列文章由 @yhl_leo 出品,转载请注明出处. 文章链接: http://blog.csdn.net/yhl_leo/article/details/52857657 把前段时间自己整理的一个 ...
- Fully Convolutional Networks for semantic Segmentation(深度学习经典论文翻译)
摘要 卷积网络在特征分层领域是非常强大的视觉模型.我们证明了经过端到端.像素到像素训练的卷积网络超过语义分割中最先进的技术.我们的核心观点是建立"全卷积"网络,输入任意尺寸,经过有 ...
- 论文学习:Fully Convolutional Networks for Semantic Segmentation
发表于2015年这篇<Fully Convolutional Networks for Semantic Segmentation>在图像语义分割领域举足轻重. 1 CNN 与 FCN 通 ...
- 【Semantic Segmentation】 Instance-sensitive Fully Convolutional Networks论文解析(转)
这篇文章比较简单,但还是不想写overview,转自: https://blog.csdn.net/zimenglan_sysu/article/details/52451098 另外,读这篇pape ...
- 【Semantic segmentation】Fully Convolutional Networks for Semantic Segmentation 论文解析
目录 0. 论文链接 1. 概述 2. Adapting classifiers for dense prediction 3. upsampling 3.1 Shift-and-stitch 3.2 ...
- [论文理解]Region-Based Convolutional Networks for Accurate Object Detection and Segmentation
Region-Based Convolutional Networks for Accurate Object Detection and Segmentation 概括 这是一篇2016年的目标检测 ...
随机推荐
- 解决docker 下来镜像出现 error pulling image configuration: Get https://dseasb33srnrn.cloudfront.net的问题
http://f2d6cb40.m.daocloud.io [root@node2 ~]# docker --version ...
- flask之基础概念
[应用]一个 Flask 应用是一个 Flask 类的实例.可以在一个被称为应用工厂的函数内部创建 Flask实例.所有应用相关的配置.注册和其他设置都会在函数内部完成,然后返回这个应用.__init ...
- laravel 接收json串
在做项目的时候发现 用平时的$request->all() 无法获取到请求值 然后这样解决了 但是还是不知道原因 学习源头: http://www.cnblogs.com/anjuncc/p/5 ...
- JavaScript 中事件绑定的三种方式
以下是在 JS 中事件绑定的三种方式. 1. HTML onclick attribute <button type="button" id="uplo ...
- Struts2中web.xml里面struts-cleanup作用
struts2.1.3之后的版本均不需要配置该过滤器 参考: struts-cleanup作用 升级到struts-2.3.14.3之后涉及的改动以及ActionContextCleanUp,Stru ...
- 使用cython把python编译so
1.需求 为了保证线上代码安全和效率,使用python编写代码,pyc可直接反编译,于是把重要代码编译so文件 2.工作 2.1 安装相关库: pip install cython yum insta ...
- 类型:Java;问题:eclipse配置maven;结果:eclipse配置maven
eclipse配置maven 下面跟大家分享的是eclipse配置maven的方法. 方法/步骤 安装maven之前,要先安装jdk及配置JAVA_HOME环境变量.JDK1.4以上. 下载maven ...
- C#实现有向无环图(DAG)拓扑排序
对一个有向无环图(Directed Acyclic Graph简称DAG)G进行拓扑排序,是将G中所有顶点排成一个线性序列,使得图中任意一对顶点u和v,若边(u,v)∈E(G),则u在线性序列中出现在 ...
- MySQL学习笔记之一---字符编码和字符集
前言: 一般来说,出现中文乱码,都是客户端和服务端字符集不匹配导致的原因. (默认未指定字符集创建的数据库表,都是latinl字符集, 强烈建议使用utf8字符集) 保证不出现乱码的思想:保证客户 ...
- xcode编译静态库选择cpu架构
此前编译了一个静态库,默认支持了armv7,armv7s,arm64 编译的话肯定是上面三个静态库分别编译出来,然后在把这三个合并成一个单独的库. 如果单个库的大小是10M,那编译总的库大概就30M了 ...
