吴裕雄--天生自然 R语言开发学习:方差分析


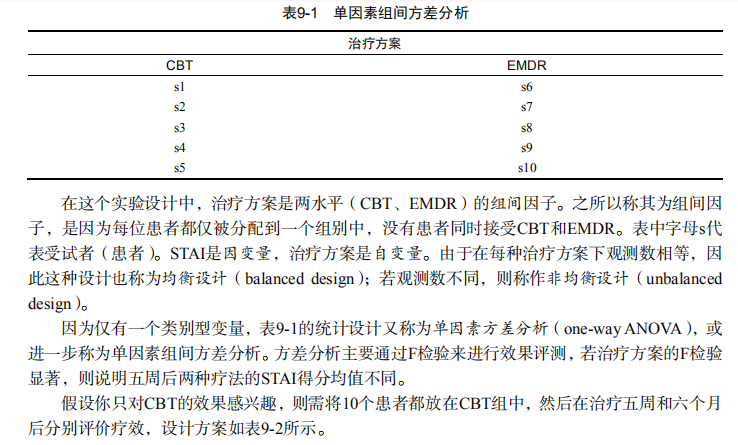
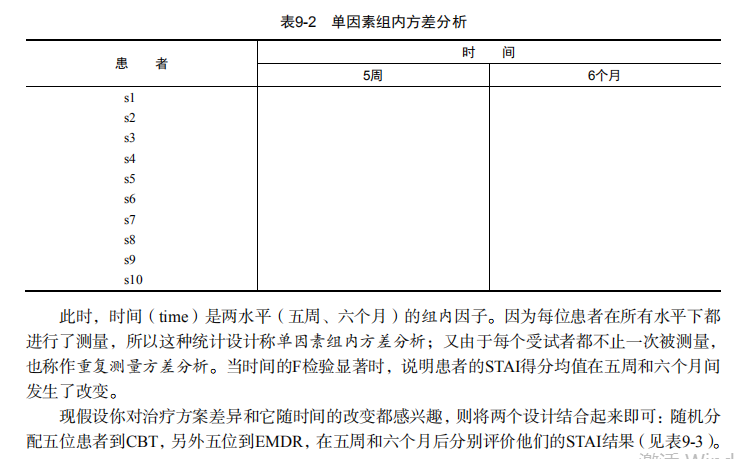
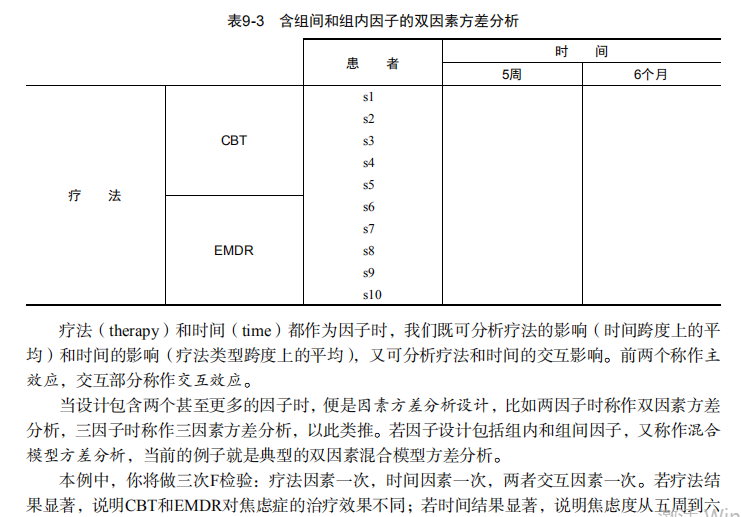









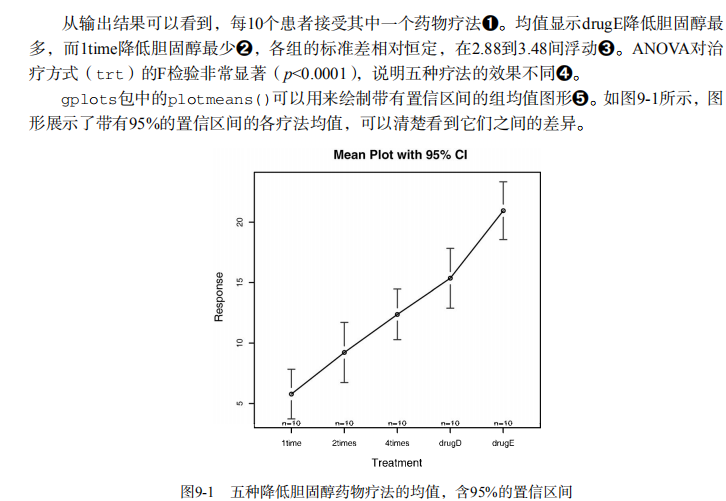













#-------------------------------------------------------------------#
# R in Action (2nd ed): Chapter 9 #
# Analysis of variance #
# requires packages multcomp, gplots, car, HH, effects, #
# rrcov, mvoutlier to be installed #
# install.packages(c("multcomp", "gplots", "car", "HH", "effects", #
# "rrcov", "mvoutlier")) #
#-------------------------------------------------------------------# par(ask=TRUE)
opar <- par(no.readonly=TRUE) # save original parameters # Listing 9.1 - One-way ANOVA
library(multcomp)
attach(cholesterol)
table(trt)
aggregate(response, by=list(trt), FUN=mean)
aggregate(response, by=list(trt), FUN=sd)
fit <- aov(response ~ trt)
summary(fit)
library(gplots)
plotmeans(response ~ trt, xlab="Treatment", ylab="Response",
main="Mean Plot\nwith 95% CI")
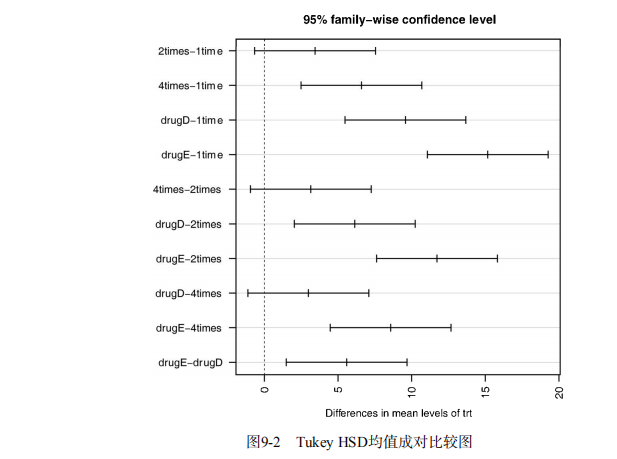
detach(cholesterol) # Listing 9.2 - Tukey HSD pairwise group comparisons
TukeyHSD(fit)
par(las=2)
par(mar=c(5,8,4,2))
plot(TukeyHSD(fit))
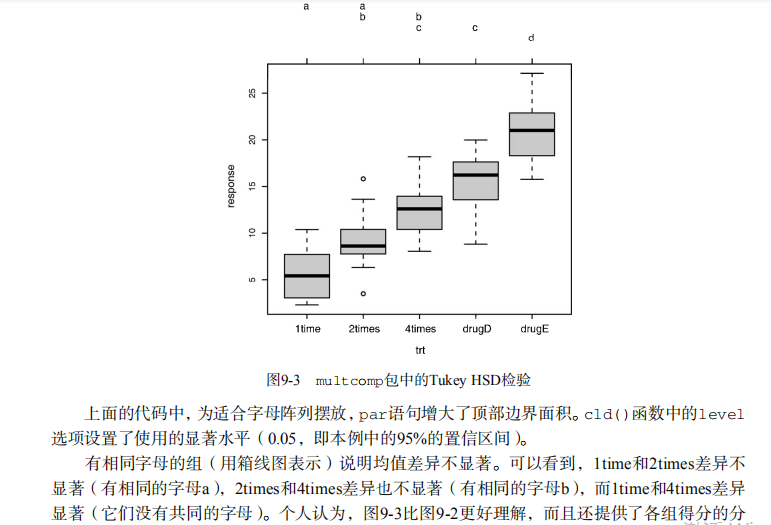
par(opar) # Multiple comparisons the multcomp package
library(multcomp)
par(mar=c(5,4,6,2))
tuk <- glht(fit, linfct=mcp(trt="Tukey"))
plot(cld(tuk, level=.05),col="lightgrey")
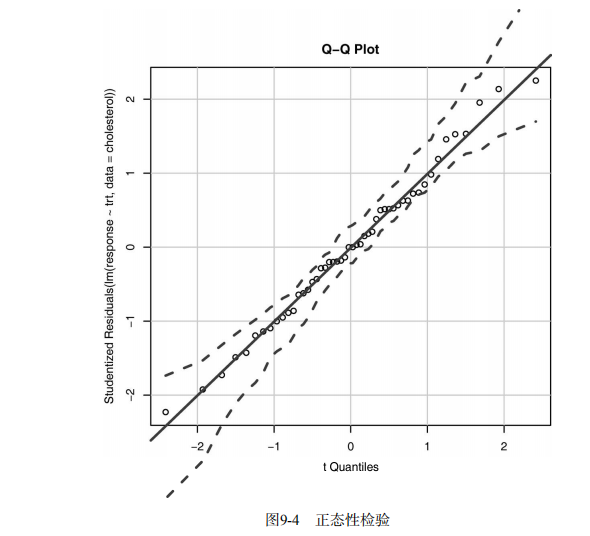
par(opar) # Assessing normality
library(car)
qqPlot(lm(response ~ trt, data=cholesterol),
simulate=TRUE, main="Q-Q Plot", labels=FALSE) # Assessing homogeneity of variances
bartlett.test(response ~ trt, data=cholesterol) # Assessing outliers
library(car)
outlierTest(fit) # Listing 9.3 - One-way ANCOVA
data(litter, package="multcomp")
attach(litter)
table(dose)
aggregate(weight, by=list(dose), FUN=mean)
fit <- aov(weight ~ gesttime + dose)
summary(fit) # Obtaining adjusted means
library(effects)
effect("dose", fit) # Listing 9.4 - Multiple comparisons using user supplied contrasts
library(multcomp)
contrast <- rbind("no drug vs. drug" = c(3, -1, -1, -1))
summary(glht(fit, linfct=mcp(dose=contrast))) # Listing 9.5 - Testing for homegeneity of regression slopes
library(multcomp)
fit2 <- aov(weight ~ gesttime*dose, data=litter)
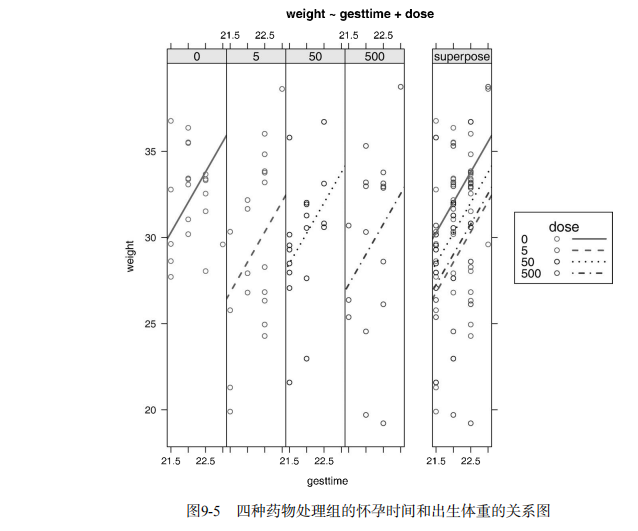
summary(fit2) # Visualizing a one-way ANCOVA
library(HH)
ancova(weight ~ gesttime + dose, data=litter) # Listing 9.6 - Two way ANOVA
attach(ToothGrowth)
table(supp,dose)
aggregate(len, by=list(supp,dose), FUN=mean)
aggregate(len, by=list(supp,dose), FUN=sd)
dose <- factor(dose)
fit <- aov(len ~ supp*dose)
summary(fit) # plotting interactions
interaction.plot(dose, supp, len, type="b",
col=c("red","blue"), pch=c(16, 18),
main = "Interaction between Dose and Supplement Type")
library(gplots)
plotmeans(len ~ interaction(supp, dose, sep=" "),
connect=list(c(1, 3, 5),c(2, 4, 6)),
col=c("red","darkgreen"),
main = "Interaction Plot with 95% CIs",
xlab="Treatment and Dose Combination")
library(HH)
interaction2wt(len~supp*dose) # Listing 9.7 - Repeated measures ANOVA with one between and within groups factor
CO2$conc <- factor(CO2$conc)
w1b1 <- subset(CO2, Treatment=='chilled')
fit <- aov(uptake ~ (conc*Type) + Error(Plant/(conc)), w1b1)
summary(fit)
par(las=2)
par(mar=c(10,4,4,2))
with(w1b1,
interaction.plot(conc,Type,uptake,
type="b", col=c("red","blue"), pch=c(16,18),
main="Interaction Plot for Plant Type and Concentration"))
boxplot(uptake ~ Type*conc, data=w1b1, col=(c("gold","green")),
main="Chilled Quebec and Mississippi Plants",
ylab="Carbon dioxide uptake rate (umol/m^2 sec)")
par(opar) # Listing 9.8 - One-way MANOVA
library(MASS)
attach(UScereal)
shelf <- factor(shelf)
y <- cbind(calories, fat, sugars)
aggregate(y, by=list(shelf), FUN=mean)
cov(y)
fit <- manova(y ~ shelf)
summary(fit)
summary.aov(fit) # Listing 9.9 - Assessing multivariate normality
center <- colMeans(y)
n <- nrow(y)
p <- ncol(y)
cov <- cov(y)
d <- mahalanobis(y,center,cov)
coord <- qqplot(qchisq(ppoints(n),df=p),
d, main="QQ Plot Assessing Multivariate Normality",
ylab="Mahalanobis D2")
abline(a=0,b=1)
identify(coord$x, coord$y, labels=row.names(UScereal)) # multivariate outliers
library(mvoutlier)
outliers <- aq.plot(y)
outliers # Listing 9.10 - Robust one-way MANOVA
library(rrcov)
Wilks.test(y,shelf, method="mcd") # this can take a while # Listing 9.11 - A regression approach to the Anova problem
fit.lm <- lm(response ~ trt, data=cholesterol)
summary(fit.lm)
contrasts(cholesterol$trt)
吴裕雄--天生自然 R语言开发学习:方差分析的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:基础知识
1.基础数据结构 1.1 向量 # 创建向量a a <- c(1,2,3) print(a) 1.2 矩阵 #创建矩阵 mymat <- matrix(c(1:10), nrow=2, n ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- echars 柱状图点击事件
drawlineCRK() { let _this = this; ///绘制echarts 柱状图 let mycharts = this.$echarts.i ...
- 求素数的一个快速算法 Python 快速输出素数算法
思想 以100以内为例. 生成一个全是True的101大小的数组 2开始,遇到2的倍数(4,6,8,10...)都赋值为False 因为这些数字都有因子 2 3开始,遇到3的倍数(6,9,12...) ...
- Python—使用列表构造栈数据结构
class Stack(object): """ 使用列表实现栈 """ def __init__(self): self.stack = ...
- cygwin下命令行下切换目录
比我们正常切换目录多个挂载的文件夹 cygdrive
- idea远程调试tomcat部署项目(windows环境)
1.tomcat启动之前,修改apache-tomcat-8.5.34\bin\catalina.bat文件,设置调试端口 如下设置(windows环境): rem ----------------- ...
- reactor-core
<dependency> <groupId>io.projectreactor</groupId> <artifactId>reactor-core&l ...
- $identify 的 “identify” 表示一个Perl标识符,即 identifier
$identify 的 “identify” 表示一个Perl标识符,即 identifier
- skip-list(跳表)原理及C++代码实现
跳表是一个很有意思的数据结构,它实现简单,但是性能又可以和平衡二叉搜索树差不多. 据MIT公开课上教授的讲解,它的想法和纽约地铁有异曲同工之妙,简而言之就是不断地增加“快线”,从而降低时间复杂度. 当 ...
- SpringBoot集成ssm-druid-通用mapper
简单介绍 springboot 首先什么是springboot? springboot是spring的另外一款框架,设计目的是用来简化新的spring应用的搭建和开发时所需要的特定的配置,从而使开发过 ...
- 二十、linux文件系统讲解
1.分区和文件系统的关系: 为什么需要格式化呢?这是因为分区文件系统在没有格式化前,操作系统是无法识别系统分区的格式的,就没办法组织文件目录属性和权限等内容,把分区格式化成操作系统支持的某个文件系统后 ...
