RNA-seq 生物学重复相关性验证
根据拿到的表达矩阵设为exprSet
1、用scale 进行标准化
数据中心化:数据集中的各个数字减去数据集的均值
数据标准化:中心化之后的数据在除以数据集的标准差。
在R中利用scale方法来对数据进行中心化和标准化
1 scale(data, center=T, scale=F)
2
3 其中,center为T,表示数据中心化
4
5 scale为T,表示数据标准化
6
7 对一个data frame的每一列进行计算
并不是表达矩阵里面的所有基因都可以进行相关性分析,首先去除reads count >1 小于5个的基因(测试样品共有6个)
1 ##过滤reads count >1 小于5
2 exprSet <- exprSet[apply(exprSet,1, function(x) sum(x>1) >5,]
3 ##reads count 差距较大,用log以及scale 缩小差距
4 M <- scale(cor(llog2(exprSet+1)))
5 ##热图
6 pheatmap(M)
2、另一种标准化
1 exprSet <- exprSet[apply(exprSet,1, function(x) sum(x>1) >5,]
2
3 ##去除文库大小差异
4 exprSet <- log(edgeR::cpm(exprSet)+1)
5
6 ##取mad(绝对中位差)(类似sd)的前50%
7 exprSet <- exprSet[names(sort(apply(exprSet,1,mad),decreasing = T)[1:500]),] ##取前500
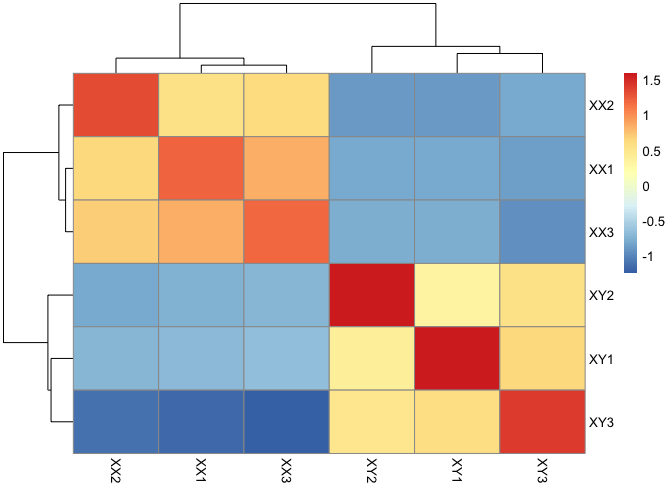
8 M <-cor(log2(exprSet+1))
9
10 ##添加group_list
11 tem = data.frame(g=group_list)
12 rownames(tem) <- colnames(M)
13 pheatmap::pheatmap(M, annotation_col = tem,filename = 'cor.png')
3、hclust 聚类分析
欧式距离(Euclidean Distance)
欧式距离是最易于理解的一种距离计算方法,源自欧式空间中两点间的距离公式。
用R语言计算距离主要是dist函数。若X是一个M×N的矩阵,则dist(X)将X矩阵M行的每一行作为一个N维向量,然后计算这M个向量两两间的距离。
表达矩阵是每一行为基因,列为样品名称,所以要进行转至才能计算每个样品之间基因表达量的距离
为了得到更好的聚类分析,也可以将表达矩阵标准化,譬如,log,或者 scale等
1 hc <- hclust(dist(t(exprSet)))
2 plot(hc)
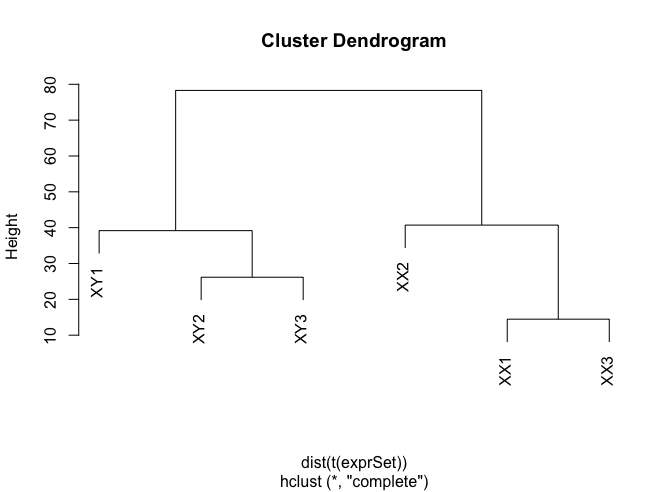
参考:生信技能树
RNA-seq 生物学重复相关性验证的更多相关文章
- 无生物学重复RNA-seq分析 CORNAS: coverage-dependent RNA-Seq analysis of gene expression data without biological replicates
无生物学重复RNA-seq分析 CORNAS: coverage-dependent RNA-Seq analysis of gene expression data without biologic ...
- RNA-seq要做几次生物学重复?找出来的100%都是真正的应答基因
尹师妹:“哈师兄,做验证实验好辛苦,老板让我提高筛选差异基因的条件,尽量降低假阳性,我该怎么筛?” 小哈打开Evernote,给尹师妹看张表: “瞧见那个100%了吗?30 million mappe ...
- RNA seq 两种计算基因表达量方法
两种RNA seq的基因表达量计算方法: 1. RPKM:http://www.plob.org/2011/10/24/294.html 2. RSEM:这个是TCGAdata中使用的.RSEM据说比 ...
- RNA -seq
RNA -seq RNA-seq目的.用处::可以帮助我们了解,各种比较条件下,所有基因的表达情况的差异. 比如:正常组织和肿瘤组织的之间的差异:检测药物治疗前后,基因表达的差异:检测发育过程中,不同 ...
- 使用RDCMan管理SharePoint虚拟机的重复要求验证的问题
首先,这个软件可以从这里下载: Remote Desktop Connection Manager 同类型的软件还有很多,我没有很多复杂功能的要求,就选择了这款微软官方的,虽然很久都没有更新过了. 为 ...
- easy UI的密码长度以及重复输入验证
自己些项目的时候找的时候也找了一会,所以存下来下次用的时候可以直接用了. 话不多说,直接上代码 <tr> <td>密码:</td> <td><in ...
- seq去除重复数据
DELETE FROM temp_fjh_2 a WHERE a.rowid!=(SELECT MAX(b.rowid) FROM temp_fjh_2 b WHERE a.a=b.a); 表名和列名 ...
- 知乎Live总结-重复nature文章笔记Single-cell
来自知乎Live-孟浩巍 1.文章重要技术及图讲解 首先在转录组RNA-seq中,有基因表达差异.基因融合.可变剪切.RNA单点突变. 在基因组中,单点变异.结构变异,CNV变异(拷贝数变异) 三类基 ...
- Circular RNA的产生机制、功能及RNA-seq数据鉴定方法
推荐关注微信公众号:AIPuFuBio,和使用免费生物信息学资源和工具AIPuFu:http://www.aipufu.com. [Circular RNA的产生机制] Circular RNA,缩写 ...
随机推荐
- 异常大讨论-抛出异常还是返回false
iteye精华帖之异常大讨论 原帖链接http://www.iteye.com/topic/2038 Robbin的观点 观点1:Exception实际上代表了一个UseCase中的异常流的处理. 绝 ...
- Alpha发布声明
项目 内容 这个作业属于哪个课程 2021春季软件工程(罗杰 任健) 这个作业的要求在哪里 Alpha-发布声明 我们是谁 删库跑路对不队 我们在做什么 题士 进度如何 进度总览 一.功能与特性 1. ...
- Github Actions 实践
Github Actions 实践 Github Actions 是 Github 的持续集成服务,通过在 repo 发生特定的行为时执行指定的命令实现自动测试.自动部署等功能. 基本术语 workf ...
- 2021.10.7考试总结[NOIP模拟71]
信心赛,但炸了.T3SB错直接炸飞,T4可以硬算的组合数非要分段打表求阶乘..T2也因为一个细节浪费了大量时间.. 会做难题很好,但首先还是要先把能拿的分都拿到. T1 签到题 结论:总可以做到对每个 ...
- 如何用PADS进行PCB设计?这6步就够了
在使用PADS进行PCB设计的过程中,需要对印制板的设计流程以及相关的注意事项进行重点关注,这样才能更好的为工作组中的设计人员提供系统的设计规范,同时也方便设计人员之间进行相互的交流和检查. 02 设 ...
- 认真讲说static关键字
static 关键字主要有以下四种使用场景 修饰成员变量和成员方法 静态代码块 修饰类(只能修饰内部类) 静态导包(用来导入类中的静态资源,1.5之后的新特性) 修饰成员变量和成员方法(常用) 被 s ...
- Swoft+Docker
Docker 以下纯属个人理解: Docker就是一种虚拟机,将环境打包成镜像,等于做了一个Linux系统裁剪. 镜像就是我们安装系统的镜像,里面包含了你的代码和环境. 容器就是一个虚拟机,你可以用一 ...
- ZooKeeper 集群搭建 Error contacting service. It is probably not running.
搭建环境:Centos 7 虚拟机 3台 按照此教程搭建:https://www.ilanni.com/?p=11393 之后出现错误:Error contacting service. It is ...
- IP数据报中如果不分片,分片标志值是什么?
过了好久才解决这个简单的问题,罪过罪过- 答案:如果IP数据报不分片,分片标志DF(Don't Fragment)会被设置为1.分片标志MF(More Fragment)设置为0. 下面是详细解释: ...
- RabbitMQ的安装及入门使(Windows)
1.安装Erlang所以在安装rabbitMQ之前,需要先安装Erlang .点击下载Erlang 执行下载下来的Erlang,全部点击"下一步"就行.安装完成设置一下环境变量. ...
