单细胞分析实录(4): doublet检测
最近Cell Systems杂志发表了一篇针对现有几种检测单细胞测序doublet的工具的评估文章,系统比较了常见的例如Scrublet、DoubletFinder等工具在检测准确性、计算效率等方面的优劣,以及比较了使用不同方法去除doublet后对下游DE分析、轨迹分析的影响。

现有的检测方法,基本都会先构造出虚拟doublet,然后将候选droplet与这些虚拟doublet比较,很相似的那些就定义为doublet。这里的虚拟doublet是通过随机组合两个(类)细胞的表达值得到的虚拟的doublet,可以作为检测时的参照。
在现有的9种方法中(Scrublet、doubletCells、cxds、bcds、Hybrid、DoubletDetection、DoubletFinder、Solo、DoubletDecon),文章的结论是DoubletFinder的准确率最高。
里面的方法我用过三种:Scrublet、DoubletFinder和DoubletDecon。前面两个用法类似,需要提供一个参数表示doublet的占比。DoubletDecon的原文我看过,算法比较简单,不需要提供doublet的占比,减少了用户额外的输入,不过也造成了一些麻烦,有时候会报告出很多doublet,多得惊人。实际分析中,我采用“两步走”的策略:选取了Scrublet和DoubletFinder的共同结果作为doublet去除掉,此外在后续聚类分亚群的过程中,根据一些已知的经典的细胞marker来判断doublet,比如CD45和EPCAM都高表达的亚群极有可能是doublet。
接下来,我会简单介绍DoubletDecon、DoubletFinder、Scrublet三个工具的使用。
1. DoubletDecon
该方法中间有一步用到了类似bulk RNA-seq里面deconvolution的思路来评估每一个细胞的表达模式,因此叫DoubletDecon。
这里用含有500个细胞的真实数据作为例子。在使用DoubletDecon之前,需要先用seurat对数据进行初聚类,seurat的使用我后面会详细讲,这里先把标准流程放上来。
library(tidyverse)
library(Seurat)
library(DoubletDecon)
test=read.table("test.count.txt",header = T,row.names = 1)
test.seu <- CreateSeuratObject(counts = test)
#Normalize
test.seu <- NormalizeData(test.seu, normalization.method = "LogNormalize", scale.factor = 10000)
#FindVariableFeatures
test.seu <- FindVariableFeatures(test.seu, selection.method = "vst", nfeatures = 2000)
#Scale
test.seu <- ScaleData(test.seu, features = rownames(test.seu))
#PCA
test.seu <- RunPCA(test.seu, features = VariableFeatures(test.seu),npcs = 50)
#cluster
test.seu <- FindNeighbors(test.seu, dims = 1:20)
test.seu <- FindClusters(test.seu, resolution = 0.5)
test.seu <- RunUMAP(test.seu, dims = 1:20)
test.seu <- RunTSNE(test.seu, dims = 1:20)
然后才是DoubletDecon的代码
#Improved_Seurat_Pre_Process()
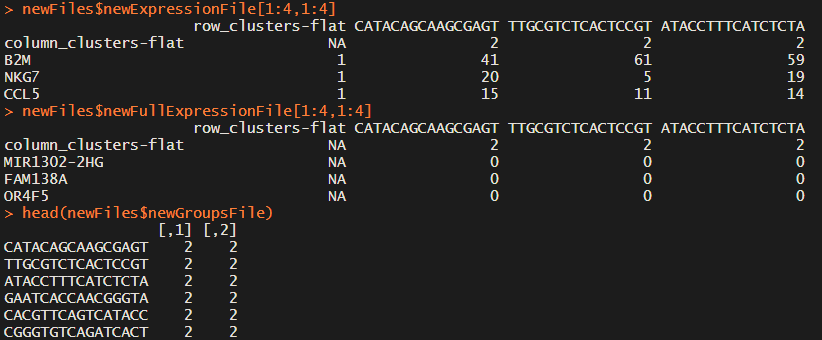
newFiles=Improved_Seurat_Pre_Process(test.seu, num_genes=50, write_files=FALSE)
这一步主要是找差异基因,会返回三个表格,分别表示marker基因的表达矩阵、所有基因的表达矩阵、细胞的seurat_cluster注释,前两个文件的第一行第一列有相应的注释。
然后就是找doublet的主要步骤了
#Main_Doublet_Decon
results=Main_Doublet_Decon(rawDataFile=newFiles$newExpressionFile,
groupsFile=newFiles$newGroupsFile,
filename="tmp",
location="./",
species="hsa",
rhop=1,
num_doubs=80,
write=FALSE,
heatmap=TRUE,
centroids=TRUE,
nCores=2)
这里面有几个很重要的参数,rhop默认值为1,用它来调节皮尔森相关系数的阈值(如下图)。在seurat聚类之后,这个软件会进一步将很相似的cluster合并,利用的就是最初cluster之间表达的相关性。这个值也很麻烦,前面提到的DoubletDecon会检测出很多doublet的问题可能就是这个参数设置不对导致的。那这个参数应该如何设置,可能软件作者也意识到了这个问题,后面又发了一篇Protocol,专门讲参数如何选择,核心思想就是多试几次。(事儿真多,准备放弃这个软件了)

接下来把DoubletDecon的检测结果保存成单独的文件,方便后面使用
doublet_df <- as.data.frame(results$Final_doublets_groups)
doublet_df$DoubletDecon="Doublet"
singlet_df <- as.data.frame(results$Final_nondoublets_groups)
singlet_df$DoubletDecon="Singlet"
DD_df <- rbind(doublet_df,singlet_df)
DD_df <- DD_df[colnames(test),]
DD_df$CB = colnames(test)
DD_df <- DD_df[,c("CB","DoubletDecon")]
write.table(DD_df, file = "DoubletDecon_result.txt", quote = FALSE, sep = '\t', row.names = FALSE, col.names = TRUE)
也可以将doublet的结果投到tsne图上看看效果
test.seu@meta.data$CB=rownames(test.seu@meta.data)
test.seu@meta.data=inner_join(test.seu@meta.data,DD_df,by="CB")
rownames(test.seu@meta.data)=test.seu@meta.data$CB
DimPlot(test.seu,reduction = "tsne",pt.size = 2,group.by = "DoubletDecon")
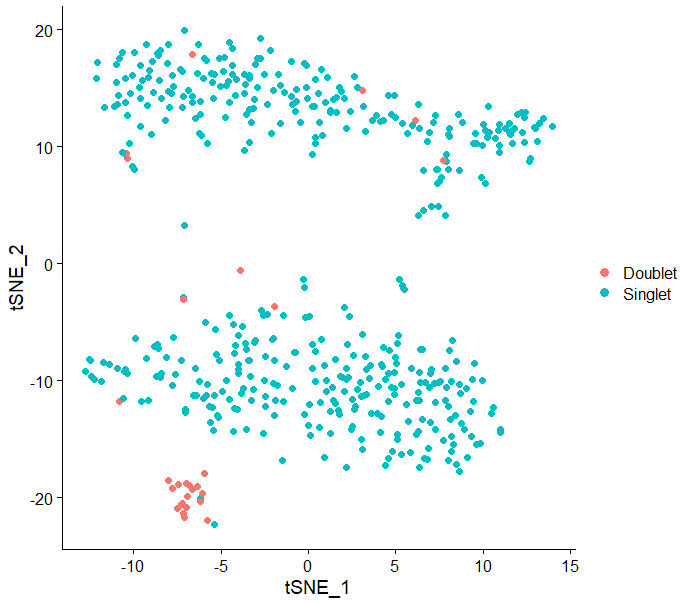
看上去还不戳,符合doublet单独成群的预期
2. DoubletFinder
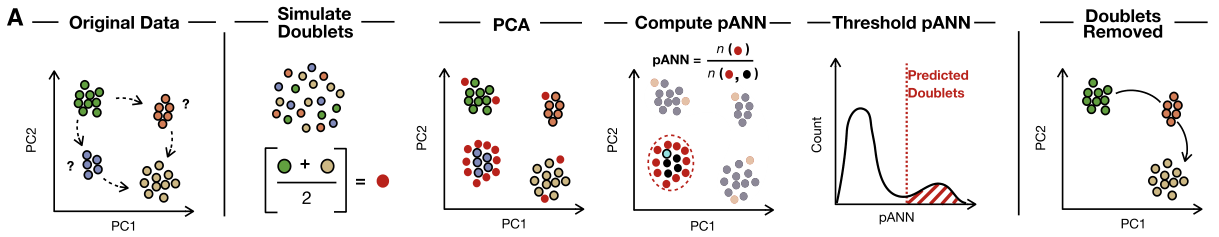
DoubletFinder找doublet的原理也比较简单,看细胞群里面虚拟doublet的占比,超过某个阈值就认定这一群的真实细胞是doublet。在运行DoubletFinder之前,需要对细胞进行初聚类,这和上一种方法是一样的。
library(Seurat)
library(DoubletFinder)
test.seu <- Createtest.seuratObject(counts = test)
test.seu <- NormalizeData(test.seu, normalization.method = "LogNormalize", scale.factor = 10000)
test.seu <- FindVariableFeatures(test.seu, selection.method = "vst", nfeatures = 2000)
test.seu <- ScaleData(test.seu, features = rownames(test.seu))
test.seu <- RunPCA(test.seu, features = VariableFeatures(test.seu),npcs = 50)
test.seu <- FindNeighbors(test.seu, dims = 1:20)
test.seu <- FindClusters(test.seu, resolution = 0.5)
test.seu <- RunUMAP(test.seu, dims = 1:20)
test.seu <- RunTSNE(test.seu, dims = 1:20)
接下来选择一个重要参数pK,这个参数定义了PC的neighborhood size
sweep.res.list <- paramSweep_v3(test.seu, PCs = 1:10, sct = FALSE)
for(i in 1:length(sweep.res.list)){
if(length(sweep.res.list[[i]]$pANN[is.nan(sweep.res.list[[i]]$pANN)]) != 0){
if(i != 1){
sweep.res.list[[i]] <- sweep.res.list[[i - 1]]
}else{
sweep.res.list[[i]] <- sweep.res.list[[i + 1]]
}
}
}
sweep.stats <- summarizeSweep(sweep.res.list, GT = FALSE)
bcmvn <- find.pK(sweep.stats)
pk_v <- as.numeric(as.character(bcmvn$pK))
pk_good <- pk_v[bcmvn$BCmetric==max(bcmvn$BCmetric)]
nExp_poi <- round(0.1*length(colnames(test.seu)))
指定期望的doublet数
test.seu <- doubletFinder_v3(test.seu, PCs = 1:10, pN = 0.25, pK = pk_good, nExp = nExp_poi, reuse.pANN = FALSE, sct = FALSE)
这一行是主要代码,会在test.seu@meta.data数据框的基础上加上两列
colnames(test.seu@meta.data)[ncol(test.seu@meta.data)]="DoubletFinder"
第二列换一个列名
DF_df <- test.seu@meta.data[,c("CB","DoubletFinder")]
write.table(DF_df, file = "DoubletFinder_result.txt", quote = FALSE, sep = '\t', row.names = FALSE, col.names = TRUE)
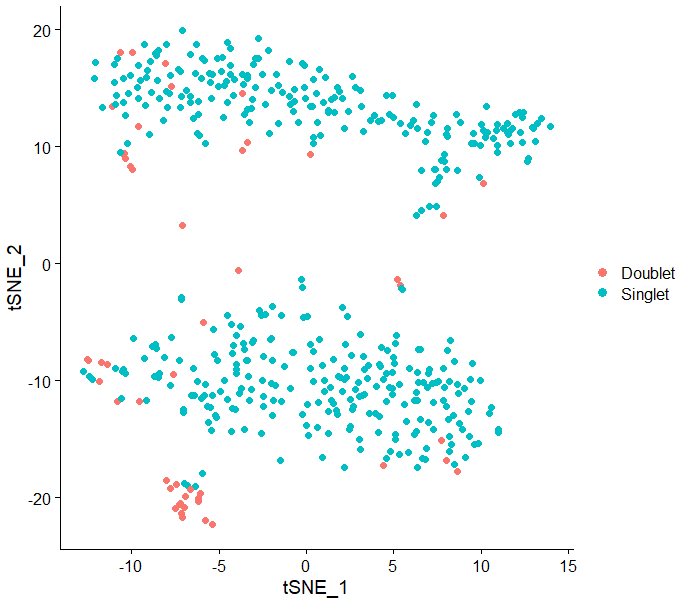
DimPlot(test.seu,reduction = "tsne",pt.size = 2,group.by = "DoubletFinder")
最终的效果如下图所示:
3. Scrublet
是一个Python包,安装可以参考:https://github.com/AllonKleinLab/scrublet
>>> import scrublet as scr
>>> import numpy as np
>>> infile = "test.count.txt"
>>> outfile = "Scrublet_result.txt"
下面的代码对输入文件做预处理,包括提取出CB,提取count矩阵并转置
>>> finallist = []
>>> with open(infile, 'r') as f:
... header = next(f)
... cell_barcodes = header.rstrip().split('\t')
... for line in f:
... tmpline = line.rstrip().split('\t')[1: ]
... tmplist = [int(s) for s in tmpline]
... finallist.append(tmplist)
>>> finalarray = np.array(finallist)
>>> count_matrix = np.transpose(finalarray)
Scrublet检测doublet主要代码如下:
>>> scrub = scr.Scrublet(count_matrix, expected_doublet_rate = 0.1)
>>> doublet_scores, predicted_doublets = scrub.scrub_doublets()
>>> predicted_doublets_final = scrub.call_doublets(threshold = 0.2)
第三行换阈值,更新注释结果,接下来保存结果
>>> with open(outfile, 'w') as f:
... f.write('\t'.join(['CB', 'Scrublet', 'Scrublet_Score']) + '\n')
... for i in range(len(doublet_scores)):
... if predicted_doublets_final[i] == 0:
... result = 'Singlet'
... else:
... result = 'Doublet'
... f.write('\t'.join([cell_barcodes[i], result, str(doublet_scores[i])]) + '\n')
切换到R环境,在tsne上看看效果
SR_df=read.table("Scrublet_result.txt",header = T,sep = "\t",stringsAsFactors = F)
test.seu@meta.data=inner_join(test.seu@meta.data,SR_df,by="CB")
rownames(test.seu@meta.data)=test.seu@meta.data$CB
DimPlot(test.seu,reduction = "tsne",pt.size = 2,group.by = "Scrublet")

到这里就把三个软件的基本使用讲完了,我只使用了一个实际数据演示,结果并不足以反映这几个软件谁好谁坏,小伙伴们需要结合自己的数据选择合适的软件。开篇提到的文献可信度还是挺高的,大家有需要的话可以认真学一下DoubletFinder这个软件。
另外,可以在github上看到这几个软件是相互推荐的,在生信圈子还挺少见~
因水平有限,有错误的地方,欢迎批评指正!
单细胞分析实录(4): doublet检测的更多相关文章
- 【代码更新】单细胞分析实录(20): 将多个样本的CNV定位到染色体臂,并画热图
之前写过三篇和CNV相关的帖子,如果你做肿瘤单细胞转录组,大概率看过: 单细胞分析实录(11): inferCNV的基本用法 单细胞分析实录(12): 如何推断肿瘤细胞 单细胞分析实录(13): in ...
- 【代码更新】单细胞分析实录(21): 非负矩阵分解(NMF)的R代码实现,只需两步,啥图都有
1. 起因 之前的代码(单细胞分析实录(17): 非负矩阵分解(NMF)代码演示)没有涉及到python语法,只有4个python命令行,就跟Linux下面的ls grep一样的.然鹅,有几个小伙伴不 ...
- 单细胞分析实录(1): 认识Cell Hashing
这是一个新系列 差不多是一年以前,我定导后没多久,接手了读研后的第一个课题.合作方是医院,和我对接的是一名博一的医学生,最开始两边的老师很排斥常规的单细胞文章思路,即各大类细胞分群.注释.描述,所以起 ...
- 单细胞分析实录(5): Seurat标准流程
前面我们已经学习了单细胞转录组分析的:使用Cell Ranger得到表达矩阵和doublet检测,今天我们开始Seurat标准流程的学习.这一部分的内容,网上有很多帖子,基本上都是把Seurat官网P ...
- 单细胞分析实录(3): Cell Hashing数据拆分
在之前的文章里,我主要讲了如下两个内容:(1) 认识Cell Hashing:(2): 使用Cell Ranger得到表达矩阵.相信大家已经知道了cell hashing与普通10X转录组的差异,以及 ...
- 单细胞分析实录(17): 非负矩阵分解(NMF)代码演示
本次演示使用的数据来自2017年发表于Cell的头颈鳞癌单细胞文章:Single-Cell Transcriptomic Analysis of Primary and Metastatic Tumo ...
- 单细胞分析实录(2): 使用Cell Ranger得到表达矩阵
Cell Ranger是一个"傻瓜"软件,你只需提供原始的fastq文件,它就会返回feature-barcode表达矩阵.为啥不说是gene-cell,举个例子,cell has ...
- 单细胞分析实录(8): 展示marker基因的4种图形(一)
今天的内容讲讲单细胞文章中经常出现的展示细胞marker的图:tsne/umap图.热图.堆叠小提琴图.气泡图,每个图我都会用两种方法绘制. 使用的数据来自文献:Single-cell transcr ...
- 单细胞分析实录(18): 基于CellPhoneDB的细胞通讯分析及可视化 (上篇)
细胞通讯分析可以给我们一些细胞类群之间相互调控/交流的信息,这种细胞之间的调控主要是通过受配体结合,传递信号来实现的.不同的分化.疾病过程,可能存在特异的细胞通讯关系,因此阐明这些通讯关系至关重要. ...
随机推荐
- Python【内置函数】、【装饰器】与【haproxyf配置文件的修改】
内置函数 •callable,检查是否能被执行/调用 def f1(): pass f2 = 123 print(callable(f1)) #输出 print(callable(f2)) #输出 T ...
- 第8章 Python类中常用的特殊变量和方法目录
第8章 Python类中常用的特殊变量和方法 第8.1节 Python类的构造方法__init__深入剖析:语法释义 第8.2节 Python类的__init__方法深入剖析:构造方法案例详解 第8. ...
- 第十六章、Model/View开发:QColumnView的作用及对应Model
老猿Python博文目录 专栏:使用PyQt开发图形界面Python应用 老猿Python博客地址 一.概述 在Qt Designer的Item Views(Model-based)部件中,Colum ...
- PyQt(Python+Qt)学习随笔:QAbstractItemView的iconSize属性
老猿Python博文目录 老猿Python博客地址 视图的iconSize属性用于控制显示icon的项上的icon图标大小,在视图可见情况下设置该属性会导致视图上的显示项重新调整布局. 可以使用ico ...
- PyQt(Python+Qt)学习随笔:gridLayout的layoutRowMinimumHeight和layoutColumnMinimumWidth属性
Qt Designer中网格布局(gridLayout)中,layoutRowMinimumHeight和layoutColumnMinimumWidth两个属性分别设置网格布局中各行的最小高度和各列 ...
- 爬虫模块-requests
title: python爬虫01 date: 2020-03-08 22:56:12 tags: 1.requests模块 requests模块的底层是urllib,但是比urllib更强大也更加简 ...
- Hive 表操作(HIVE的数据存储、数据库、表、分区、分桶)
1.Hive的数据存储 Hive的数据存储基于Hadoop HDFS Hive没有专门的数据存储格式 存储结构主要包括:数据库.文件.表.试图 Hive默认可以直接加载文本文件(TextFile),还 ...
- 数组编程题(github每日一题)
/** * 随机生成一个长度为 10 的整数类型的数组,例如 [2, 10, 3, 4, 5, 11, 10, 11, 20], * 将其排列成一个新数组,要求新数组形式如下,例如 [[2, 3, 4 ...
- hashmap底层:jdk1.8前后的改变
将hashmap和currenthashmap放一块进行比较,是因为二者的结构相差不多,只不过后者是线程安全的. 首先说hashmap,在jdk1.8之前,hashmap的存储结构是数组+链表的形式, ...
- 容器服务 TKE 存储插件与云硬盘 CBS 最佳实践应用
引言 随着自研上云的深入,越来越多的有状态服务对于在 TKE 集群中使用云上存储能力的需求也越来越强烈. 目前腾讯云容器服务 TKE(Tencent Kubernetes Engine已支持在 TKE ...
