吴裕雄--天生自然 R语言开发学习:基本统计分析













#---------------------------------------------------------------------#
# R in Action (2nd ed): Chapter 7 #
# Basic statistics #
# requires packages npmc, ggm, gmodels, vcd, Hmisc, #
# pastecs, psych, doBy to be installed #
# install.packages(c("ggm", "gmodels", "vcd", "Hmisc", #
# "pastecs", "psych", "doBy")) #
#---------------------------------------------------------------------# mt <- mtcars[c("mpg", "hp", "wt", "am")]
head(mt) # Listing 7.1 - Descriptive stats via summary
mt <- mtcars[c("mpg", "hp", "wt", "am")]
summary(mt) # Listing 7.2 - descriptive stats via sapply
mystats <- function(x, na.omit=FALSE){
if (na.omit)
x <- x[!is.na(x)]
m <- mean(x)
n <- length(x)
s <- sd(x)
skew <- sum((x-m)^3/s^3)/n
kurt <- sum((x-m)^4/s^4)/n - 3
return(c(n=n, mean=m, stdev=s, skew=skew, kurtosis=kurt))
} myvars <- c("mpg", "hp", "wt")
sapply(mtcars[myvars], mystats) # Listing 7.3 - Descriptive stats via describe (Hmisc)
library(Hmisc)
myvars <- c("mpg", "hp", "wt")
describe(mtcars[myvars]) # Listing 7.,4 - Descriptive stats via stat.desc (pastecs)
library(pastecs)
myvars <- c("mpg", "hp", "wt")
stat.desc(mtcars[myvars]) # Listing 7.5 - Descriptive stats via describe (psych)
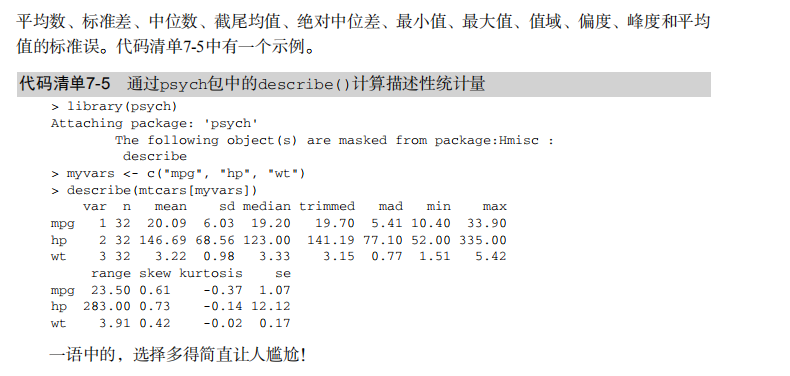
library(psych)
myvars <- c("mpg", "hp", "wt")
describe(mtcars[myvars]) # Listing 7.6 - Descriptive stats by group with aggregate
myvars <- c("mpg", "hp", "wt")
aggregate(mtcars[myvars], by=list(am=mtcars$am), mean)
aggregate(mtcars[myvars], by=list(am=mtcars$am), sd) # Listing 7.7 - Descriptive stats by group via by
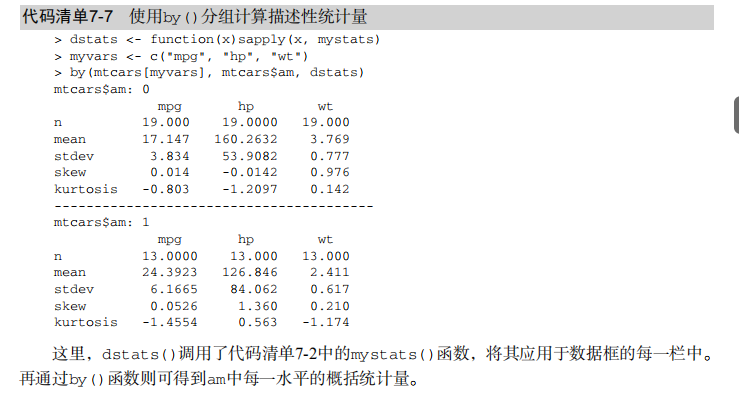
dstats <- function(x)sapply(x, mystats)
myvars <- c("mpg", "hp", "wt")
by(mtcars[myvars], mtcars$am, dstats) # Listing 7.8 - Descriptive stats by group via summaryBy
library(doBy)
summaryBy(mpg+hp+wt~am, data=mtcars, FUN=mystats) # Listing 7.9 - Descriptive stats by group via describe.by (psych)
library(psych)
myvars <- c("mpg", "hp", "wt")
describeBy(mtcars[myvars], list(am=mtcars$am)) # summary statistics by group via the reshape package
library(reshape)
dstats <- function(x)(c(n=length(x), mean=mean(x), sd=sd(x)))
dfm <- melt(mtcars, measure.vars=c("mpg", "hp", "wt"),
id.vars=c("am", "cyl"))
cast(dfm, am + cyl + variable ~ ., dstats) # frequency tables
library(vcd)
head(Arthritis) # one way table
mytable <- with(Arthritis, table(Improved))
mytable # frequencies
prop.table(mytable) # proportions
prop.table(mytable)*100 # percentages # two way table
mytable <- xtabs(~ Treatment+Improved, data=Arthritis)
mytable # frequencies
margin.table(mytable,1) #row sums
margin.table(mytable, 2) # column sums
prop.table(mytable) # cell proportions
prop.table(mytable, 1) # row proportions
prop.table(mytable, 2) # column proportions
addmargins(mytable) # add row and column sums to table # more complex tables
addmargins(prop.table(mytable))
addmargins(prop.table(mytable, 1), 2)
addmargins(prop.table(mytable, 2), 1) # Listing 7.10 - Two way table using CrossTable
library(gmodels)
CrossTable(Arthritis$Treatment, Arthritis$Improved) # Listing 7.11 - Three way table
mytable <- xtabs(~ Treatment+Sex+Improved, data=Arthritis)
mytable
ftable(mytable)
margin.table(mytable, 1)
margin.table(mytable, 2)
margin.table(mytable, 2)
margin.table(mytable, c(1,3))
ftable(prop.table(mytable, c(1,2)))
ftable(addmargins(prop.table(mytable, c(1, 2)), 3)) # Listing 7.12 - Chi-square test of independence
library(vcd)
mytable <- xtabs(~Treatment+Improved, data=Arthritis)
chisq.test(mytable)
mytable <- xtabs(~Improved+Sex, data=Arthritis)
chisq.test(mytable) # Fisher's exact test
mytable <- xtabs(~Treatment+Improved, data=Arthritis)
fisher.test(mytable) # Chochran-Mantel-Haenszel test
mytable <- xtabs(~Treatment+Improved+Sex, data=Arthritis)
mantelhaen.test(mytable) # Listing 7.13 - Measures of association for a two-way table
library(vcd)
mytable <- xtabs(~Treatment+Improved, data=Arthritis)
assocstats(mytable) # Listing 7.14 Covariances and correlations
states<- state.x77[,1:6]
cov(states)
cor(states)
cor(states, method="spearman") x <- states[,c("Population", "Income", "Illiteracy", "HS Grad")]
y <- states[,c("Life Exp", "Murder")]
cor(x,y) # partial correlations
library(ggm)
# partial correlation of population and murder rate, controlling
# for income, illiteracy rate, and HS graduation rate
pcor(c(1,5,2,3,6), cov(states)) # Listing 7.15 - Testing a correlation coefficient for significance
cor.test(states[,3], states[,5]) # Listing 7.16 - Correlation matrix and tests of significance via corr.test
library(psych)
corr.test(states, use="complete") # t test
library(MASS)
t.test(Prob ~ So, data=UScrime) # dependent t test
sapply(UScrime[c("U1","U2")], function(x)(c(mean=mean(x),sd=sd(x))))
with(UScrime, t.test(U1, U2, paired=TRUE)) # Wilcoxon two group comparison
with(UScrime, by(Prob, So, median))
wilcox.test(Prob ~ So, data=UScrime) sapply(UScrime[c("U1", "U2")], median)
with(UScrime, wilcox.test(U1, U2, paired=TRUE)) # Kruskal Wallis test
states <- data.frame(state.region, state.x77)
kruskal.test(Illiteracy ~ state.region, data=states) # Listing 7.17 - Nonparametric multiple comparisons
source("http://www.statmethods.net/RiA/wmc.txt")
states <- data.frame(state.region, state.x77)
wmc(Illiteracy ~ state.region, data=states, method="holm")
吴裕雄--天生自然 R语言开发学习:基本统计分析的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:基础知识
1.基础数据结构 1.1 向量 # 创建向量a a <- c(1,2,3) print(a) 1.2 矩阵 #创建矩阵 mymat <- matrix(c(1:10), nrow=2, n ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- 吴裕雄--天生自然 JAVA开发学习:基本数据类型
public class PrimitiveTypeTest { public static void main(String[] args) { // byte System.out.println ...
- Git上传代码命令
对于Git在这不做说明:只介绍Git使用过程中的常用命令: 一.创建仓库,提交文件 1.初始化一个Git仓库,使用git init命令. 2.添加文件到Git仓库,分两步: 第一步,使用命令git a ...
- ZJNU 1138 - 小兔的棋盘——中级
二维图的动态规划因为不能穿越对角线,则选取对角线的一边dp即可选取对角线右下侧则x轴上每个点只能由其左侧的点走过去(只有1条)对角线上的点只能由对角线下方的点走过去其他点可以由左侧和下侧两种方式到达因 ...
- 897B. Chtholly's request#长度为偶数的回文数(模拟)
题目出处:http://codeforces.com/problemset/problem/897/B 题目大意:构造一个题意要求的zcy数之后取模 #include<iostream> ...
- CodeForces 91B Queue (线段树,区间最值)
http://codeforces.com/problemset/problem/91/B B. Queue time limit per test: 2 seconds memory limit p ...
- SLAM——视觉里程计(一)feature
从现在开始下面两篇文章来介绍SLAM中的视觉里程计(Visual Odometry).这个是我们正式进入SLAM工程的第一步,而之前介绍的更多的是一些基础理论.视觉里程计完成的事情是视觉里程计VO的目 ...
- Yahoo!团队:网站性能优化的35条黄金守则
Yahoo!的 Exceptional Performance团队为改善 Web性能带来最佳实践.他们为此进行了一系列的实验.开发了各种工具.写了大量的文章和博客并在各种会议上参与探讨.最佳实践的核心 ...
- erp和crm的区别
CRM(Customer Relationship Management)即客户关系管理.从字面上来看,是指企业用CRM来管理与客户之间的关系.在不同场合下,CRM可能是一个管理学术语,可能是一个软件 ...
- 《运筹学基础及应用》习题1.3(b)
习题1.3(b):分别用图解法和单纯形法求解下述线性规划问题,并对照指出单纯形表中的各基可行解分别对应图解法中可行域的哪一顶点.$\max z=2x_1+x_2$,$$s.t.\begin{cases ...
- Hibernate学习之hql 与sql
Hibernate中查询: createQuery( String qlString)使用的是HQL语句: createNativeQuery (String sqlString)使用的是SQL语句: ...
