Geatpy遗传算法在曲线寻优上的初步探究
园子里关于遗传算法的教案不少,但基于geatpy框架的并未多见,故分享此文以作参考,还望广大园友多多指教!
Geatpy出自三所名校联合团队之手,是遗传算法领域的权威框架(python),其效率之高、应用领域之广远胜诸多第三方工具,此处不作赘述,直接上链接:
官网:http://www.geatpy.com/start
源码:https://github.com/geatpy-dev/geatpy/tree/master/geatpy
使用Geatpy需要安装geatpy模块(pip install geatpy),linux下如果装完后import时出现报错,可以下载我帖尾链接里的wheel文件进行安装。
言归正传,根据经典的遗传算法流程,无外乎这几个步骤:种群初始化 ->(适应度评价 -> 遴选 -> 交叉 -> 变异)<- 循环进化直至终止条件达标。
当然,有关遗传算法的原理和过程不做深讨,本文旨在剖析遗传算法在阵曲线寻优上的高效应用,我写了一个简单示例来帮助大家更好的理解,代码如下:
# -*- coding: utf-8 -*-
"""punishing.py - 罚函数demo""" import numpy as np def punishing(LegV, FitnV):
FitnV[np.where(LegV == 0)[0]] = 0
return FitnV
# -*- coding: utf-8 -*-
""" aimfc.py即目标函数,本例通过输入每一代的染色体,由自定义评价函数计算与目标曲线的面积差,作为目标函数值ObjV来输出 """ import numpy as np def MakeObjCurve(width):
''' 创建目标曲线,此处定义为一组正弦波拼接序列 '''
n1 = width//3
n2 = width - 2*n1
x1=np.cos(np.arange(0,n1)) * 1
x2=np.cos(np.arange(0,n1)) * 4
x3=np.cos(np.arange(0,n2)) * 2
ObjCurve=np.hstack((x1,x2,x3))
return ObjCurve def CalScore(chrom):
''' 返回染色体与目标曲线之间的面积的倒数作为评分值 '''
objCurve = MakeObjCurve(len(chrom))
area = chrom - objCurve
area *= 10**5 #调整系数确保分值不受小数项干扰
score = 1 / np.dot(area, area) #计算差值的平方和以简化求面积过程
return score def myEvaFunc(chroms):
''' 自定义评价函数,以评分值作为目标函数值 '''
scores = []
for chrom in chroms:
score = CalScore(chrom)
scores.append(score)
scores = np.array([scores]).T
return scores def aimfuc(Phen, LegV): ObjV = myEvaFunc(Phen)
exIdx = np.argmin(ObjV[:, 0]) # 惩罚方法2: 标记非可行解在可行性列向量中对应的值为0,并编写punishing罚函数来修改非可行解的适应度。
# 也可以不写punishing,因为Geatpy内置的算法模板及内核已经对LegV标记为0的个体的适应度作出了修改。
# 使用punishing罚函数实质上是对非可行解个体的适应度作进一步的修改
LegV[exIdx] = 0 # 对非可行解作出标记,使其在可行性列向量中对应的值为0,此处标记的是得分最小项 return [ObjV, LegV]
# -*- coding: utf-8 -*-
""" main.py即主函数,本例仅用于演示“已知曲线寻优”的过程 """ import numpy as np
import geatpy as ga
import time
import matplotlib.pyplot as plt def search_objects(directory):
directory=os.path.normpath(directory) #规格化,防止分隔符造成的差异
if not os.path.isdir(directory):
raise IOError("The directory '"+"' doesn't exist!")
objects={}
for curdir,substrs,files in os.walk(directory):
for jpeg in (file for file in files if file.endswith('.csv')):
path=os.path.join(curdir,jpeg)
label=path.split(os.path.sep)[-2]
if label not in objects:
objects[label]=[]
objects[label].append(path)
return objects def sga_mps_real_templet(AIM_M, AIM_F, PUN_M, PUN_F, FieldDRs, problem, maxormin, MAXGEN, NIND, SUBPOP, GGAP, selectStyle, recombinStyle, recopt, pm, distribute, drawing = 1):
""" 基于多种群独立进化单目标编程模板(实值编码),各种群独立将父子两代合并进行选择,采取精英保留机制 """
#==========================初始化配置===========================
GGAP = 0.5 # 因为父子合并后选择,因此要将代沟设为0.5以维持种群规模
# 获取目标函数和罚函数
aimfuc = getattr(AIM_M, AIM_F) # 获得目标函数
if PUN_F is not None:
punishing = getattr(PUN_M, PUN_F) # 获得罚函数
NVAR = FieldDRs[0].shape[1] # 得到控制变量的个数
# 定义全局进化记录器,初始值为nan
pop_trace = (np.zeros((MAXGEN ,2)) * np.nan)
pop_trace[:, 0] = 0
# 定义变量记录器,记录控制变量值,初始值为nan
var_trace = (np.zeros((MAXGEN ,NVAR)) * np.nan)
"""=========================开始遗传算法进化======================="""
start_time = time.time() # 开始计时
# 对于各个网格分别进行进化,采用全局进化记录器记录最优值
for index in range(len(FieldDRs)): # 遍历各个子种群,各子种群独立进化,互相不竞争
FieldDR = FieldDRs[index]
if problem == 'R':
Chrom = ga.crtrp(NIND, FieldDR) # 生成初始种群
elif problem == 'I':
Chrom = ga.crtip(NIND, FieldDR)
LegV = np.ones((NIND, 1)) # 初始化种群的可行性列向量
[ObjV, LegV] = aimfuc(Chrom, LegV) # 求初始种群的目标函数值
repnum = 0 # 初始化重复个体数为0
ax = None # 存储上一帧图形
gen = 0
badCounter = 0 # 用于记录在“遗忘策略下”被忽略的代数
# 开始进化!!
while gen < MAXGEN:
if badCounter >= 10 * MAXGEN: # 若多花了10倍的迭代次数仍没有可行解出现,则跳出
break
# 进行遗传算子,生成子代
SelCh = ga.recombin(recombinStyle, Chrom, recopt, SUBPOP) # 重组
if problem == 'R':
SelCh = ga.mutbga(SelCh,FieldDR, pm) # 变异
if repnum > Chrom.shape[0] * 0.01: # 当最优个体重复率高达1%时,进行一次高斯变异
SelCh = ga.mutgau(SelCh, FieldDR, pm) # 高斯变异
elif problem == 'I':
SelCh = ga.mutint(SelCh, FieldDR, pm)
LegVSel = np.ones((SelCh.shape[0], 1)) # 初始化育种种群的可行性列向量
[ObjVSel, LegVSel] = aimfuc(SelCh, LegVSel) # 求育种种群的目标函数值
# 父子合并
Chrom = np.vstack([Chrom, SelCh])
ObjV = np.vstack([ObjV, ObjVSel])
LegV = np.vstack([LegV, LegVSel])
FitnV = ga.ranking(maxormin * ObjV, LegV, None, SUBPOP) # 适应度评价
if PUN_F is not None:
FitnV = punishing(LegV, FitnV) # 调用惩罚函数
repnum = len(np.where(ObjV[np.argmax(FitnV)] == ObjV)[0]) # 计算最优个体重复数
# 记录进化过程
bestIdx = np.argmax(FitnV)
if (LegV[bestIdx] != 0) and ((np.isnan(pop_trace[gen,1])) or ((maxormin == 1) & (pop_trace[gen,1] >= ObjV[bestIdx])) or ((maxormin == -1) & (pop_trace[gen,1] <= ObjV[bestIdx]))):
feasible = np.where(LegV != 0)[0] # 排除非可行解
pop_trace[gen,0] += np.sum(ObjV[feasible]) / ObjV[feasible].shape[0] / len(FieldDRs) # 记录种群个体平均目标函数值
pop_trace[gen,1] = ObjV[bestIdx] # 记录当代目标函数的最优值
var_trace[gen,:] = Chrom[bestIdx, :] # 记录当代最优的控制变量值
# 绘制动态图
if drawing == 2:
ax = ga.sgaplot(pop_trace[:,[1]],'子种群'+str(index+1)+'各代种群最优个体目标函数值', False, ax, gen)
badCounter = 0 # badCounter计数器清零
else:
gen -= 1 # 忽略这一代(遗忘策略)
badCounter += 1
if distribute == True: # 若要增强种群的分布性(可能会造成收敛慢)
idx = np.argsort(ObjV[:, 0], 0)
dis = np.diff(ObjV[idx,0]) / (np.max(ObjV[idx,0]) - np.min(ObjV[idx,0]) + 1)# 差分计算距离的修正偏移量
dis = np.hstack([dis, dis[-1]])
dis = dis + np.min(dis) # 修正偏移量+最小量=修正绝对量
FitnV[idx, 0] *= np.exp(dis) # 根据相邻距离修改适应度,突出相邻距离大的个体,以增加种群的多样性
[Chrom, ObjV, LegV] = ga.selecting(selectStyle, Chrom, FitnV, GGAP, SUBPOP, ObjV, LegV) # 选择
gen += 1
end_time = time.time() # 结束计时
times = end_time - start_time
# 后处理进化记录器
delIdx = np.where(np.isnan(pop_trace))[0]
pop_trace = np.delete(pop_trace, delIdx, 0)
var_trace = np.delete(var_trace, delIdx, 0)
if pop_trace.shape[0] == 0:
raise RuntimeError('error: no feasible solution. (有效进化代数为0,没找到可行解。)')
# 输出结果
if maxormin == 1:
best_gen = np.argmin(pop_trace[:, 1]) # 记录最优种群是在哪一代
best_ObjV = np.min(pop_trace[:, 1])
elif maxormin == -1:
best_gen = np.argmax(pop_trace[:, 1]) # 记录最优种群是在哪一代
best_ObjV = np.max(pop_trace[:, 1])
print('最优的目标函数值为:%s'%(best_ObjV))
print('最优的控制变量值为:')
for i in range(NVAR):
print(var_trace[best_gen, i])
print('有效进化代数:%s'%(pop_trace.shape[0]))
print('最优的一代是第 %s 代'%(best_gen + 1))
print('时间已过 %s 秒'%(times))
# 绘图
if drawing != 0:
ga.trcplot(pop_trace, [['种群个体平均目标函数值', '种群最优个体目标函数值']])
# 返回进化记录器、变量记录器以及执行时间
return [pop_trace, var_trace, times, best_gen] # 获取函数接口地址
AIM_M = __import__('aimfuc')
PUN_M = __import__('punishing')
POP_SIZE = 300 # 种群高度
CHROM_LENGTH = 20 # 染色体宽度
max_generation = 150 # 进化代数
chrom_bottom = -4 #染色体数值下限
chrom_top = 4 #染色体数值上限 # 变量设置
x = []; b = []
for i in range(CHROM_LENGTH):
x.append([chrom_bottom, chrom_top]) # 自变量的范围
b.append([0, 0]) # 自变量是否包含下界
ranges=np.vstack(x).T # 生成自变量的范围矩阵
borders = np.vstack(b).T # 生成自变量的边界矩阵
precisions = [1]*CHROM_LENGTH # 在二进制/格雷码编码中代表自变量的编码精度,当控制变量是连续型时,根据crtfld参考资料,该变量只表示边界精度,故设置为一定的正数即可
# 生成网格化后的区域描述器集合
FieldDRs = []
for i in range(1):
FieldDRs.append(ga.crtfld(ranges, borders, precisions)) # 调用编程模板(设置problem = 'R'处理实数型变量问题,详见该算法模板的源代码)
[pop_trace, var_trace, times, best_gen] = sga_mps_real_templet(AIM_M, 'aimfuc', PUN_M, 'punishing',
FieldDRs, problem = 'R', maxormin = -1, MAXGEN = max_generation, NIND = POP_SIZE, SUBPOP = 1, GGAP = 0.9, \
selectStyle = 'tour', recombinStyle = 'xovdprs', recopt = 0.9, pm = 0.3, distribute = True, drawing = 1) bstChrom = var_trace[best_gen]
objCurve = AIM_M.MakeObjCurve(CHROM_LENGTH) plt.ion()
fig = plt.figure('曲线寻优演示',facecolor='lightgray')
ax1 = fig.add_subplot(2, 1, 1)
ax2 = fig.add_subplot(2, 1, 2)
ax1.set_title("Evaluation Map")
ax1.grid(axis='y', linestyle=':')
for i in range(max_generation):
if i%5==0:
ax1.plot(var_trace[i], 'o-')
ax2.cla()
ax2.set_title("最优染色体[gen:%i]"%(i+1))
ax2.plot(var_trace[i], 'o-', c='dodgerblue')
plt.pause(0.001)
ax2.cla()
ax2.grid(axis='y', linestyle=':')
ax2.plot(objCurve, 'o-', c='orangered', label='目标曲线')
ax2.plot(bstChrom, 'o-', c='dodgerblue', label='最优染色体[gen:%i]'%(best_gen+1))
plt.legend()
plt.ioff()
plt.show()
请注意,此处我已将模板函数单独放到主函数中以便大家更好的理解,返回值中增加了最优代数以便后续图例的显示。
本例采用的进化模板是sga_mps_real_templet,基于多种群进化单目标(实数值),用于实现寻找目标曲线。
目标曲线的定义函数在aimfc.py的MakeObjCurve函数中,本例为3段振幅不同的cos函数拼接而成的模拟曲线,宽度20。
种群初始值设置:种群高度300、染色体宽度20(与目标曲线宽度保持一致)、进化代数150、染色体数值上下限[-4,4](与目标曲线的上下限保持一致)。
接着我们开始寻优,通过CalScore函数计算每代种群的每条染色体与目标函数之间的差值,经过一定的系数转换得到评分值(差值越大,评分越低),以单目标(1列)形式输出,由geatpy的ranking函数来决定适应度评价,然后继续遴选、交叉、变异,如此循环往复,直至达到近似目标值时终止(本例设定为150代时终止),寻优过程如下图:
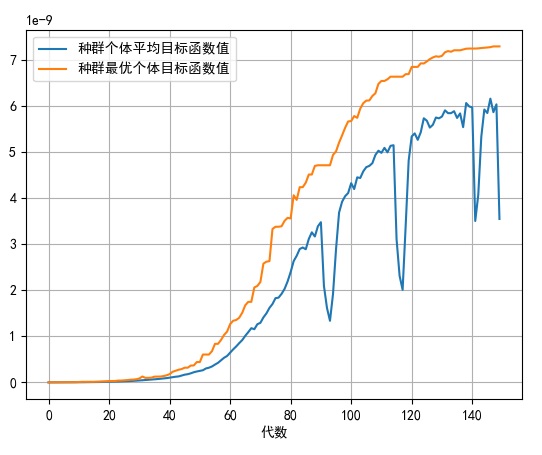
最优的目标函数值为:7.287567405118076e-09
有效进化代数:150
最优的一代是第 148 代
时间已过 1.3259999752044678 秒
可以看到从50代左右优化曲线开始显著上扬,直到130代左右逐渐平缓,并且耗时非常少,来看寻优结果图:
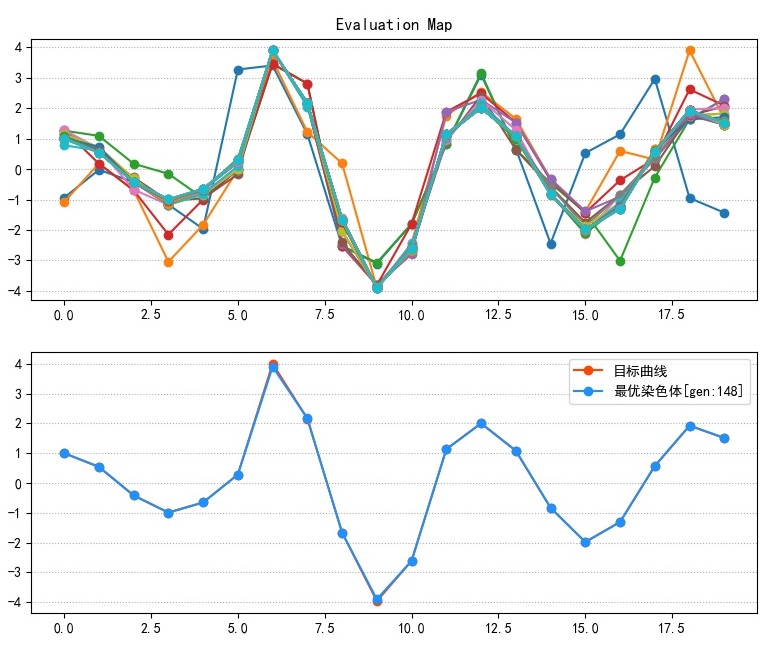
可以看到除了第6个点的数值有细微差异之外,其他点几乎都是吻合的,基本实现了目标曲线的寻求。
之所以抛出本例,最重要的一点在于geatpy遗传算法不仅能寻求已知的目标函数,还可以通过自定义的评分体系或第三方接口来参与实现寻优过程,只需将CalScore函数稍作改动即可,以上。
【wheel文件】: https://pan.baidu.com/s/1BwLq_m3Dd5RMqatvTYXrAw 提取码: vgkz
Geatpy遗传算法在曲线寻优上的初步探究的更多相关文章
- MATLAB神经网络(4) 神经网络遗传算法函数极值寻优——非线性函数极值寻优
4.1 案例背景 \[y = {x_1}^2 + {x_2}^2\] 4.2 模型建立 神经网络训练拟合根据寻优函数的特点构建合适的BP神经网络,用非线性函数的输入输出数据训练BP神经网络,训练后的B ...
- 小程序组件化框架 WePY 在性能调优上做出的探究
作者:龚澄 导语 性能调优是一个亘古不变的话题,无论是在传统H5上还是小程序中.因为实现机制不同,可能导致传统H5中的某些优化方式在小程序上并不适用.因此必须另开辟蹊径找出适合小程序的调估方式. 本文 ...
- 模拟退火算法SA原理及python、java、php、c++语言代码实现TSP旅行商问题,智能优化算法,随机寻优算法,全局最短路径
模拟退火算法SA原理及python.java.php.c++语言代码实现TSP旅行商问题,智能优化算法,随机寻优算法,全局最短路径 模拟退火算法(Simulated Annealing,SA)最早的思 ...
- paper 36 :[教程] 基于GridSearch的svm参数寻优
尊重原创~~~ 转载出处:http://www.matlabsky.com/thread-12411-1-1.html 交叉验证(Cross Validation)方法思想简介http://www.m ...
- grid search 超参数寻优
http://scikit-learn.org/stable/modules/grid_search.html 1. 超参数寻优方法 gridsearchCV 和 RandomizedSearchC ...
- 吴裕雄 python 机器学习——模型选择参数优化暴力搜索寻优GridSearchCV模型
import scipy from sklearn.datasets import load_digits from sklearn.metrics import classification_rep ...
- 吴裕雄 python 机器学习——模型选择参数优化随机搜索寻优RandomizedSearchCV模型
import scipy from sklearn.datasets import load_digits from sklearn.metrics import classification_rep ...
- 体验CoreCLR的stack unwinding特性在Linux/Mac上的初步实现
有了stack unwinding特性,才能在.NET程序中获取调用堆栈(call stack)信息,才能在异常时显示调用堆栈信息.这个特性之前只在Windows上有实现,Linux/Mac上的实现最 ...
- kinect在ros上的初步测试---17
摘要: 原创博客:转载请表明出处:http://www.cnblogs.com/zxouxuewei/ 1.在使用本贴前必须先按照我的上一个博文正确在ubuntu上安装kinect驱动:http:// ...
随机推荐
- 模糊控制——(3)模糊自适应整定PID控制
1.原理 这种控制必须精确地确定对象模型,首先将操作人员(专家)长期实践积累的经验知识用控制规则模型化,然后运用推理便可对PID参数实现最佳调整. 自适应模糊PID控制器以误差e和误差变化ec作为输入 ...
- c++——深拷贝和浅拷贝
深拷贝和浅拷贝 默认复制构造函数可以完成对象的数据成员值简单的复制 对象的数据资源是由指针指示的堆时,默认复制构造函数仅作指针值复制 1浅拷贝问题 1.c++默认的拷贝构造函数 2.=号操作符 都是浅 ...
- vbs获取当前主机IP
Function GetIP GetIP = "" Dim objWMIService, colAdapters, objAdapter strComputer ...
- mysql5.7.22 下载过程
进入官网www.mysql.com ,选择downloads: 选择 Community 再选择MySQL community server 选择5.7的版本,这个看自己选择,有问题看标题 选择5.7 ...
- bootstrap-treeview使用
1.数据 var tree = [{ text: "车型A", nodes: [{ text: "车系1", }, { text: "车系2" ...
- 评价指标1--F1值和MSE
1,F1=2*(准确率*召回率)/(准确率+召回率) F1的值是精准率与召回率的调和平均数.F1的取值范围从0到1的数量越大,表明实现越理想. Precision(精准率)=TP/(TP+FP) Re ...
- scribe日志系统安装笔记
版本历史 2011-02-12 17:36:57 完成文章2011-02-23 10:49:12 更新,修正部分文字 目前web访问日志为crontab定时清空,而且负载均衡后,访问随机分配到一台服务 ...
- 06 django的用户认证组件
1.用户认证组件 用户认证组件: 功能:用session记录登录验证状态 前提:用户表:django自带的auth_user 创建超级用户: python3 manage.py createsuper ...
- Python Machine Learning: Scikit-Learn Tutorial
这是一篇翻译的博客,原文链接在这里.这是我看的为数不多的介绍scikit-learn简介而全面的文章,特别适合入门.我这里把这篇文章翻译一下,英语好的同学可以直接看原文. 大部分喜欢用Python来学 ...
- Java虚拟机笔记(二):GC垃圾回收和对象的引用
为什么要了解GC 我们都知道Java开发者在开发过程中是不需要关心对象的回收的,因为Java虚拟机的原因,它会自动回收那些失效的垃圾对象.那我们为什么还要去了解GC和内存分配呢? 答案很简单:当我们需 ...
